Aritalab:Lecture/Math/PCA/R PCA
From Metabolomics.JP
RによるPCA
主成分分析は prcomp, princomp (statsパッケージ)や acp (amapパッケージ) という関数で実行します。 ここでは Rによる回帰分析のページで使った菖蒲のデータを再び使います。
prcomp による解析
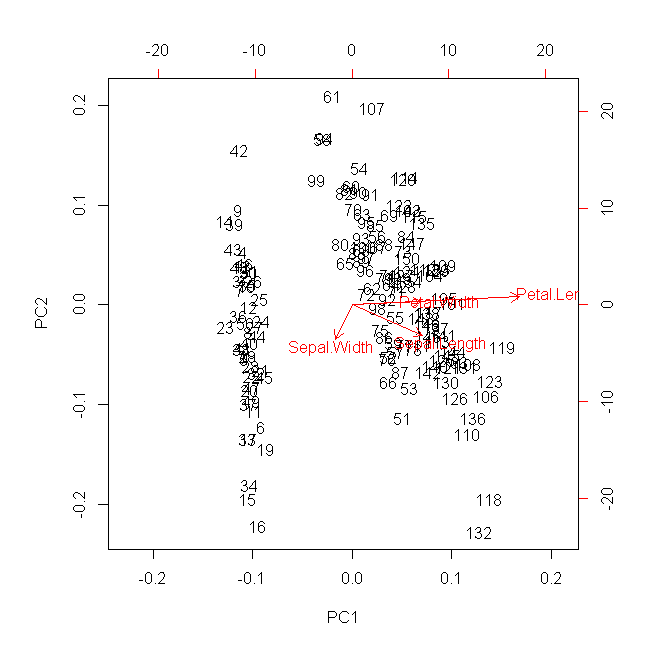
> s <- prcomp(iris[1:4], scale=FALSE)
> biplot(s,xlabs = iris[,1])
> summary(s)
Importance of components:
PC1 PC2 PC3 PC4
Standard deviation 2.0563 0.49262 0.2797 0.15439
Proportion of Variance 0.9246 0.05307 0.0171 0.00521
Cumulative Proportion 0.9246 0.97769 0.9948 1.00000
biplot 関数はローディング値を表示します。
主成分 PC1 でほとんどの分散を説明できることがわかります。
説明変数それぞれの寄与は s をみるとわかります。
PC1は Petal.Length とほぼ同じ方向です。PC2はSepalから構成されています。
> s
Standard deviations:
[1] 2.0562689 0.4926162 0.2796596 0.1543862
Rotation:
PC1 PC2 PC3 PC4
Sepal.Length 0.36138659 -0.65658877 0.58202985 0.3154872
Sepal.Width -0.08452251 -0.73016143 -0.59791083 -0.3197231
Petal.Length 0.85667061 0.17337266 -0.07623608 -0.4798390
Petal.Width 0.35828920 0.07548102 -0.54583143 0.7536574
変数構造の中身を見るには names(s) とします。
acpによる解析
amapパッケージでもほとんど同じ事ができるようです。
> install.packages("amap")
...
> library(amap)
...
> s <- acp(iris[,-5])
> s
Standard deviations:
Comp 1 Comp 2 Comp 3 Comp 4
1.7083611 0.9560494 0.3830886 0.1439265
Eigen values:
[1] 20.853205 11.670070 4.676192 1.756847
> s$loadings # s$scoresでスコアも表示できます
Comp 1 Comp 2 Comp 3 Comp 4
Sepal.Length 0.5210659 -0.37741762 -0.7195664 0.2612863
Sepal.Width -0.2693474 -0.92329566 0.2443818 -0.1235096
Petal.Length 0.5804131 -0.02449161 0.1421264 -0.8014492
Petal.Width 0.5648565 -0.06694199 0.6342727 0.5235971
> plot(s)
> biplot(s)