Aritalab:Lecture/Bioinformatics/Alignment
Needleman-Wunsch アルゴリズム
1970年、分子生物学者の Saul B. NeedlemanとChristian D. Wunsch は、大域アライメントのアルゴリズムを Journal of Molecular Biology誌 (1970) 48: 443-453 に発表しました。いまではNeedleman-Wunsch アルゴリズムと呼ばれています。
基本は最長共通部分列(LCS)アルゴリズムと同じで、動的計画法によるLCSのページで紹介した以下の漸化式に基づいてスコア行列を計算します。ギャップとミスマッチに対してそれぞれペナルティスコア (gap penalty σ, mismatch penalty δ)が与えられています。
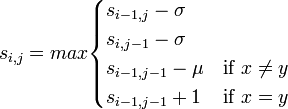
|
ここではギャップペナルティを 2、ミスマッチペナルティを 1 として、2つの配列 gctagg と aattgaagg の間のアライメントを考えてみましょう。スコア行列は右のようになります。 1行目と1列目は 0, -2, -4, -6, ... -18 とギャップペナルティに基づく等差数列ができています。これは左隣(または上)のセルからギャップペナルティ 2 を順次マイナスしてできる部分です。大域アライメントでは配列の最初と最後を揃えるために、初期状態としてギャップの数に比例するペナルティを与えます。 残りのセルは動的計画法に基づいてスコアが埋められ、最適アライメントは以下のようになります。
1:aattgaagg:9
| | ||
1:gct--a-gg:6
右図のトレースバックと見比べてみてください。大域アライメントのスコアは マッチ 4 × 1 + ミスマッチ 2 × (-1) + ギャップ 3 × (-2) = -4 となっています。大域アライメントにおけるギャップの数は配列長の差になります。具体的なアルゴリズムとJavaコードはこのページを参照してください。 |
|