Aritalab:Lecture/Bioinformatics/Alignment
| Line 1: | Line 1: | ||
| − | ==Needleman-Wunsch アルゴリズム== | + | ==Needleman-Wunsch (-Sellers) アルゴリズム== |
| − | + | 1970年、分子生物学者の Saul B. NeedlemanとChristian D. Wunsch は、大域アライメントのアルゴリズムを Journal of Molecular Biology誌 (1970) 48: 443-453 に発表しました。いまでは誤って Needleman-Wunsch アルゴリズムと呼ばれるようになりましたが、論文の内容はずっと効率が悪い手法、O(n<sup>3</sup>) 時間、の紹介でした。ここで紹介されるアルゴリズムの原型を作ったのは Peter Sellers (1974) です<ref>P Sellers "On the theory and computation of evolutionary distances" SIAM J. Appl. Math. 26:787–793, 1974 にアフィンギャップつきのアルゴリズムが紹介されています。</ref>。したがって正確には Needleman-Wunsch-Sellers アルゴリズムと呼ぶべきでしょう <ref>1970年当時の様子は David Sankoff "The early introduction of dynamic programming into computational biology" Bioinformatics 16(1):41-47 ([http://bioinformatics.oxfordjournals.org/content/16/1/41.full.pdf pdf])を見るとよいでしょう。</ref>。 | |
| − | 1970年、分子生物学者の Saul B. NeedlemanとChristian D. Wunsch は、大域アライメントのアルゴリズムを Journal of Molecular Biology誌 (1970) 48: 443-453 | + | |
基本は最長共通部分列(LCS)アルゴリズムと同じで、[[Aritalab:Lecture/Bioinformatics/Homology|動的計画法によるLCSのページ]]で紹介した以下の漸化式に基づいてスコア行列を計算します。ギャップとミスマッチに対してそれぞれペナルティスコア (gap penalty σ, mismatch penalty δ)が与えられています。 | 基本は最長共通部分列(LCS)アルゴリズムと同じで、[[Aritalab:Lecture/Bioinformatics/Homology|動的計画法によるLCSのページ]]で紹介した以下の漸化式に基づいてスコア行列を計算します。ギャップとミスマッチに対してそれぞれペナルティスコア (gap penalty σ, mismatch penalty δ)が与えられています。 | ||
| Line 76: | Line 75: | ||
|} | |} | ||
|} | |} | ||
| + | |||
| + | ==Smith-Waterman アルゴリズム== | ||
| + | |||
| + | バイオインフォマティクスの草分けである Temple F. Smith と Michael S. Waterman が 1981 年に Journal of Molecular Biology誌 (147: 195–197) に発表した局所アライメントを求めるアルゴリズムです。MS Waterman は動的計画法を RNA 二次構造の解析などに応用して活躍した数学者ですが、TF Smithは配列アライメントに関する実績もなく、このアルゴリズムをWalther Goad から聞いて論文にしたといわれる曰くつきのアルゴリズムです<ref>Walter Goad は後日 W Goad "Sequence Analysis: Contributions by Ulam to Molecular Genetics" Los Alamos Science (special issue) 288-291, 1987 ([http://library.lanl.gov/cgi-bin/getfile?15-21.pdf pdf]) に、自分のほうが先に見つけたと言いたげな記述をしています。</ref>。局所アライメントは以下のように定義されます。 | ||
| + | |||
| + | <center> | ||
| + | 局所アライメント = 与えられた2つの配列それぞれの全ての部分配列のペア中で、大域アライメントのスコアを最大にするもの | ||
| + | </center> | ||
| + | |||
| + | そのまま適用するとすべての部分配列を列挙して大域アライメントを施す必要が思えますが、動的計画法において常にスコア 0 を候補に置くだけで局所アライメントを求められるところが大変エレガントです。 | ||
| + | |||
| + | <math> s_{i,j} = max \begin{cases}0 \\ s_{i-1,j} - \sigma \\ s_{i,j-1} - \sigma \\ s_{i-1,j-1} - \mu & \mbox{if } x \ne y \\ s_{i-1,j-1} +1 & \mbox{if } x = y | ||
| + | \end{cases}</math> | ||
| + | |||
| + | |||
| + | ;参考文献 | ||
| + | <references/> | ||
Revision as of 06:38, 15 December 2011
Needleman-Wunsch (-Sellers) アルゴリズム
1970年、分子生物学者の Saul B. NeedlemanとChristian D. Wunsch は、大域アライメントのアルゴリズムを Journal of Molecular Biology誌 (1970) 48: 443-453 に発表しました。いまでは誤って Needleman-Wunsch アルゴリズムと呼ばれるようになりましたが、論文の内容はずっと効率が悪い手法、O(n3) 時間、の紹介でした。ここで紹介されるアルゴリズムの原型を作ったのは Peter Sellers (1974) です[1]。したがって正確には Needleman-Wunsch-Sellers アルゴリズムと呼ぶべきでしょう [2]。
基本は最長共通部分列(LCS)アルゴリズムと同じで、動的計画法によるLCSのページで紹介した以下の漸化式に基づいてスコア行列を計算します。ギャップとミスマッチに対してそれぞれペナルティスコア (gap penalty σ, mismatch penalty δ)が与えられています。
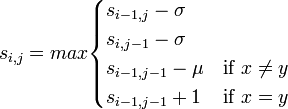
|
ここではギャップペナルティを 2、ミスマッチペナルティを 1 として、2つの配列 gctagg と aattgaagg の間のアライメントを考えてみましょう。スコア行列は右のようになります。 1行目と1列目は 0, -2, -4, -6, ... -18 とギャップペナルティに基づく等差数列ができています。これは左隣(または上)のセルからギャップペナルティ 2 を順次マイナスしてできる部分です。大域アライメントでは配列の最初と最後を揃えるために、初期状態としてギャップの数に比例するペナルティを与えます。 残りのセルは動的計画法に基づいてスコアが埋められ、最適アライメントは以下のようになります。
1:aattgaagg:9
| | ||
1:gct--a-gg:6
右図のトレースバックと見比べてみてください。大域アライメントのスコアは マッチ 4 × 1 + ミスマッチ 2 × (-1) + ギャップ 3 × (-2) = -4 となっています。大域アライメントにおけるギャップの数は配列長の差になります。具体的なアルゴリズムとJavaコードはこのページを参照してください。 |
|
Smith-Waterman アルゴリズム
バイオインフォマティクスの草分けである Temple F. Smith と Michael S. Waterman が 1981 年に Journal of Molecular Biology誌 (147: 195–197) に発表した局所アライメントを求めるアルゴリズムです。MS Waterman は動的計画法を RNA 二次構造の解析などに応用して活躍した数学者ですが、TF Smithは配列アライメントに関する実績もなく、このアルゴリズムをWalther Goad から聞いて論文にしたといわれる曰くつきのアルゴリズムです[3]。局所アライメントは以下のように定義されます。
局所アライメント = 与えられた2つの配列それぞれの全ての部分配列のペア中で、大域アライメントのスコアを最大にするもの
そのまま適用するとすべての部分配列を列挙して大域アライメントを施す必要が思えますが、動的計画法において常にスコア 0 を候補に置くだけで局所アライメントを求められるところが大変エレガントです。
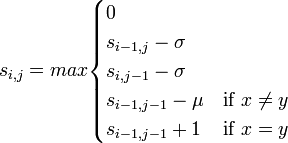
- 参考文献
- ↑ P Sellers "On the theory and computation of evolutionary distances" SIAM J. Appl. Math. 26:787–793, 1974 にアフィンギャップつきのアルゴリズムが紹介されています。
- ↑ 1970年当時の様子は David Sankoff "The early introduction of dynamic programming into computational biology" Bioinformatics 16(1):41-47 (pdf)を見るとよいでしょう。
- ↑ Walter Goad は後日 W Goad "Sequence Analysis: Contributions by Ulam to Molecular Genetics" Los Alamos Science (special issue) 288-291, 1987 (pdf) に、自分のほうが先に見つけたと言いたげな記述をしています。