Mol:PR100462
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | ACD/Labs08070915122D | + | ACD/Labs08070915122D |
| − | + | ||
| − | 53 58 0 0 1 0 0 0 0 0 2 V2000 | + | 53 58 0 0 1 0 0 0 0 0 2 V2000 |
| − | 14.5058 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.5058 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.5058 -7.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.5058 -7.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.3384 -7.7477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.3384 -7.7477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.1711 -7.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.1711 -7.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.1711 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.1711 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.3384 -5.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.3384 -5.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.0037 -7.7477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.0037 -7.7477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.8363 -7.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.8363 -7.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.8363 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.8363 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.0037 -5.8248 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 17.0037 -5.8248 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 22.7347 -8.9405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.7347 -8.9405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.5673 -8.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.5673 -8.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.5673 -7.4984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.5673 -7.4984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.7347 -7.0176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.7347 -7.0176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.9021 -7.4984 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.9021 -7.4984 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.9021 -8.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.9021 -8.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.7638 -8.4522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.7638 -8.4522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.7347 -6.0562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.7347 -6.0562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.3616 -8.8191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.3616 -8.8191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.6732 -5.8248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.6732 -5.8248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.6690 -5.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.6690 -5.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.5016 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.5016 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.3343 -5.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.3343 -5.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.3343 -4.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.3343 -4.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.5016 -4.3826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.5016 -4.3826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.6690 -4.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.6690 -4.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.5016 -3.4212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.5016 -3.4212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.1669 -4.3826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.1669 -4.3826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.4000 -7.0176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.4000 -7.0176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.7653 -10.6867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.7653 -10.6867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.4960 -8.7086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.4960 -8.7086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.5673 -5.5755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.5673 -5.5755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.0331 -10.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.0331 -10.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.0331 -11.6815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.0331 -11.6815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.8658 -12.1622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.8658 -12.1622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.6984 -11.6815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.6984 -11.6815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.6984 -10.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.6984 -10.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.8658 -10.2393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.8658 -10.2393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.0331 -9.7586 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.0331 -9.7586 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.8658 -9.2779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.8658 -9.2779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 27.5310 -12.1622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 27.5310 -12.1622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 27.5310 -10.2393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 27.5310 -10.2393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.5602 -9.3193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.5602 -9.3193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.5602 -10.2808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.5602 -10.2808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.3928 -10.7615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.3928 -10.7615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.2254 -10.2808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.2254 -10.2808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.2254 -9.3193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.2254 -9.3193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.3928 -8.8386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.3928 -8.8386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.7275 -8.8386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.7275 -8.8386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.3928 -11.7229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.3928 -11.7229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.3928 -7.8772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.3928 -7.8772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.7275 -10.7615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.7275 -10.7615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.5602 -12.2037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.5602 -12.2037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 18 1 6 0 0 0 | + | 14 18 1 6 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 9 21 1 0 0 0 0 | + | 9 21 1 0 0 0 0 |
| − | 21 22 2 0 0 0 0 | + | 21 22 2 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 17 8 1 0 0 0 0 | + | 17 8 1 0 0 0 0 |
| − | 13 29 1 1 0 0 0 | + | 13 29 1 1 0 0 0 |
| − | 11 30 1 1 0 0 0 | + | 11 30 1 1 0 0 0 |
| − | 12 31 1 6 0 0 0 | + | 12 31 1 6 0 0 0 |
| − | 18 32 1 0 0 0 0 | + | 18 32 1 0 0 0 0 |
| − | 17 16 1 6 0 0 0 | + | 17 16 1 6 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 33 39 1 6 0 0 0 | + | 33 39 1 6 0 0 0 |
| − | 38 40 1 6 0 0 0 | + | 38 40 1 6 0 0 0 |
| − | 36 41 1 6 0 0 0 | + | 36 41 1 6 0 0 0 |
| − | 37 42 1 1 0 0 0 | + | 37 42 1 1 0 0 0 |
| − | 33 30 1 0 0 0 0 | + | 33 30 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 43 1 0 0 0 0 | + | 48 43 1 0 0 0 0 |
| − | 43 49 1 6 0 0 0 | + | 43 49 1 6 0 0 0 |
| − | 45 50 1 6 0 0 0 | + | 45 50 1 6 0 0 0 |
| − | 48 51 1 1 0 0 0 | + | 48 51 1 1 0 0 0 |
| − | 44 52 1 1 0 0 0 | + | 44 52 1 1 0 0 0 |
| − | 50 53 1 0 0 0 0 | + | 50 53 1 0 0 0 0 |
| − | 19 47 1 6 0 0 0 | + | 19 47 1 6 0 0 0 |
| − | M CHG 1 10 1 | + | M CHG 1 10 1 |
| − | S SKP | + | S SKP 7 |
| − | CAS_RN 73036-94-9 | + | CAS_RN 73036-94-9 |
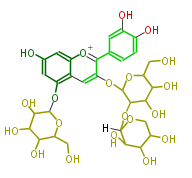
| − | NAME Cyanidin-3-O-(2''-O-beta-xylopyranosyl-beta-glucopyranoside)-5-O-beta-glucopyranoside | + | NAME Cyanidin-3-O-(2''-O-beta-xylopyranosyl-beta-glucopyranoside)-5-O-beta-glucopyranoside |
| + | ID PR100462 | ||
| + | FORMULA C32H39O20 | ||
| + | EXACTMASS 743.2034686879999 | ||
| + | AVERAGEMASS 743.64006 | ||
| + | SMILES c(c1)(O)c(O)ccc1c(c4OC(C5OC([H])(O6)C(C(C(C6)O)O)O)OC(C(O)C5O)CO)[o+1]c(c2c4)cc(O)cc2OC(O3)C(C(C(C3CO)O)O)O | ||
M END | M END | ||
Latest revision as of 16:44, 14 January 2011
ACD/Labs08070915122D 53 58 0 0 1 0 0 0 0 0 2 V2000 14.5058 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 14.5058 -7.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 15.3384 -7.7477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 16.1711 -7.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 16.1711 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 15.3384 -5.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.0037 -7.7477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.8363 -7.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.8363 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.0037 -5.8248 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 22.7347 -8.9405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 23.5673 -8.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 23.5673 -7.4984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.7347 -7.0176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 21.9021 -7.4984 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 21.9021 -8.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 19.7638 -8.4522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 22.7347 -6.0562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 15.3616 -8.8191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 13.6732 -5.8248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 18.6690 -5.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 19.5016 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 20.3343 -5.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 20.3343 -4.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 19.5016 -4.3826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 18.6690 -4.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 19.5016 -3.4212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 21.1669 -4.3826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 24.4000 -7.0176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 22.7653 -10.6867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 24.4960 -8.7086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 23.5673 -5.5755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 25.0331 -10.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 25.0331 -11.6815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 25.8658 -12.1622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 26.6984 -11.6815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 26.6984 -10.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 25.8658 -10.2393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 25.0331 -9.7586 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 25.8658 -9.2779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 27.5310 -12.1622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 27.5310 -10.2393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 12.5602 -9.3193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 12.5602 -10.2808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 13.3928 -10.7615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 14.2254 -10.2808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 14.2254 -9.3193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 13.3928 -8.8386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 11.7275 -8.8386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 13.3928 -11.7229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 13.3928 -7.8772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 11.7275 -10.7615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 12.5602 -12.2037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 1 2 2 0 0 0 0 2 3 1 0 0 0 0 3 4 2 0 0 0 0 4 5 1 0 0 0 0 5 6 2 0 0 0 0 6 1 1 0 0 0 0 4 7 1 0 0 0 0 7 8 2 0 0 0 0 8 9 1 0 0 0 0 9 10 2 0 0 0 0 10 5 1 0 0 0 0 11 12 1 0 0 0 0 12 13 1 0 0 0 0 13 14 1 0 0 0 0 14 15 1 0 0 0 0 15 16 1 0 0 0 0 16 11 1 0 0 0 0 14 18 1 6 0 0 0 3 19 1 0 0 0 0 1 20 1 0 0 0 0 9 21 1 0 0 0 0 21 22 2 0 0 0 0 22 23 1 0 0 0 0 23 24 2 0 0 0 0 24 25 1 0 0 0 0 25 26 2 0 0 0 0 26 21 1 0 0 0 0 25 27 1 0 0 0 0 24 28 1 0 0 0 0 17 8 1 0 0 0 0 13 29 1 1 0 0 0 11 30 1 1 0 0 0 12 31 1 6 0 0 0 18 32 1 0 0 0 0 17 16 1 6 0 0 0 33 34 1 0 0 0 0 34 35 1 0 0 0 0 35 36 1 0 0 0 0 36 37 1 0 0 0 0 37 38 1 0 0 0 0 38 33 1 0 0 0 0 33 39 1 6 0 0 0 38 40 1 6 0 0 0 36 41 1 6 0 0 0 37 42 1 1 0 0 0 33 30 1 0 0 0 0 43 44 1 0 0 0 0 44 45 1 0 0 0 0 45 46 1 0 0 0 0 46 47 1 0 0 0 0 47 48 1 0 0 0 0 48 43 1 0 0 0 0 43 49 1 6 0 0 0 45 50 1 6 0 0 0 48 51 1 1 0 0 0 44 52 1 1 0 0 0 50 53 1 0 0 0 0 19 47 1 6 0 0 0 M CHG 1 10 1 S SKP 7 CAS_RN 73036-94-9 NAME Cyanidin-3-O-(2''-O-beta-xylopyranosyl-beta-glucopyranoside)-5-O-beta-glucopyranoside ID PR100462 FORMULA C32H39O20 EXACTMASS 743.2034686879999 AVERAGEMASS 743.64006 SMILES c(c1)(O)c(O)ccc1c(c4OC(C5OC([H])(O6)C(C(C(C6)O)O)O)OC(C(O)C5O)CO)[o+1]c(c2c4)cc(O)cc2OC(O3)C(C(C(C3CO)O)O)O M END