Mol:PR100459
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | ACD/Labs08070915092D | + | ACD/Labs08070915092D |
| − | + | ||
| − | 44 48 0 0 1 0 0 0 0 0 2 V2000 | + | 44 48 0 0 1 0 0 0 0 0 2 V2000 |
| − | 17.0074 -5.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.0074 -5.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.0074 -6.3084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.0074 -6.3084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.8400 -6.7892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.8400 -6.7892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.6727 -6.3084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.6727 -6.3084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.6727 -5.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.6727 -5.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.8400 -4.8663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.8400 -4.8663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.5053 -6.7892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.5053 -6.7892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.3379 -6.3084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.3379 -6.3084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.3379 -5.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.3379 -5.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.5053 -4.8663 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 19.5053 -4.8663 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 21.1677 -4.8679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.1677 -4.8679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.0003 -5.3487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.0003 -5.3487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.8330 -4.8679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.8330 -4.8679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.8330 -3.9065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.8330 -3.9065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.0003 -3.4258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.0003 -3.4258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.1677 -3.9065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.1677 -3.9065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.6627 -3.4274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.6627 -3.4274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.6627 -5.3470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.6627 -5.3470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.1777 -4.8679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.1777 -4.8679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.1677 -6.7875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.1677 -6.7875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.1677 -7.7456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.1677 -7.7456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.8400 -7.7473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.8400 -7.7473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.0103 -8.2263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.0103 -8.2263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.1777 -7.7456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.1777 -7.7456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.3450 -8.2263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.3450 -8.2263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.3450 -9.1878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.3450 -9.1878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.1777 -9.6685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.1777 -9.6685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.0103 -9.1878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.0103 -9.1878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.3351 -8.2263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.3351 -8.2263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.3351 -9.1878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.3351 -9.1878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.1677 -9.6685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.1677 -9.6685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.0003 -9.1878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.0003 -9.1878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.0003 -8.2263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.0003 -8.2263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.4925 -4.8679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.4925 -4.8679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.1777 -6.7875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.1777 -6.7875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.5153 -9.6668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.5153 -9.6668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.5053 -7.7473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.5053 -7.7473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.1677 -10.6266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.1677 -10.6266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.5153 -7.7473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.5153 -7.7473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.1777 -10.6266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.1777 -10.6266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.5053 -9.6668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.5053 -9.6668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.8301 -9.6668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.8301 -9.6668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.6598 -9.1878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.6598 -9.1878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.3479 -11.1056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.3479 -11.1056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 13 18 1 0 0 0 0 | + | 13 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 21 20 1 6 0 0 0 | + | 21 20 1 6 0 0 0 |
| − | 3 22 1 0 0 0 0 | + | 3 22 1 0 0 0 0 |
| − | 23 22 1 6 0 0 0 | + | 23 22 1 6 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 21 29 1 0 0 0 0 | + | 21 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 21 1 0 0 0 0 | + | 33 21 1 0 0 0 0 |
| − | 18 34 1 0 0 0 0 | + | 18 34 1 0 0 0 0 |
| − | 24 35 1 1 0 0 0 | + | 24 35 1 1 0 0 0 |
| − | 26 36 1 1 0 0 0 | + | 26 36 1 1 0 0 0 |
| − | 29 37 1 1 0 0 0 | + | 29 37 1 1 0 0 0 |
| − | 31 38 1 1 0 0 0 | + | 31 38 1 1 0 0 0 |
| − | 25 39 1 6 0 0 0 | + | 25 39 1 6 0 0 0 |
| − | 27 40 1 6 0 0 0 | + | 27 40 1 6 0 0 0 |
| − | 30 41 1 6 0 0 0 | + | 30 41 1 6 0 0 0 |
| − | 32 42 1 6 0 0 0 | + | 32 42 1 6 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 40 44 1 0 0 0 0 | + | 40 44 1 0 0 0 0 |
| − | M CHG 1 10 1 | + | M CHG 1 10 1 |
| − | S SKP | + | S SKP 7 |
| − | CAS_RN 47851-83-2 | + | CAS_RN 47851-83-2 |
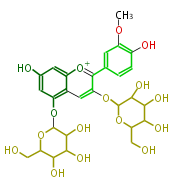
| − | NAME Peonidin-3,5-O-di-beta-glucopyranoside | + | NAME Peonidin-3,5-O-di-beta-glucopyranoside |
| + | ID PR100459 | ||
| + | FORMULA C28H33O16 | ||
| + | EXACTMASS 625.176860008 | ||
| + | AVERAGEMASS 625.55202 | ||
| + | SMILES c(O)(c(OC)1)ccc(c(c(OC(O5)C(O)C(O)C(O)C5CO)4)[o+1]c(c(c4)2)cc(O)cc2OC(O3)C(O)C(O)C(O)C3CO)c1 | ||
M END | M END | ||
Latest revision as of 16:44, 14 January 2011
ACD/Labs08070915092D 44 48 0 0 1 0 0 0 0 0 2 V2000 17.0074 -5.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.0074 -6.3084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.8400 -6.7892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 18.6727 -6.3084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 18.6727 -5.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.8400 -4.8663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 19.5053 -6.7892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 20.3379 -6.3084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 20.3379 -5.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 19.5053 -4.8663 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 21.1677 -4.8679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.0003 -5.3487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.8330 -4.8679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.8330 -3.9065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.0003 -3.4258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 21.1677 -3.9065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 23.6627 -3.4274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 23.6627 -5.3470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 16.1777 -4.8679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 21.1677 -6.7875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 21.1677 -7.7456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.8400 -7.7473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 17.0103 -8.2263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 16.1777 -7.7456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 15.3450 -8.2263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 15.3450 -9.1878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 16.1777 -9.6685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.0103 -9.1878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 20.3351 -8.2263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 20.3351 -9.1878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 21.1677 -9.6685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.0003 -9.1878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.0003 -8.2263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 24.4925 -4.8679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 16.1777 -6.7875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 14.5153 -9.6668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 19.5053 -7.7473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 21.1677 -10.6266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 14.5153 -7.7473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 16.1777 -10.6266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 19.5053 -9.6668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 22.8301 -9.6668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 23.6598 -9.1878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 15.3479 -11.1056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 1 2 2 0 0 0 0 2 3 1 0 0 0 0 3 4 2 0 0 0 0 4 5 1 0 0 0 0 5 6 2 0 0 0 0 6 1 1 0 0 0 0 4 7 1 0 0 0 0 7 8 2 0 0 0 0 8 9 1 0 0 0 0 9 10 2 0 0 0 0 10 5 1 0 0 0 0 9 11 1 0 0 0 0 11 12 2 0 0 0 0 12 13 1 0 0 0 0 13 14 2 0 0 0 0 14 15 1 0 0 0 0 15 16 2 0 0 0 0 16 11 1 0 0 0 0 14 17 1 0 0 0 0 13 18 1 0 0 0 0 1 19 1 0 0 0 0 8 20 1 0 0 0 0 21 20 1 6 0 0 0 3 22 1 0 0 0 0 23 22 1 6 0 0 0 23 24 1 0 0 0 0 24 25 1 0 0 0 0 25 26 1 0 0 0 0 26 27 1 0 0 0 0 27 28 1 0 0 0 0 28 23 1 0 0 0 0 21 29 1 0 0 0 0 29 30 1 0 0 0 0 30 31 1 0 0 0 0 31 32 1 0 0 0 0 32 33 1 0 0 0 0 33 21 1 0 0 0 0 18 34 1 0 0 0 0 24 35 1 1 0 0 0 26 36 1 1 0 0 0 29 37 1 1 0 0 0 31 38 1 1 0 0 0 25 39 1 6 0 0 0 27 40 1 6 0 0 0 30 41 1 6 0 0 0 32 42 1 6 0 0 0 42 43 1 0 0 0 0 40 44 1 0 0 0 0 M CHG 1 10 1 S SKP 7 CAS_RN 47851-83-2 NAME Peonidin-3,5-O-di-beta-glucopyranoside ID PR100459 FORMULA C28H33O16 EXACTMASS 625.176860008 AVERAGEMASS 625.55202 SMILES c(O)(c(OC)1)ccc(c(c(OC(O5)C(O)C(O)C(O)C5CO)4)[o+1]c(c(c4)2)cc(O)cc2OC(O3)C(O)C(O)C(O)C3CO)c1 M END