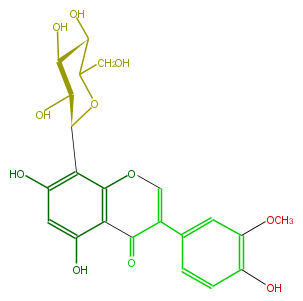
Mol:FLIAADCS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 33 36 0 0 0 0 0 0 0 0999 V2000 | + | 33 36 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.2974 -0.7815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2974 -0.7815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2974 -1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2974 -1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7411 -1.7450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7411 -1.7450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1848 -1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1848 -1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1848 -0.7815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1848 -0.7815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7411 -0.4603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7411 -0.4603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6285 -1.7450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6285 -1.7450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0722 -1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0722 -1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0722 -0.7815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0722 -0.7815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6285 -0.4603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6285 -0.4603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6285 -2.2459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6285 -2.2459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8535 -0.4604 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8535 -0.4604 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4957 2.2242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4957 2.2242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1013 1.7654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1013 1.7654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8444 1.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8444 1.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8375 0.4673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8375 0.4673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3742 0.9305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3742 0.9305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6792 1.5085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6792 1.5085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7922 2.7376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7922 2.7376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1361 2.4806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1361 2.4806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4798 0.7262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4798 0.7262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7411 -2.3872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7411 -2.3872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3808 -2.3992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3808 -2.3992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9670 -2.7376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9670 -2.7376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5532 -2.3992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5532 -2.3992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5532 -1.7223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5532 -1.7223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9670 -1.3838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9670 -1.3838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3808 -1.7223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3808 -1.7223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1391 -2.7374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1391 -2.7374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2441 1.7597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2441 1.7597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5296 1.3472 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5296 1.3472 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1391 -1.3840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1391 -1.3840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8535 -1.7965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8535 -1.7965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 1 1 0 0 0 | + | 13 14 1 1 0 0 0 |
| − | 14 15 1 1 0 0 0 | + | 14 15 1 1 0 0 0 |
| − | 16 15 1 1 0 0 0 | + | 16 15 1 1 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 19 1 0 0 0 0 | + | 13 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 6 16 1 0 0 0 0 | + | 6 16 1 0 0 0 0 |
| − | 3 22 1 0 0 0 0 | + | 3 22 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 28 8 1 0 0 0 0 | + | 28 8 1 0 0 0 0 |
| − | 18 30 1 0 0 0 0 | + | 18 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 26 32 1 0 0 0 0 | + | 26 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 30 31 | + | M SAL 1 2 30 31 |
| − | M SBL 1 1 33 | + | M SBL 1 1 33 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 33 -7.2619 5.7810 | + | M SBV 1 33 -7.2619 5.7810 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 32 33 | + | M SAL 2 2 32 33 |
| − | M SBL 2 1 35 | + | M SBL 2 1 35 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 35 -7.1112 5.8680 | + | M SBV 2 35 -7.1112 5.8680 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLIAADCS0001 | + | ID FLIAADCS0001 |
| − | KNApSAcK_ID C00006132 | + | KNApSAcK_ID C00006132 |
| − | NAME Dalpanitin | + | NAME Dalpanitin |
| − | CAS_RN 40522-83-6 | + | CAS_RN 40522-83-6 |
| − | FORMULA C22H22O11 | + | FORMULA C22H22O11 |
| − | EXACTMASS 462.116211546 | + | EXACTMASS 462.116211546 |
| − | AVERAGEMASS 462.40348000000006 | + | AVERAGEMASS 462.40348000000006 |
| − | SMILES C(C4CO)(C(C(O)C(O4)c(c12)c(O)cc(O)c(C(C(c(c3)cc(c(c3)O)OC)=CO2)=O)1)O)O | + | SMILES C(C4CO)(C(C(O)C(O4)c(c12)c(O)cc(O)c(C(C(c(c3)cc(c(c3)O)OC)=CO2)=O)1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
33 36 0 0 0 0 0 0 0 0999 V2000
-2.2974 -0.7815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2974 -1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7411 -1.7450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1848 -1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1848 -0.7815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7411 -0.4603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6285 -1.7450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0722 -1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0722 -0.7815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6285 -0.4603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6285 -2.2459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8535 -0.4604 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4957 2.2242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1013 1.7654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8444 1.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8375 0.4673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3742 0.9305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6792 1.5085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7922 2.7376 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1361 2.4806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4798 0.7262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7411 -2.3872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3808 -2.3992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9670 -2.7376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5532 -2.3992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5532 -1.7223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9670 -1.3838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3808 -1.7223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1391 -2.7374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2441 1.7597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5296 1.3472 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1391 -1.3840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8535 -1.7965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 1 1 0 0 0
14 15 1 1 0 0 0
16 15 1 1 0 0 0
16 17 1 0 0 0 0
17 18 1 0 0 0 0
18 13 1 0 0 0 0
13 19 1 0 0 0 0
14 20 1 0 0 0 0
15 21 1 0 0 0 0
6 16 1 0 0 0 0
3 22 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 23 1 0 0 0 0
25 29 1 0 0 0 0
28 8 1 0 0 0 0
18 30 1 0 0 0 0
30 31 1 0 0 0 0
26 32 1 0 0 0 0
32 33 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 30 31
M SBL 1 1 33
M SMT 1 CH2OH
M SBV 1 33 -7.2619 5.7810
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 32 33
M SBL 2 1 35
M SMT 2 OCH3
M SBV 2 35 -7.1112 5.8680
S SKP 8
ID FLIAADCS0001
KNApSAcK_ID C00006132
NAME Dalpanitin
CAS_RN 40522-83-6
FORMULA C22H22O11
EXACTMASS 462.116211546
AVERAGEMASS 462.40348000000006
SMILES C(C4CO)(C(C(O)C(O4)c(c12)c(O)cc(O)c(C(C(c(c3)cc(c(c3)O)OC)=CO2)=O)1)O)O
M END