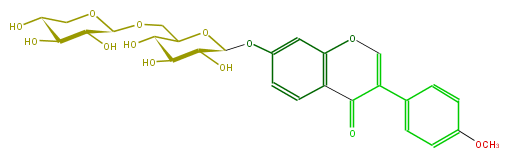
Mol:FLIA1AGS0013
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 40 44 0 0 0 0 0 0 0 0999 V2000 | + | 40 44 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.5603 1.3252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5603 1.3252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0926 1.0250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0926 1.0250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8329 1.4523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8329 1.4523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5730 1.0252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5730 1.0252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5730 0.1390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5730 0.1390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3404 -0.3042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3404 -0.3042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1081 0.1390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1081 0.1390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1081 1.0252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1081 1.0252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3404 1.4683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3404 1.4683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0930 0.1706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0930 0.1706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8329 -0.2566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8329 -0.2566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3404 -1.1898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3404 -1.1898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8750 -0.3038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8750 -0.3038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8750 -1.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8750 -1.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6088 -1.5747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6088 -1.5747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3423 -1.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3423 -1.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3423 -0.3038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3423 -0.3038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6088 0.1198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6088 0.1198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2333 1.4995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2333 1.4995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7213 0.8237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7213 0.8237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9842 1.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9842 1.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2158 1.1021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2158 1.1021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7897 1.6353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7897 1.6353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5428 1.3649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5428 1.3649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8022 1.2948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8022 1.2948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2904 0.8032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2904 0.8032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2959 0.6558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2959 0.6558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9886 1.8173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9886 1.8173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6508 1.8641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6508 1.8641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.4019 2.0316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.4019 2.0316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8898 1.3557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8898 1.3557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1525 1.6425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1525 1.6425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3843 1.6338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3843 1.6338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9582 2.1671 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9582 2.1671 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7112 1.8967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7112 1.8967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.0359 1.8116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.0359 1.8116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.5923 1.3860 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.5923 1.3860 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5623 1.2362 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5623 1.2362 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8833 -1.5016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8833 -1.5016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0359 -2.1671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0359 -2.1671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 4 1 0 0 0 0 | + | 9 4 1 0 0 0 0 |
| − | 2 10 1 0 0 0 0 | + | 2 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 5 1 0 0 0 0 | + | 11 5 1 0 0 0 0 |
| − | 6 12 2 0 0 0 0 | + | 6 12 2 0 0 0 0 |
| − | 7 13 1 0 0 0 0 | + | 7 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 1 1 0 0 0 0 | + | 22 1 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 29 1 0 0 0 0 | + | 33 29 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 16 39 1 0 0 0 0 | + | 16 39 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 44 -0.5410 0.3506 | + | M SBV 1 44 -0.5410 0.3506 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FLIA1AGS0013 | + | ID FLIA1AGS0013 |
| − | FORMULA C27H30O13 | + | FORMULA C27H30O13 |
| − | EXACTMASS 562.168641046 | + | EXACTMASS 562.168641046 |
| − | AVERAGEMASS 562.5193 | + | AVERAGEMASS 562.5193 |
| − | SMILES C(C(C(O)5)OC(C(O)C5O)Oc(c4)cc(c(c4)2)OC=C(c(c3)ccc(OC)c3)C2=O)OC(O1)C(O)C(C(O)C1)O | + | SMILES C(C(C(O)5)OC(C(O)C5O)Oc(c4)cc(c(c4)2)OC=C(c(c3)ccc(OC)c3)C2=O)OC(O1)C(O)C(C(O)C1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
40 44 0 0 0 0 0 0 0 0999 V2000
-0.5603 1.3252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0926 1.0250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8329 1.4523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5730 1.0252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5730 0.1390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3404 -0.3042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1081 0.1390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1081 1.0252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3404 1.4683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0930 0.1706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8329 -0.2566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3404 -1.1898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8750 -0.3038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8750 -1.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6088 -1.5747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3423 -1.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3423 -0.3038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6088 0.1198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2333 1.4995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7213 0.8237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9842 1.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2158 1.1021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7897 1.6353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5428 1.3649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8022 1.2948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2904 0.8032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2959 0.6558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9886 1.8173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6508 1.8641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.4019 2.0316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8898 1.3557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1525 1.6425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3843 1.6338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9582 2.1671 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7112 1.8967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.0359 1.8116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.5923 1.3860 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5623 1.2362 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.8833 -1.5016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.0359 -2.1671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 4 1 0 0 0 0
2 10 1 0 0 0 0
10 11 2 0 0 0 0
11 5 1 0 0 0 0
6 12 2 0 0 0 0
7 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 1 1 0 0 0 0
24 28 1 0 0 0 0
28 29 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 29 1 0 0 0 0
39 40 1 0 0 0 0
16 39 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 44
M SMT 1 OCH3
M SBV 1 44 -0.5410 0.3506
S SKP 5
ID FLIA1AGS0013
FORMULA C27H30O13
EXACTMASS 562.168641046
AVERAGEMASS 562.5193
SMILES C(C(C(O)5)OC(C(O)C5O)Oc(c4)cc(c(c4)2)OC=C(c(c3)ccc(OC)c3)C2=O)OC(O1)C(O)C(C(O)C1)O
M END