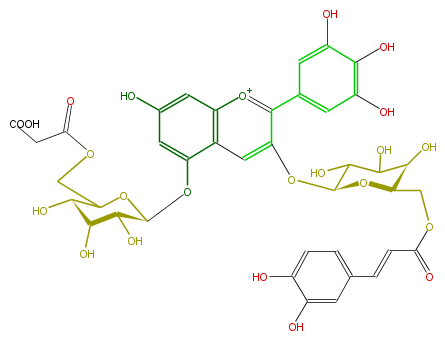
Mol:FL7AAGGL0024
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 62 67 0 0 0 0 0 0 0 0999 V2000 | + | 62 67 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.8608 1.1749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8608 1.1749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8608 0.5325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8608 0.5325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3045 0.2113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3045 0.2113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2518 0.5325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2518 0.5325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2518 1.1749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2518 1.1749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3045 1.4960 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3045 1.4960 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8081 0.2113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8081 0.2113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3644 0.5325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3644 0.5325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3644 1.1749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3644 1.1749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8081 1.4960 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.8081 1.4960 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9205 1.4959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9205 1.4959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4875 1.1686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4875 1.1686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0545 1.4959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0545 1.4959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0545 2.1506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0545 2.1506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4875 2.4780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4875 2.4780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9205 2.1506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9205 2.1506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4169 1.4959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4169 1.4959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6213 2.4779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6213 2.4779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3045 -0.4308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3045 -0.4308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7805 -0.1882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7805 -0.1882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9380 0.1119 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.9380 0.1119 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.6371 -0.4092 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.6371 -0.4092 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.2156 -0.2438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2156 -0.2438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7976 -0.4092 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.7976 -0.4092 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.0985 0.1119 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.0985 0.1119 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.5200 -0.0534 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.5200 -0.0534 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.3793 -0.0378 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3793 -0.0378 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5615 0.4212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5615 0.4212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4529 0.4663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4529 0.4663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3492 -0.4092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3492 -0.4092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6282 -0.6019 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.6282 -0.6019 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.2570 -1.0918 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.2570 -1.0918 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.7226 -0.8840 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.7226 -0.8840 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.1116 -1.0320 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.1116 -1.0320 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.5816 -0.5035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5816 -0.5035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0492 -0.7503 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.0492 -0.7503 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.1857 -0.8047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1857 -0.8047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3796 -1.6580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3796 -1.6580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4163 -1.3981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4163 -1.3981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9069 -0.2361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9069 -0.2361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5397 -1.1203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5397 -1.1203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2503 -1.6216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2503 -1.6216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7352 -1.6216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7352 -1.6216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4408 -2.1314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4408 -2.1314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9515 -2.1314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9515 -2.1314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6623 -1.6305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6623 -1.6305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0839 -1.6305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0839 -1.6305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7947 -2.1314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7947 -2.1314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0839 -2.6323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0839 -2.6323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6623 -2.6323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6623 -2.6323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2172 -2.1314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2172 -2.1314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5460 -2.1337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5460 -2.1337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4875 3.1325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4875 3.1325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2487 0.3392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2487 0.3392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7485 0.7586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7485 0.7586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3291 0.5473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3291 0.5473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7966 0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7966 0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3202 0.7489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3202 0.7489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6353 1.4004 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6353 1.4004 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6908 1.5394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6908 1.5394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6213 1.1687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6213 1.1687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7952 -3.1325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7952 -3.1325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 34 19 1 0 0 0 0 | + | 34 19 1 0 0 0 0 |
| − | 30 41 1 0 0 0 0 | + | 30 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 48 51 1 0 0 0 0 | + | 48 51 1 0 0 0 0 |
| − | 42 52 2 0 0 0 0 | + | 42 52 2 0 0 0 0 |
| − | 15 53 1 0 0 0 0 | + | 15 53 1 0 0 0 0 |
| − | 40 54 1 0 0 0 0 | + | 40 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 57 58 2 0 0 0 0 | + | 57 58 2 0 0 0 0 |
| − | 55 59 2 0 0 0 0 | + | 55 59 2 0 0 0 0 |
| − | 57 60 1 0 0 0 0 | + | 57 60 1 0 0 0 0 |
| − | 13 61 1 0 0 0 0 | + | 13 61 1 0 0 0 0 |
| − | 49 62 1 0 0 0 0 | + | 49 62 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 57 58 60 | + | M SAL 1 3 57 58 60 |
| − | M SBL 1 1 62 | + | M SBL 1 1 62 |
| − | M SMT 1 COOH | + | M SMT 1 COOH |
| − | M SVB 1 62 -4.0635 0.6104 | + | M SVB 1 62 -4.0635 0.6104 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAGGL0024 | + | ID FL7AAGGL0024 |
| − | KNApSAcK_ID C00006887 | + | KNApSAcK_ID C00006887 |
| − | NAME Monodemalonylsalviadelphin;Delphinidin 3-[6-(caffeoylglucoside)]-5-[6-(malonylglucoside)] | + | NAME Monodemalonylsalviadelphin;Delphinidin 3-[6-(caffeoylglucoside)]-5-[6-(malonylglucoside)] |
| − | CAS_RN 128508-46-3,144607-77-2 | + | CAS_RN 128508-46-3,144607-77-2 |
| − | FORMULA C39H39O23 | + | FORMULA C39H39O23 |
| − | EXACTMASS 875.1882125540001 | + | EXACTMASS 875.1882125540001 |
| − | AVERAGEMASS 875.71316 | + | AVERAGEMASS 875.71316 |
| − | SMILES [C@H](O)([C@@H]1Oc(c6)c(c3cc6O)cc(O[C@@H](C5O)O[C@@H]([C@@H](C(O)5)O)COC(=O)C=Cc(c4)ccc(O)c4O)c([o+1]3)c(c2)cc(O)c(O)c2O)[C@@H](O)[C@@H](O)C(COC(CC(O)=O)=O)O1 | + | SMILES [C@H](O)([C@@H]1Oc(c6)c(c3cc6O)cc(O[C@@H](C5O)O[C@@H]([C@@H](C(O)5)O)COC(=O)C=Cc(c4)ccc(O)c4O)c([o+1]3)c(c2)cc(O)c(O)c2O)[C@@H](O)[C@@H](O)C(COC(CC(O)=O)=O)O1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
62 67 0 0 0 0 0 0 0 0999 V2000
-0.8608 1.1749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8608 0.5325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3045 0.2113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2518 0.5325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2518 1.1749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3045 1.4960 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8081 0.2113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3644 0.5325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3644 1.1749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8081 1.4960 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
1.9205 1.4959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4875 1.1686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0545 1.4959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0545 2.1506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4875 2.4780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9205 2.1506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4169 1.4959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6213 2.4779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3045 -0.4308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7805 -0.1882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9380 0.1119 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.6371 -0.4092 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.2156 -0.2438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7976 -0.4092 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.0985 0.1119 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.5200 -0.0534 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.3793 -0.0378 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5615 0.4212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4529 0.4663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3492 -0.4092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6282 -0.6019 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.2570 -1.0918 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.7226 -0.8840 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.1116 -1.0320 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.5816 -0.5035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0492 -0.7503 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.1857 -0.8047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3796 -1.6580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4163 -1.3981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9069 -0.2361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5397 -1.1203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2503 -1.6216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7352 -1.6216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4408 -2.1314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9515 -2.1314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6623 -1.6305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0839 -1.6305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7947 -2.1314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0839 -2.6323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6623 -2.6323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2172 -2.1314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5460 -2.1337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4875 3.1325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2487 0.3392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7485 0.7586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3291 0.5473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7966 0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3202 0.7489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6353 1.4004 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6908 1.5394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6213 1.1687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7952 -3.1325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
22 20 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
36 40 1 0 0 0 0
34 19 1 0 0 0 0
30 41 1 0 0 0 0
41 42 1 0 0 0 0
42 43 1 0 0 0 0
43 44 2 0 0 0 0
44 45 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 45 1 0 0 0 0
48 51 1 0 0 0 0
42 52 2 0 0 0 0
15 53 1 0 0 0 0
40 54 1 0 0 0 0
54 55 1 0 0 0 0
55 56 1 0 0 0 0
56 57 1 0 0 0 0
57 58 2 0 0 0 0
55 59 2 0 0 0 0
57 60 1 0 0 0 0
13 61 1 0 0 0 0
49 62 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 57 58 60
M SBL 1 1 62
M SMT 1 COOH
M SVB 1 62 -4.0635 0.6104
S SKP 8
ID FL7AAGGL0024
KNApSAcK_ID C00006887
NAME Monodemalonylsalviadelphin;Delphinidin 3-[6-(caffeoylglucoside)]-5-[6-(malonylglucoside)]
CAS_RN 128508-46-3,144607-77-2
FORMULA C39H39O23
EXACTMASS 875.1882125540001
AVERAGEMASS 875.71316
SMILES [C@H](O)([C@@H]1Oc(c6)c(c3cc6O)cc(O[C@@H](C5O)O[C@@H]([C@@H](C(O)5)O)COC(=O)C=Cc(c4)ccc(O)c4O)c([o+1]3)c(c2)cc(O)c(O)c2O)[C@@H](O)[C@@H](O)C(COC(CC(O)=O)=O)O1
M END