Mol:FL7AACGL0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.8099 0.8105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8099 0.8105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8099 0.1681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8099 0.1681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2536 -0.1531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2536 -0.1531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6973 0.1681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6973 0.1681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6973 0.8105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6973 0.8105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2536 1.1317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2536 1.1317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1410 -0.1531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1410 -0.1531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5847 0.1681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5847 0.1681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5847 0.8105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5847 0.8105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1410 1.1317 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -1.1410 1.1317 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0286 1.1315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0286 1.1315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5384 0.8042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5384 0.8042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1053 1.1315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1053 1.1315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1053 1.7862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1053 1.7862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5384 2.1136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5384 2.1136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0286 1.7862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0286 1.7862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6721 2.1135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6721 2.1135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3660 1.1315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3660 1.1315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2536 -0.7952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2536 -0.7952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0082 -0.2620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0082 -0.2620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5384 2.7681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5384 2.7681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2067 -1.9452 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -0.2067 -1.9452 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 0.2673 -2.4276 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.2673 -2.4276 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.1219 -1.6888 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.1219 -1.6888 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.2746 -1.1512 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.2746 -1.1512 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.2067 -0.8733 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.2067 -0.8733 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.0540 -1.4077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0540 -1.4077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5347 -3.3912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5347 -3.3912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5553 -1.9390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5553 -1.9390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9211 -0.4008 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 0.9211 -0.4008 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.4067 -0.8865 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.4067 -0.8865 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.0035 -0.5464 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.0035 -0.5464 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.6903 -0.5464 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.6903 -0.5464 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.2046 -0.0606 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.2046 -0.0606 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.6078 -0.4008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6078 -0.4008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2467 -0.6333 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2467 -0.6333 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8996 -0.8865 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8996 -0.8865 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6869 -0.6284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6869 -0.6284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3227 -1.0993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3227 -1.0993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3538 -2.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3538 -2.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3469 -2.4054 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3469 -2.4054 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6058 0.1366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6058 0.1366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6725 1.1344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6725 1.1344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 23 28 1 0 0 0 0 | + | 23 28 1 0 0 0 0 |
| − | 24 29 1 0 0 0 0 | + | 24 29 1 0 0 0 0 |
| − | 26 20 1 0 0 0 0 | + | 26 20 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 25 36 1 0 0 0 0 | + | 25 36 1 0 0 0 0 |
| − | 22 40 1 0 0 0 0 | + | 22 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 46 2.6516 -0.0514 | + | M SVB 2 46 2.6516 -0.0514 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 44 -0.2922 -2.3083 | + | M SVB 1 44 -0.2922 -2.3083 |
| − | S SKP 8 | + | S SKP 8 |
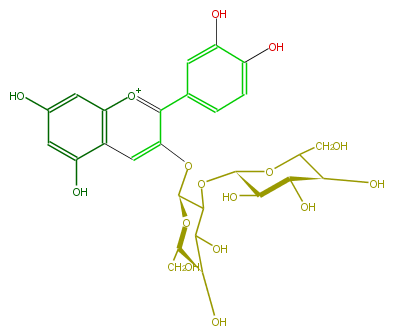
| − | ID FL7AACGL0006 | + | ID FL7AACGL0006 |
| − | KNApSAcK_ID C00006658 | + | KNApSAcK_ID C00006658 |
| − | NAME Cyanidin 3-sophoroside | + | NAME Cyanidin 3-sophoroside |
| − | CAS_RN 38820-68-7,18376-31-3 | + | CAS_RN 38820-68-7,18376-31-3 |
| − | FORMULA C27H31O16 | + | FORMULA C27H31O16 |
| − | EXACTMASS 611.161209944 | + | EXACTMASS 611.161209944 |
| − | AVERAGEMASS 611.52544 | + | AVERAGEMASS 611.52544 |
| − | SMILES Oc(c(O)1)ccc(c(c(O[C@H](O4)C(O[C@H]([C@@H]5O)OC([C@H]([C@H]5O)O)CO)C([C@H]([C@H]4CO)O)O)3)[o+1]c(c2c3)cc(O)cc2O)c1 | + | SMILES Oc(c(O)1)ccc(c(c(O[C@H](O4)C(O[C@H]([C@@H]5O)OC([C@H]([C@H]5O)O)CO)C([C@H]([C@H]4CO)O)O)3)[o+1]c(c2c3)cc(O)cc2O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-2.8099 0.8105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8099 0.1681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2536 -0.1531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6973 0.1681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6973 0.8105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2536 1.1317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1410 -0.1531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5847 0.1681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5847 0.8105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1410 1.1317 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-0.0286 1.1315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5384 0.8042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1053 1.1315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1053 1.7862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5384 2.1136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0286 1.7862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6721 2.1135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3660 1.1315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2536 -0.7952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0082 -0.2620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5384 2.7681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2067 -1.9452 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
0.2673 -2.4276 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.1219 -1.6888 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.2746 -1.1512 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.2067 -0.8733 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.0540 -1.4077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5347 -3.3912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5553 -1.9390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9211 -0.4008 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.4067 -0.8865 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.0035 -0.5464 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.6903 -0.5464 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.2046 -0.0606 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.6078 -0.4008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2467 -0.6333 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8996 -0.8865 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6869 -0.6284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3227 -1.0993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3538 -2.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3469 -2.4054 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6058 0.1366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6725 1.1344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
8 20 1 0 0 0 0
15 21 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
23 28 1 0 0 0 0
24 29 1 0 0 0 0
26 20 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
33 38 1 0 0 0 0
32 39 1 0 0 0 0
25 36 1 0 0 0 0
22 40 1 0 0 0 0
40 41 1 0 0 0 0
34 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 CH2OH
M SVB 2 46 2.6516 -0.0514
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 CH2OH
M SVB 1 44 -0.2922 -2.3083
S SKP 8
ID FL7AACGL0006
KNApSAcK_ID C00006658
NAME Cyanidin 3-sophoroside
CAS_RN 38820-68-7,18376-31-3
FORMULA C27H31O16
EXACTMASS 611.161209944
AVERAGEMASS 611.52544
SMILES Oc(c(O)1)ccc(c(c(O[C@H](O4)C(O[C@H]([C@@H]5O)OC([C@H]([C@H]5O)O)CO)C([C@H]([C@H]4CO)O)O)3)[o+1]c(c2c3)cc(O)cc2O)c1
M END