Mol:FL7AAAGL0029
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 65 71 0 0 0 0 0 0 0 0999 V2000 | + | 65 71 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.5421 1.8827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5421 1.8827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5421 1.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5421 1.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9858 0.9192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9858 0.9192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4295 1.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4295 1.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4295 1.8827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4295 1.8827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9858 2.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9858 2.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1268 0.9192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1268 0.9192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6831 1.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6831 1.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6831 1.8827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6831 1.8827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1268 2.2039 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.1268 2.2039 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2392 2.2038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2392 2.2038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8062 1.8765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8062 1.8765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3732 2.2038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3732 2.2038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3732 2.8585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3732 2.8585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8062 3.1858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8062 3.1858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2392 2.8585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2392 2.8585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0982 2.2038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0982 2.2038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9400 3.1857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9400 3.1857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9858 0.2771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9858 0.2771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0992 0.5197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0992 0.5197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2566 0.8197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2566 0.8197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9558 0.2987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9558 0.2987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5343 0.4641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5343 0.4641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1163 0.2987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1163 0.2987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4172 0.8197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4172 0.8197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8386 0.6545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8386 0.6545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6979 0.6700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6979 0.6700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0768 1.0670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0768 1.0670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8512 0.5691 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8512 0.5691 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6678 0.2987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6678 0.2987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8584 -0.4125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8584 -0.4125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5690 -0.9137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5690 -0.9137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0538 -0.9137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0538 -0.9137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7595 -1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7595 -1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2702 -1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2702 -1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9810 -0.9226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9810 -0.9226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4026 -0.9226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4026 -0.9226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1134 -1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1134 -1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4026 -1.9245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4026 -1.9245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9810 -1.9245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9810 -1.9245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5359 -1.4236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5359 -1.4236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8646 -1.4258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8646 -1.4258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1138 -2.4246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1138 -2.4246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3708 -2.3896 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3708 -2.3896 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9996 -2.8796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9996 -2.8796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4651 -2.6717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4651 -2.6717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1459 -2.8198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1459 -2.8198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3242 -2.2913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3242 -2.2913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7918 -2.5381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7918 -2.5381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8846 -2.6863 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8846 -2.6863 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5783 -2.9077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5783 -2.9077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1589 -3.1858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1589 -3.1858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3508 0.1060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3508 0.1060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9796 -0.3840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9796 -0.3840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4451 -0.1761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4451 -0.1761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8341 -0.3241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8341 -0.3241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3042 0.2043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3042 0.2043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7718 -0.0425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7718 -0.0425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8646 -0.1907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8646 -0.1907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5583 -0.4121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5583 -0.4121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1389 -0.6902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1389 -0.6902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1902 -1.7539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1902 -1.7539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3933 -1.9674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3933 -1.9674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1702 0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1702 0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3733 0.5282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3733 0.5282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 38 41 1 0 0 0 0 | + | 38 41 1 0 0 0 0 |
| − | 32 42 2 0 0 0 0 | + | 32 42 2 0 0 0 0 |
| − | 39 43 1 0 0 0 0 | + | 39 43 1 0 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 47 46 1 1 0 0 0 | + | 47 46 1 1 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | 46 52 1 0 0 0 0 | + | 46 52 1 0 0 0 0 |
| − | 53 54 1 1 0 0 0 | + | 53 54 1 1 0 0 0 |
| − | 54 55 1 1 0 0 0 | + | 54 55 1 1 0 0 0 |
| − | 56 55 1 1 0 0 0 | + | 56 55 1 1 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 53 1 0 0 0 0 | + | 58 53 1 0 0 0 0 |
| − | 53 59 1 0 0 0 0 | + | 53 59 1 0 0 0 0 |
| − | 54 60 1 0 0 0 0 | + | 54 60 1 0 0 0 0 |
| − | 55 61 1 0 0 0 0 | + | 55 61 1 0 0 0 0 |
| − | 56 19 1 0 0 0 0 | + | 56 19 1 0 0 0 0 |
| − | 47 43 1 0 0 0 0 | + | 47 43 1 0 0 0 0 |
| − | 49 62 1 0 0 0 0 | + | 49 62 1 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | 58 64 1 0 0 0 0 | + | 58 64 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 62 63 | + | M SAL 1 2 62 63 |
| − | M SBL 1 1 68 | + | M SBL 1 1 68 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 68 -7.1339 7.4519 | + | M SBV 1 68 -7.1339 7.4519 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 64 65 | + | M SAL 2 2 64 65 |
| − | M SBL 2 1 70 | + | M SBL 2 1 70 |
| − | M SMT 2 ^CH2OH | + | M SMT 2 ^CH2OH |
| − | M SBV 2 70 -7.1339 7.4519 | + | M SBV 2 70 -7.1339 7.4519 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAAGL0029 | + | ID FL7AAAGL0029 |
| − | KNApSAcK_ID C00006768 | + | KNApSAcK_ID C00006768 |
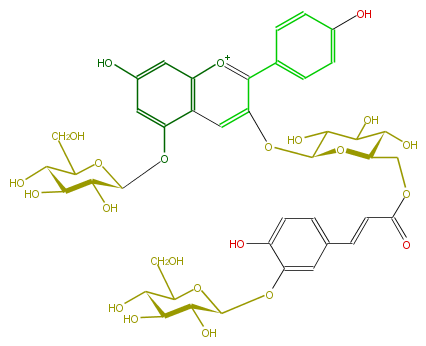
| − | NAME Pelargonidin 3-[6-(3-glucosylcaffeyl)glucoside]-5-glucoside | + | NAME Pelargonidin 3-[6-(3-glucosylcaffeyl)glucoside]-5-glucoside |
| − | CAS_RN 154850-21-2 | + | CAS_RN 154850-21-2 |
| − | FORMULA C42H47O23 | + | FORMULA C42H47O23 |
| − | EXACTMASS 919.25081281 | + | EXACTMASS 919.25081281 |
| − | AVERAGEMASS 919.8087800000001 | + | AVERAGEMASS 919.8087800000001 |
| − | SMILES c(c(OC(O7)C(C(O)C(O)C7CO)O)1)(ccc(C=CC(=O)OCC(C2O)OC(Oc(c(c(c6)ccc(c6)O)3)cc(c(OC(C5O)OC(C(O)C5O)CO)4)c(cc(O)c4)[o+1]3)C(C2O)O)c1)O | + | SMILES c(c(OC(O7)C(C(O)C(O)C7CO)O)1)(ccc(C=CC(=O)OCC(C2O)OC(Oc(c(c(c6)ccc(c6)O)3)cc(c(OC(C5O)OC(C(O)C5O)CO)4)c(cc(O)c4)[o+1]3)C(C2O)O)c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
65 71 0 0 0 0 0 0 0 0999 V2000
-1.5421 1.8827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5421 1.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9858 0.9192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4295 1.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4295 1.8827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9858 2.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1268 0.9192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6831 1.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6831 1.8827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1268 2.2039 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
1.2392 2.2038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8062 1.8765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3732 2.2038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3732 2.8585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8062 3.1858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2392 2.8585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0982 2.2038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9400 3.1857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9858 0.2771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0992 0.5197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2566 0.8197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9558 0.2987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5343 0.4641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1163 0.2987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4172 0.8197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8386 0.6545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6979 0.6700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0768 1.0670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8512 0.5691 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6678 0.2987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8584 -0.4125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5690 -0.9137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0538 -0.9137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7595 -1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2702 -1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9810 -0.9226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4026 -0.9226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1134 -1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4026 -1.9245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9810 -1.9245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5359 -1.4236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8646 -1.4258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1138 -2.4246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3708 -2.3896 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9996 -2.8796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4651 -2.6717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1459 -2.8198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3242 -2.2913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7918 -2.5381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8846 -2.6863 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5783 -2.9077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1589 -3.1858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3508 0.1060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9796 -0.3840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4451 -0.1761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8341 -0.3241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3042 0.2043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7718 -0.0425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8646 -0.1907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5583 -0.4121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1389 -0.6902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1902 -1.7539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3933 -1.9674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1702 0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3733 0.5282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
22 20 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
34 35 1 0 0 0 0
35 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 35 1 0 0 0 0
38 41 1 0 0 0 0
32 42 2 0 0 0 0
39 43 1 0 0 0 0
44 45 1 1 0 0 0
45 46 1 1 0 0 0
47 46 1 1 0 0 0
47 48 1 0 0 0 0
48 49 1 0 0 0 0
49 44 1 0 0 0 0
44 50 1 0 0 0 0
45 51 1 0 0 0 0
46 52 1 0 0 0 0
53 54 1 1 0 0 0
54 55 1 1 0 0 0
56 55 1 1 0 0 0
56 57 1 0 0 0 0
57 58 1 0 0 0 0
58 53 1 0 0 0 0
53 59 1 0 0 0 0
54 60 1 0 0 0 0
55 61 1 0 0 0 0
56 19 1 0 0 0 0
47 43 1 0 0 0 0
49 62 1 0 0 0 0
62 63 1 0 0 0 0
58 64 1 0 0 0 0
64 65 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 62 63
M SBL 1 1 68
M SMT 1 ^CH2OH
M SBV 1 68 -7.1339 7.4519
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 64 65
M SBL 2 1 70
M SMT 2 ^CH2OH
M SBV 2 70 -7.1339 7.4519
S SKP 8
ID FL7AAAGL0029
KNApSAcK_ID C00006768
NAME Pelargonidin 3-[6-(3-glucosylcaffeyl)glucoside]-5-glucoside
CAS_RN 154850-21-2
FORMULA C42H47O23
EXACTMASS 919.25081281
AVERAGEMASS 919.8087800000001
SMILES c(c(OC(O7)C(C(O)C(O)C7CO)O)1)(ccc(C=CC(=O)OCC(C2O)OC(Oc(c(c(c6)ccc(c6)O)3)cc(c(OC(C5O)OC(C(O)C5O)CO)4)c(cc(O)c4)[o+1]3)C(C2O)O)c1)O
M END