Mol:FL5FDEGS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | 4.0816 0.4587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0816 0.4587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3671 0.8712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3671 0.8712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3671 1.6962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3671 1.6962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0816 2.1088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0816 2.1088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7960 1.6963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7960 1.6963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7960 0.8713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7960 0.8713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6526 0.4587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6526 0.4587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9382 0.8712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9382 0.8712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2237 0.4587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2237 0.4587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2237 -0.3663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2237 -0.3663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9382 -0.7787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9382 -0.7787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6526 -0.3663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6526 -0.3663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5092 0.8712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5092 0.8712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2053 0.4587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2053 0.4587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2053 -0.3663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2053 -0.3663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5092 -0.7788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5092 -0.7788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9382 -1.6037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9382 -1.6037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9197 0.8712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9197 0.8712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3671 -0.7788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3671 -0.7788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5092 -1.6038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5092 -1.6038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5105 2.1088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5105 2.1088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0816 2.9338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0816 2.9338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0804 -0.3669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0804 -0.3669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2238 1.6969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2238 1.6969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9871 -0.9658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9871 -0.9658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1899 -1.1794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1899 -1.1794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7733 -0.4669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7733 -0.4669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0519 -0.0663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0519 -0.0663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8490 0.1474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8490 0.1474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2657 -0.5650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2657 -0.5650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1229 -0.5527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1229 -0.5527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8609 -1.1177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8609 -1.1177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3201 -1.5426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3201 -1.5426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7654 -1.5116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7654 -1.5116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8472 -0.4669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8472 -0.4669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4137 -2.2913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4137 -2.2913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2297 -2.9338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2297 -2.9338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4137 -1.2826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4137 -1.2826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8619 -1.9313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8619 -1.9313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7790 -1.9313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7790 -1.9313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2297 -2.2913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2297 -2.2913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8198 -1.6726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8198 -1.6726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7790 -2.6215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7790 -2.6215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.2238 -2.8783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.2238 -2.8783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 19 23 1 0 0 0 0 | + | 19 23 1 0 0 0 0 |
| − | 21 24 1 0 0 0 0 | + | 21 24 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 28 27 1 1 0 0 0 | + | 28 27 1 1 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 25 1 0 0 0 0 | + | 30 25 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 26 34 1 0 0 0 0 | + | 26 34 1 0 0 0 0 |
| − | 27 35 1 0 0 0 0 | + | 27 35 1 0 0 0 0 |
| − | 28 18 1 0 0 0 0 | + | 28 18 1 0 0 0 0 |
| − | 41 36 1 1 0 0 0 | + | 41 36 1 1 0 0 0 |
| − | 40 36 1 1 0 0 0 | + | 40 36 1 1 0 0 0 |
| − | 39 41 1 1 0 0 0 | + | 39 41 1 1 0 0 0 |
| − | 41 37 1 0 0 0 0 | + | 41 37 1 0 0 0 0 |
| − | 42 39 1 0 0 0 0 | + | 42 39 1 0 0 0 0 |
| − | 40 42 1 0 0 0 0 | + | 40 42 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
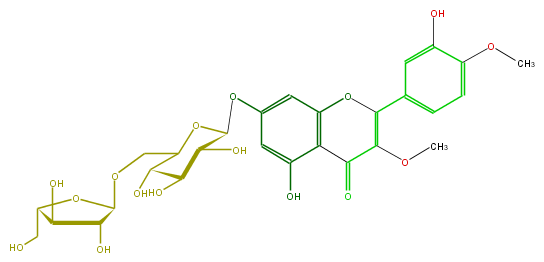
| − | ID FL5FDEGS0002 | + | ID FL5FDEGS0002 |
| − | FORMULA C28H32O16 | + | FORMULA C28H32O16 |
| − | EXACTMASS 624.1690349759999 | + | EXACTMASS 624.1690349759999 |
| − | AVERAGEMASS 624.54408 | + | AVERAGEMASS 624.54408 |
| − | SMILES c(c3)(cc(c(C(=O)4)c3OC(c(c5)cc(O)c(OC)c5)=C4OC)O)OC(O1)C(O)C(O)C(O)C1COC(O2)C(C(C2CO)O)O | + | SMILES c(c3)(cc(c(C(=O)4)c3OC(c(c5)cc(O)c(OC)c5)=C4OC)O)OC(O1)C(O)C(O)C(O)C1COC(O2)C(C(C2CO)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
4.0816 0.4587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3671 0.8712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3671 1.6962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0816 2.1088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7960 1.6963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7960 0.8713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6526 0.4587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9382 0.8712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2237 0.4587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2237 -0.3663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9382 -0.7787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6526 -0.3663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5092 0.8712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2053 0.4587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2053 -0.3663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5092 -0.7788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9382 -1.6037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9197 0.8712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3671 -0.7788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5092 -1.6038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5105 2.1088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0816 2.9338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0804 -0.3669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2238 1.6969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9871 -0.9658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1899 -1.1794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7733 -0.4669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0519 -0.0663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8490 0.1474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2657 -0.5650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1229 -0.5527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8609 -1.1177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3201 -1.5426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7654 -1.5116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8472 -0.4669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.4137 -2.2913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2297 -2.9338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.4137 -1.2826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8619 -1.9313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7790 -1.9313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2297 -2.2913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8198 -1.6726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7790 -2.6215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.2238 -2.8783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
19 23 1 0 0 0 0
21 24 1 0 0 0 0
25 26 1 1 0 0 0
26 27 1 1 0 0 0
28 27 1 1 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
30 25 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
25 33 1 0 0 0 0
26 34 1 0 0 0 0
27 35 1 0 0 0 0
28 18 1 0 0 0 0
41 36 1 1 0 0 0
40 36 1 1 0 0 0
39 41 1 1 0 0 0
41 37 1 0 0 0 0
42 39 1 0 0 0 0
40 42 1 0 0 0 0
36 38 1 0 0 0 0
40 43 1 0 0 0 0
43 44 1 0 0 0 0
32 39 1 0 0 0 0
S SKP 5
ID FL5FDEGS0002
FORMULA C28H32O16
EXACTMASS 624.1690349759999
AVERAGEMASS 624.54408
SMILES c(c3)(cc(c(C(=O)4)c3OC(c(c5)cc(O)c(OC)c5)=C4OC)O)OC(O1)C(O)C(O)C(O)C1COC(O2)C(C(C2CO)O)O
M END