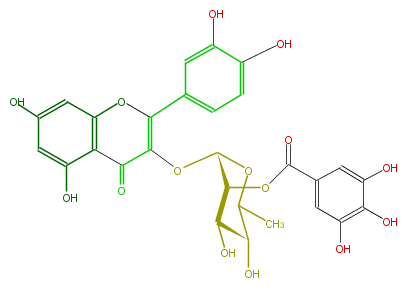
Mol:FL5FACGS0050
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.2350 0.5175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2350 0.5175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2350 -0.1248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2350 -0.1248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6787 -0.4460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6787 -0.4460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1224 -0.1248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1224 -0.1248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1224 0.5175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1224 0.5175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6787 0.8387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6787 0.8387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5661 -0.4460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5661 -0.4460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0098 -0.1248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0098 -0.1248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0098 0.5175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0098 0.5175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5661 0.8387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5661 0.8387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5661 -0.9468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5661 -0.9468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3093 0.9624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3093 0.9624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2577 0.6350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2577 0.6350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8246 0.9624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8246 0.9624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8246 1.6170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8246 1.6170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2577 1.9444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2577 1.9444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3093 1.6170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3093 1.6170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6787 -1.0881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6787 -1.0881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6183 1.9964 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6183 1.9964 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7299 0.8387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7299 0.8387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4465 -0.5343 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4465 -0.5343 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3381 -1.5869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3381 -1.5869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9663 -1.9496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9663 -1.9496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7670 -1.2521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7670 -1.2521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9663 -0.5504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9663 -0.5504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3381 -0.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3381 -0.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5374 -0.8851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5374 -0.8851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3077 -0.8851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3077 -0.8851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4995 -2.1891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4995 -2.1891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9663 -2.5989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9663 -2.5989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3872 -1.6102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3872 -1.6102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2577 2.5989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2577 2.5989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7615 -0.4313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7615 -0.4313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7615 0.0844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7615 0.0844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3282 -0.7585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3282 -0.7585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3282 -1.2983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3282 -1.2983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7957 -1.5682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7957 -1.5682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2632 -1.2983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2632 -1.2983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2632 -0.7585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2632 -0.7585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7957 -0.4886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7957 -0.4886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7299 -0.4890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7299 -0.4890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7299 -1.5678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7299 -1.5678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7957 -2.1072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7957 -2.1072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 1 1 0 0 0 0 | + | 20 1 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 16 32 1 0 0 0 0 | + | 16 32 1 0 0 0 0 |
| − | 28 33 1 0 0 0 0 | + | 28 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 38 42 1 0 0 0 0 | + | 38 42 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGS0050 | + | ID FL5FACGS0050 |
| − | KNApSAcK_ID C00005966 | + | KNApSAcK_ID C00005966 |
| − | NAME Quercetin 3-(2''-galloylrhamnoside) | + | NAME Quercetin 3-(2''-galloylrhamnoside) |
| − | CAS_RN 80229-08-9 | + | CAS_RN 80229-08-9 |
| − | FORMULA C28H24O15 | + | FORMULA C28H24O15 |
| − | EXACTMASS 600.111520098 | + | EXACTMASS 600.111520098 |
| − | AVERAGEMASS 600.48116 | + | AVERAGEMASS 600.48116 |
| − | SMILES c(c31)(C(=O)C(OC(C(OC(c(c5)cc(O)c(O)c(O)5)=O)4)OC(C(O)C(O)4)C)=C(O3)c(c2)cc(c(c2)O)O)c(cc(O)c1)O | + | SMILES c(c31)(C(=O)C(OC(C(OC(c(c5)cc(O)c(O)c(O)5)=O)4)OC(C(O)C(O)4)C)=C(O3)c(c2)cc(c(c2)O)O)c(cc(O)c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-3.2350 0.5175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2350 -0.1248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6787 -0.4460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1224 -0.1248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1224 0.5175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6787 0.8387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5661 -0.4460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0098 -0.1248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0098 0.5175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5661 0.8387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5661 -0.9468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3093 0.9624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2577 0.6350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8246 0.9624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8246 1.6170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2577 1.9444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3093 1.6170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6787 -1.0881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6183 1.9964 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7299 0.8387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4465 -0.5343 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3381 -1.5869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9663 -1.9496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7670 -1.2521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9663 -0.5504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3381 -0.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5374 -0.8851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3077 -0.8851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4995 -2.1891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9663 -2.5989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3872 -1.6102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2577 2.5989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7615 -0.4313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7615 0.0844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3282 -0.7585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3282 -1.2983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7957 -1.5682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2632 -1.2983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2632 -0.7585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7957 -0.4886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7299 -0.4890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7299 -1.5678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7957 -2.1072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 1 1 0 0 0 0
21 8 1 0 0 0 0
4 3 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
26 21 1 0 0 0 0
16 32 1 0 0 0 0
28 33 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
35 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 35 1 0 0 0 0
39 41 1 0 0 0 0
38 42 1 0 0 0 0
37 43 1 0 0 0 0
S SKP 8
ID FL5FACGS0050
KNApSAcK_ID C00005966
NAME Quercetin 3-(2''-galloylrhamnoside)
CAS_RN 80229-08-9
FORMULA C28H24O15
EXACTMASS 600.111520098
AVERAGEMASS 600.48116
SMILES c(c31)(C(=O)C(OC(C(OC(c(c5)cc(O)c(O)c(O)5)=O)4)OC(C(O)C(O)4)C)=C(O3)c(c2)cc(c(c2)O)O)c(cc(O)c1)O
M END