Mol:FL5FACGS0014
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 33 36 0 0 0 0 0 0 0 0999 V2000 | + | 33 36 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.4176 -0.1760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4176 -0.1760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4176 -0.8183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4176 -0.8183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1387 -1.1395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1387 -1.1395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6950 -0.8183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6950 -0.8183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6950 -0.1760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6950 -0.1760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1387 0.1452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1387 0.1452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2513 -1.1395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2513 -1.1395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8076 -0.8183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8076 -0.8183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8076 -0.1760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8076 -0.1760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2513 0.1452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2513 0.1452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2513 -1.6403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2513 -1.6403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3637 0.1451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3637 0.1451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9307 -0.1822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9307 -0.1822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4977 0.1451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4977 0.1451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4977 0.7998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4977 0.7998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9307 1.1271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9307 1.1271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3637 0.7998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3637 0.7998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9737 0.1451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9737 0.1451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1387 -1.7816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1387 -1.7816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9820 1.2167 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9820 1.2167 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3637 -1.1394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3637 -1.1394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9307 1.7816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9307 1.7816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3021 -0.0297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3021 -0.0297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8966 -0.5649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8966 -0.5649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3128 -0.3379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3128 -0.3379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7043 -0.3446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7043 -0.3446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1588 0.0777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1588 0.0777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7552 -0.1365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7552 -0.1365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9820 -0.4223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9820 -0.4223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5287 -0.5957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5287 -0.5957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9783 -0.8994 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9783 -0.8994 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5034 0.4214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5034 0.4214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7890 0.0089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7890 0.0089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 8 21 1 0 0 0 0 | + | 8 21 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 26 18 1 0 0 0 0 | + | 26 18 1 0 0 0 0 |
| − | 28 32 1 0 0 0 0 | + | 28 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 32 33 | + | M SAL 1 2 32 33 |
| − | M SBL 1 1 35 | + | M SBL 1 1 35 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 35 -4.8023 4.2800 | + | M SBV 1 35 -4.8023 4.2800 |
| − | S SKP 8 | + | S SKP 8 |
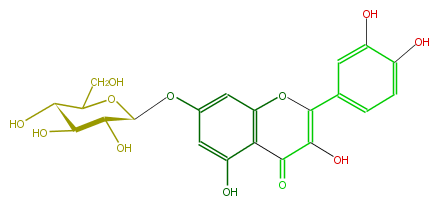
| − | ID FL5FACGS0014 | + | ID FL5FACGS0014 |
| − | KNApSAcK_ID C00005382 | + | KNApSAcK_ID C00005382 |
| − | NAME Quercimeritrin | + | NAME Quercimeritrin |
| − | CAS_RN 491-50-9 | + | CAS_RN 491-50-9 |
| − | FORMULA C21H20O12 | + | FORMULA C21H20O12 |
| − | EXACTMASS 464.095476104 | + | EXACTMASS 464.095476104 |
| − | AVERAGEMASS 464.37629999999996 | + | AVERAGEMASS 464.37629999999996 |
| − | SMILES c(O)(c(O)4)ccc(c4)C(=C3O)Oc(c1)c(C3=O)c(cc(OC(O2)C(O)C(O)C(O)C2CO)1)O | + | SMILES c(O)(c(O)4)ccc(c4)C(=C3O)Oc(c1)c(C3=O)c(cc(OC(O2)C(O)C(O)C(O)C2CO)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
33 36 0 0 0 0 0 0 0 0999 V2000
-0.4176 -0.1760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4176 -0.8183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1387 -1.1395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6950 -0.8183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6950 -0.1760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1387 0.1452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2513 -1.1395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8076 -0.8183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8076 -0.1760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2513 0.1452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2513 -1.6403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3637 0.1451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9307 -0.1822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4977 0.1451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4977 0.7998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9307 1.1271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3637 0.7998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9737 0.1451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1387 -1.7816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9820 1.2167 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3637 -1.1394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9307 1.7816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3021 -0.0297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8966 -0.5649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3128 -0.3379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7043 -0.3446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1588 0.0777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7552 -0.1365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9820 -0.4223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5287 -0.5957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9783 -0.8994 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5034 0.4214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7890 0.0089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
20 15 1 0 0 0 0
8 21 1 0 0 0 0
16 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 31 1 0 0 0 0
26 18 1 0 0 0 0
28 32 1 0 0 0 0
32 33 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 32 33
M SBL 1 1 35
M SMT 1 CH2OH
M SBV 1 35 -4.8023 4.2800
S SKP 8
ID FL5FACGS0014
KNApSAcK_ID C00005382
NAME Quercimeritrin
CAS_RN 491-50-9
FORMULA C21H20O12
EXACTMASS 464.095476104
AVERAGEMASS 464.37629999999996
SMILES c(O)(c(O)4)ccc(c4)C(=C3O)Oc(c1)c(C3=O)c(cc(OC(O2)C(O)C(O)C(O)C2CO)1)O
M END