Mol:FL5FACGL0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.1319 0.6340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1319 0.6340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1319 -0.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1319 -0.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4174 -0.6034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4174 -0.6034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7030 -0.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7030 -0.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7030 0.6340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7030 0.6340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4174 1.0465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4174 1.0465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9885 -0.6034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9885 -0.6034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2740 -0.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2740 -0.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2740 0.6340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2740 0.6340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9885 1.0465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9885 1.0465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9885 -1.2467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9885 -1.2467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4402 1.0464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4402 1.0464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1684 0.6259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1684 0.6259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8966 1.0464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8966 1.0464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8966 1.8872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8966 1.8872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1684 2.3076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1684 2.3076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4402 1.8872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4402 1.8872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8461 1.0464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8461 1.0464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4174 -1.4281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4174 -1.4281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3841 2.1688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3841 2.1688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6173 -0.5469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6173 -0.5469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2151 -1.2436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2151 -1.2436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9886 -1.0226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9886 -1.0226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7669 -1.2436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7669 -1.2436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1693 -0.5469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1693 -0.5469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3956 -0.7679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3956 -0.7679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0877 -0.5895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0877 -0.5895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8599 -0.2794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8599 -0.2794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8461 -0.7021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8461 -0.7021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4554 -1.1781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4554 -1.1781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0696 -2.3900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0696 -2.3900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7867 -2.9472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7867 -2.9472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4699 -2.4463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4699 -2.4463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2037 -2.2411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2037 -2.2411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5255 -1.8495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5255 -1.8495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8214 -2.3380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8214 -2.3380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0916 -1.7451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0916 -1.7451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1175 -3.1482 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1175 -3.1482 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3110 -2.7135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3110 -2.7135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7346 -1.7284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7346 -1.7284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5275 -0.9566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5275 -0.9566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1684 3.1482 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1684 3.1482 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 40 30 1 0 0 0 0 | + | 40 30 1 0 0 0 0 |
| − | 22 41 1 0 0 0 0 | + | 22 41 1 0 0 0 0 |
| − | 41 8 1 0 0 0 0 | + | 41 8 1 0 0 0 0 |
| − | 16 42 1 0 0 0 0 | + | 16 42 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGL0004 | + | ID FL5FACGL0004 |
| − | FORMULA C26H28O16 | + | FORMULA C26H28O16 |
| − | EXACTMASS 596.137734848 | + | EXACTMASS 596.137734848 |
| − | AVERAGEMASS 596.49092 | + | AVERAGEMASS 596.49092 |
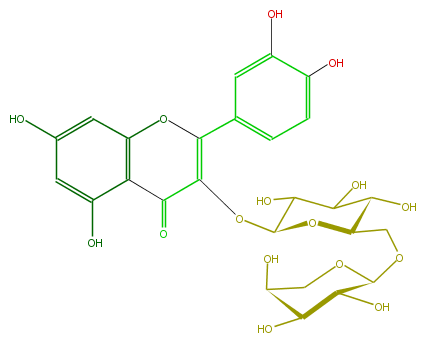
| − | SMILES C(O)(C(O)4)C(O)C(OC(COC(O5)C(O)C(C(O)C5)O)4)OC(=C1c(c3)ccc(O)c3O)C(c(c(O)2)c(cc(O)c2)O1)=O | + | SMILES C(O)(C(O)4)C(O)C(OC(COC(O5)C(O)C(C(O)C5)O)4)OC(=C1c(c3)ccc(O)c3O)C(c(c(O)2)c(cc(O)c2)O1)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-3.1319 0.6340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1319 -0.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4174 -0.6034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7030 -0.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7030 0.6340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4174 1.0465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9885 -0.6034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2740 -0.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2740 0.6340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9885 1.0465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9885 -1.2467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4402 1.0464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1684 0.6259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8966 1.0464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8966 1.8872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1684 2.3076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4402 1.8872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8461 1.0464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4174 -1.4281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3841 2.1688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6173 -0.5469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2151 -1.2436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9886 -1.0226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7669 -1.2436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1693 -0.5469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3956 -0.7679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0877 -0.5895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8599 -0.2794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8461 -0.7021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4554 -1.1781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0696 -2.3900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7867 -2.9472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4699 -2.4463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2037 -2.2411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5255 -1.8495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8214 -2.3380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0916 -1.7451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1175 -3.1482 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3110 -2.7135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7346 -1.7284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5275 -0.9566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1684 3.1482 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
20 15 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
40 30 1 0 0 0 0
22 41 1 0 0 0 0
41 8 1 0 0 0 0
16 42 1 0 0 0 0
S SKP 5
ID FL5FACGL0004
FORMULA C26H28O16
EXACTMASS 596.137734848
AVERAGEMASS 596.49092
SMILES C(O)(C(O)4)C(O)C(OC(COC(O5)C(O)C(C(O)C5)O)4)OC(=C1c(c3)ccc(O)c3O)C(c(c(O)2)c(cc(O)c2)O1)=O
M END