Mol:FL5FACGA0013
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.2159 -0.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2159 -0.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2159 -0.8802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2159 -0.8802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6596 -1.2014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6596 -1.2014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1033 -0.8802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1033 -0.8802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1033 -0.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1033 -0.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6596 0.0833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6596 0.0833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4530 -1.2014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4530 -1.2014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0093 -0.8802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0093 -0.8802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0093 -0.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0093 -0.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4530 0.0833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4530 0.0833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4530 -1.7022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4530 -1.7022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7097 0.2070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7097 0.2070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2767 -0.1204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2767 -0.1204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8437 0.2070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8437 0.2070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8437 0.8617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8437 0.8617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2767 1.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2767 1.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7097 0.8617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7097 0.8617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6596 -1.8435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6596 -1.8435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4505 1.2120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4505 1.2120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5726 -1.2897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5726 -1.2897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8804 0.1458 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8804 0.1458 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2767 1.8435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2767 1.8435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9727 0.0962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9727 0.0962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6015 -0.3937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6015 -0.3937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0670 -0.1859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0670 -0.1859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5513 -0.1803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5513 -0.1803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9261 0.1946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9261 0.1946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3937 -0.0522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3937 -0.0522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4480 -0.1782 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4480 -0.1782 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1802 -0.4219 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1802 -0.4219 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7608 -0.7000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7608 -0.7000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5209 -0.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5209 -0.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2201 -1.3887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2201 -1.3887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7986 -1.2234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7986 -1.2234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3806 -1.3887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3806 -1.3887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6814 -0.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6814 -0.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1029 -1.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1029 -1.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9622 -1.0174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9622 -1.0174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3411 -0.6204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3411 -0.6204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6814 -0.1553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6814 -0.1553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4323 0.3462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4323 0.3462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2292 0.5597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2292 0.5597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7335 -1.3620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7335 -1.3620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4480 -1.7745 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4480 -1.7745 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 21 1 0 0 0 0 | + | 1 21 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 33 20 1 0 0 0 0 | + | 33 20 1 0 0 0 0 |
| − | 28 41 1 0 0 0 0 | + | 28 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 45 -5.9117 7.5650 | + | M SBV 1 45 -5.9117 7.5650 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 43 44 | + | M SAL 2 2 43 44 |
| − | M SBL 2 1 47 | + | M SBL 2 1 47 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 47 -5.5201 7.1932 | + | M SBV 2 47 -5.5201 7.1932 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGA0013 | + | ID FL5FACGA0013 |
| − | KNApSAcK_ID C00005424 | + | KNApSAcK_ID C00005424 |
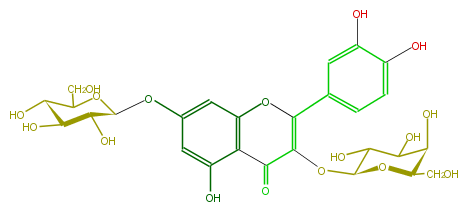
| − | NAME Quercetin 3-galactoside-7-glucoside | + | NAME Quercetin 3-galactoside-7-glucoside |
| − | CAS_RN 56782-99-1,174175-14-5 | + | CAS_RN 56782-99-1,174175-14-5 |
| − | FORMULA C27H30O17 | + | FORMULA C27H30O17 |
| − | EXACTMASS 626.148299534 | + | EXACTMASS 626.148299534 |
| − | AVERAGEMASS 626.5169000000001 | + | AVERAGEMASS 626.5169000000001 |
| − | SMILES C(C(O)5)(OC(CO)C(O)C5O)Oc(c4)cc(c(c4O)1)OC(c(c3)ccc(O)c3O)=C(OC(C(O)2)OC(C(C(O)2)O)CO)C(=O)1 | + | SMILES C(C(O)5)(OC(CO)C(O)C5O)Oc(c4)cc(c(c4O)1)OC(c(c3)ccc(O)c3O)=C(OC(C(O)2)OC(C(C(O)2)O)CO)C(=O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-1.2159 -0.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2159 -0.8802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6596 -1.2014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1033 -0.8802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1033 -0.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6596 0.0833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4530 -1.2014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0093 -0.8802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0093 -0.2378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4530 0.0833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4530 -1.7022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7097 0.2070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2767 -0.1204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8437 0.2070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8437 0.8617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2767 1.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7097 0.8617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6596 -1.8435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4505 1.2120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5726 -1.2897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8804 0.1458 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2767 1.8435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9727 0.0962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6015 -0.3937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0670 -0.1859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5513 -0.1803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9261 0.1946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3937 -0.0522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4480 -0.1782 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1802 -0.4219 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7608 -0.7000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5209 -0.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2201 -1.3887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7986 -1.2234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3806 -1.3887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6814 -0.8677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1029 -1.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9622 -1.0174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3411 -0.6204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6814 -0.1553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4323 0.3462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2292 0.5597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7335 -1.3620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4480 -1.7745 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 8 1 0 0 0 0
4 3 1 0 0 0 0
1 21 1 0 0 0 0
16 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 31 1 0 0 0 0
26 21 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
37 39 1 0 0 0 0
36 40 1 0 0 0 0
33 20 1 0 0 0 0
28 41 1 0 0 0 0
41 42 1 0 0 0 0
35 43 1 0 0 0 0
43 44 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 45
M SMT 1 ^CH2OH
M SBV 1 45 -5.9117 7.5650
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 43 44
M SBL 2 1 47
M SMT 2 CH2OH
M SBV 2 47 -5.5201 7.1932
S SKP 8
ID FL5FACGA0013
KNApSAcK_ID C00005424
NAME Quercetin 3-galactoside-7-glucoside
CAS_RN 56782-99-1,174175-14-5
FORMULA C27H30O17
EXACTMASS 626.148299534
AVERAGEMASS 626.5169000000001
SMILES C(C(O)5)(OC(CO)C(O)C5O)Oc(c4)cc(c(c4O)1)OC(c(c3)ccc(O)c3O)=C(OC(C(O)2)OC(C(C(O)2)O)CO)C(=O)1
M END