Mol:FL5FAAGL0042
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 53 58 0 0 0 0 0 0 0 0999 V2000 | + | 53 58 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.0060 1.5675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0060 1.5675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0060 0.9251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0060 0.9251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4497 0.6039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4497 0.6039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1066 0.9251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1066 0.9251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1066 1.5675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1066 1.5675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4497 1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4497 1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6629 0.6039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6629 0.6039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2192 0.9251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2192 0.9251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2192 1.5675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2192 1.5675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6629 1.8887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6629 1.8887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6629 0.1031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6629 0.1031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7753 1.8885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7753 1.8885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3423 1.5612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3423 1.5612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9093 1.8885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9093 1.8885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9093 2.5432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9093 2.5432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3423 2.8706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3423 2.8706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7753 2.5432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7753 2.5432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4497 -0.0382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4497 -0.0382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5621 1.8885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5621 1.8885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4761 2.8705 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4761 2.8705 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6134 0.5362 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6134 0.5362 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4020 -0.4909 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.4020 -0.4909 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.7306 -0.1033 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.7306 -0.1033 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.9436 -0.8488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9436 -0.8488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7306 -1.5987 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.7306 -1.5987 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.4020 -1.9865 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.4020 -1.9865 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.1890 -1.2409 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.1890 -1.2409 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.2091 0.2290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2091 0.2290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7207 -1.5479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7207 -1.5479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7378 -2.9284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7378 -2.9284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2792 -0.5928 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 3.2792 -0.5928 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 4.0046 -0.5928 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.0046 -0.5928 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.4832 -0.0885 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.4832 -0.0885 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 3.3050 0.6189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3050 0.6189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5795 0.6190 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.5795 0.6190 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.1009 0.1145 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.1009 0.1145 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.7680 0.4997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7680 0.4997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7200 -1.0337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7200 -1.0337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1562 -1.1585 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1562 -1.1585 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1994 -0.0885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1994 -0.0885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7461 1.7344 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.7461 1.7344 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.3406 1.1992 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.3406 1.1992 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.7568 1.4262 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.7568 1.4262 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.1483 1.4195 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.1483 1.4195 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.6028 1.8418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6028 1.8418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1992 1.6276 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.1992 1.6276 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.2674 1.7800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2674 1.7800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7233 0.6953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7233 0.6953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4223 0.8647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4223 0.8647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2123 -2.4539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2123 -2.4539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2125 -2.4326 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2125 -2.4326 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4198 2.3336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4198 2.3336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3955 2.5526 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3955 2.5526 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 8 21 1 0 0 0 0 | + | 8 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 23 21 1 0 0 0 0 | + | 23 21 1 0 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 36 31 1 1 0 0 0 | + | 36 31 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 31 38 1 0 0 0 0 | + | 31 38 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 33 40 1 0 0 0 0 | + | 33 40 1 0 0 0 0 |
| − | 35 28 1 0 0 0 0 | + | 35 28 1 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 41 1 0 0 0 0 | + | 46 41 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 19 1 0 0 0 0 | + | 44 19 1 0 0 0 0 |
| − | 25 50 1 0 0 0 0 | + | 25 50 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 46 52 1 0 0 0 0 | + | 46 52 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 52 53 | + | M SAL 2 2 52 53 |
| − | M SBL 2 1 57 | + | M SBL 2 1 57 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 57 -3.4198 2.3336 | + | M SVB 2 57 -3.4198 2.3336 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 50 51 | + | M SAL 1 2 50 51 |
| − | M SBL 1 1 55 | + | M SBL 1 1 55 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 55 2.2047 -2.2872 | + | M SVB 1 55 2.2047 -2.2872 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAAGL0042 | + | ID FL5FAAGL0042 |
| − | KNApSAcK_ID C00005236 | + | KNApSAcK_ID C00005236 |
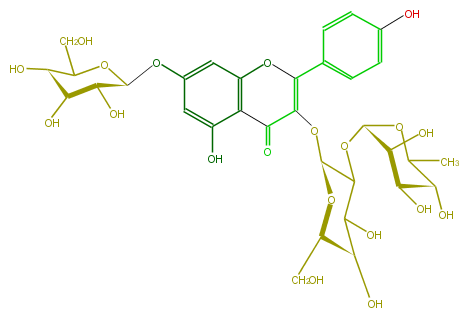
| − | NAME Kaempferol 3-neohesperidoside-7-glucoside | + | NAME Kaempferol 3-neohesperidoside-7-glucoside |
| − | CAS_RN 78527-48-7 | + | CAS_RN 78527-48-7 |
| − | FORMULA C33H40O20 | + | FORMULA C33H40O20 |
| − | EXACTMASS 756.21129372 | + | EXACTMASS 756.21129372 |
| − | AVERAGEMASS 756.6587 | + | AVERAGEMASS 756.6587 |
| − | SMILES Oc(c1)ccc(C(O2)=C(O[C@H](O6)C(C(O)[C@@H](O)[C@H]6CO)O[C@@H](O5)[C@@H](O)[C@H](O)[C@H](O)C5C)C(c(c3O)c2cc(O[C@@H]([C@@H](O)4)OC(CO)[C@H](O)[C@@H]4O)c3)=O)c1 | + | SMILES Oc(c1)ccc(C(O2)=C(O[C@H](O6)C(C(O)[C@@H](O)[C@H]6CO)O[C@@H](O5)[C@@H](O)[C@H](O)[C@H](O)C5C)C(c(c3O)c2cc(O[C@@H]([C@@H](O)4)OC(CO)[C@H](O)[C@@H]4O)c3)=O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
53 58 0 0 0 0 0 0 0 0999 V2000
-1.0060 1.5675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0060 0.9251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4497 0.6039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1066 0.9251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1066 1.5675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4497 1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6629 0.6039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2192 0.9251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2192 1.5675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6629 1.8887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6629 0.1031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7753 1.8885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3423 1.5612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9093 1.8885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9093 2.5432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3423 2.8706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7753 2.5432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4497 -0.0382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5621 1.8885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4761 2.8705 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6134 0.5362 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4020 -0.4909 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.7306 -0.1033 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.9436 -0.8488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7306 -1.5987 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.4020 -1.9865 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.1890 -1.2409 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.2091 0.2290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7207 -1.5479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7378 -2.9284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2792 -0.5928 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
4.0046 -0.5928 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.4832 -0.0885 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
3.3050 0.6189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5795 0.6190 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.1009 0.1145 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.7680 0.4997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7200 -1.0337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1562 -1.1585 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1994 -0.0885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7461 1.7344 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.3406 1.1992 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.7568 1.4262 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.1483 1.4195 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.6028 1.8418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1992 1.6276 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.2674 1.7800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7233 0.6953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4223 0.8647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2123 -2.4539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2125 -2.4326 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4198 2.3336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3955 2.5526 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
15 20 1 0 0 0 0
8 21 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
23 21 1 0 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 1 0 0 0
36 31 1 1 0 0 0
36 37 1 0 0 0 0
31 38 1 0 0 0 0
32 39 1 0 0 0 0
33 40 1 0 0 0 0
35 28 1 0 0 0 0
41 42 1 1 0 0 0
42 43 1 1 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
46 41 1 0 0 0 0
41 47 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
44 19 1 0 0 0 0
25 50 1 0 0 0 0
50 51 1 0 0 0 0
46 52 1 0 0 0 0
52 53 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 52 53
M SBL 2 1 57
M SMT 2 CH2OH
M SVB 2 57 -3.4198 2.3336
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 50 51
M SBL 1 1 55
M SMT 1 CH2OH
M SVB 1 55 2.2047 -2.2872
S SKP 8
ID FL5FAAGL0042
KNApSAcK_ID C00005236
NAME Kaempferol 3-neohesperidoside-7-glucoside
CAS_RN 78527-48-7
FORMULA C33H40O20
EXACTMASS 756.21129372
AVERAGEMASS 756.6587
SMILES Oc(c1)ccc(C(O2)=C(O[C@H](O6)C(C(O)[C@@H](O)[C@H]6CO)O[C@@H](O5)[C@@H](O)[C@H](O)[C@H](O)C5C)C(c(c3O)c2cc(O[C@@H]([C@@H](O)4)OC(CO)[C@H](O)[C@@H]4O)c3)=O)c1
M END