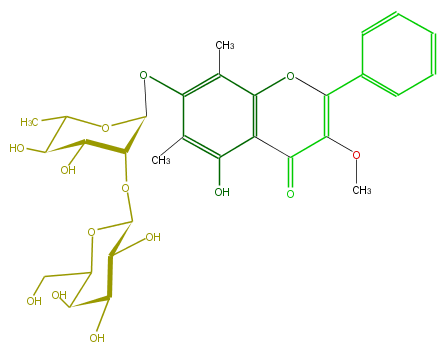
Mol:FL5FA9GM0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | 3.4107 1.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4107 1.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6963 2.0018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6963 2.0018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6963 2.8268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6963 2.8268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4107 3.2394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4107 3.2394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1252 2.8268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1252 2.8268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1252 2.0018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1252 2.0018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9819 1.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9819 1.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2674 2.0018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2674 2.0018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5528 1.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5528 1.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5528 0.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5528 0.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2674 0.3519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2674 0.3519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9819 0.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9819 0.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1616 2.0018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1616 2.0018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8760 1.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8760 1.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8760 0.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8760 0.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1616 0.3519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1616 0.3519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2674 -0.3552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2674 -0.3552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5909 0.4127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5909 0.4127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1616 -0.3237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1616 -0.3237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6563 2.0211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6563 2.0211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1602 2.6192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1602 2.6192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4271 0.3260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4271 0.3260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5909 -0.2739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5909 -0.2739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2164 1.1604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2164 1.1604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6290 0.4458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6290 0.4458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8355 0.6725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8355 0.6725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0372 0.4633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0372 0.4633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6245 1.1779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6245 1.1779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4181 0.9512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4181 0.9512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0212 -0.2346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0212 -0.2346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2485 0.1105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2485 0.1105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1252 0.5547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1252 0.5547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8271 1.0775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8271 1.0775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1571 -2.6475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1571 -2.6475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3560 -2.4491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3560 -2.4491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3357 -1.6241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3357 -1.6241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8978 -0.9247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8978 -0.9247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6989 -1.1229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6989 -1.1229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7194 -1.9480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7194 -1.9480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6601 -2.0245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6601 -2.0245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9274 -2.5095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9274 -2.5095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4232 -2.3747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4232 -2.3747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6564 -3.2394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6564 -3.2394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5469 -1.2137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5469 -1.2137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 18 1 0 0 0 0 | + | 12 18 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 13 21 1 0 0 0 0 | + | 13 21 1 0 0 0 0 |
| − | 15 22 1 0 0 0 0 | + | 15 22 1 0 0 0 0 |
| − | 18 23 1 0 0 0 0 | + | 18 23 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 28 27 1 1 0 0 0 | + | 28 27 1 1 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 26 31 1 0 0 0 0 | + | 26 31 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 24 33 1 0 0 0 0 | + | 24 33 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 36 44 1 0 0 0 0 | + | 36 44 1 0 0 0 0 |
| − | 28 20 1 0 0 0 0 | + | 28 20 1 0 0 0 0 |
| − | 37 30 1 0 0 0 0 | + | 37 30 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FA9GM0001 | + | ID FL5FA9GM0001 |
| − | FORMULA C30H36O14 | + | FORMULA C30H36O14 |
| − | EXACTMASS 620.21050586 | + | EXACTMASS 620.21050586 |
| − | AVERAGEMASS 620.59844 | + | AVERAGEMASS 620.59844 |
| − | SMILES c(c(C)3)(c(C(=O)1)c(c(c3OC(C4OC(C(O)5)OC(CO)C(O)C(O)5)OC(C(C4O)O)C)C)OC(c(c2)cccc2)=C1OC)O | + | SMILES c(c(C)3)(c(C(=O)1)c(c(c3OC(C4OC(C(O)5)OC(CO)C(O)C(O)5)OC(C(C4O)O)C)C)OC(c(c2)cccc2)=C1OC)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
3.4107 1.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6963 2.0018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6963 2.8268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4107 3.2394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1252 2.8268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1252 2.0018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9819 1.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2674 2.0018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5528 1.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5528 0.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2674 0.3519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9819 0.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1616 2.0018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8760 1.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8760 0.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1616 0.3519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2674 -0.3552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5909 0.4127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1616 -0.3237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6563 2.0211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1602 2.6192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4271 0.3260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5909 -0.2739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2164 1.1604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6290 0.4458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8355 0.6725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0372 0.4633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6245 1.1779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4181 0.9512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0212 -0.2346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2485 0.1105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1252 0.5547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8271 1.0775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1571 -2.6475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3560 -2.4491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3357 -1.6241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8978 -0.9247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6989 -1.1229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7194 -1.9480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6601 -2.0245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9274 -2.5095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4232 -2.3747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6564 -3.2394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5469 -1.2137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
11 17 2 0 0 0 0
12 18 1 0 0 0 0
16 19 1 0 0 0 0
14 20 1 0 0 0 0
13 21 1 0 0 0 0
15 22 1 0 0 0 0
18 23 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
28 27 1 1 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
27 30 1 0 0 0 0
26 31 1 0 0 0 0
25 32 1 0 0 0 0
24 33 1 0 0 0 0
34 35 1 1 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
34 42 1 0 0 0 0
35 43 1 0 0 0 0
36 44 1 0 0 0 0
28 20 1 0 0 0 0
37 30 1 0 0 0 0
S SKP 5
ID FL5FA9GM0001
FORMULA C30H36O14
EXACTMASS 620.21050586
AVERAGEMASS 620.59844
SMILES c(c(C)3)(c(C(=O)1)c(c(c3OC(C4OC(C(O)5)OC(CO)C(O)C(O)5)OC(C(C4O)O)C)C)OC(c(c2)cccc2)=C1OC)O
M END