Mol:FL3FFCGSS002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 38 41 0 0 0 0 0 0 0 0999 V2000 | + | 38 41 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.8288 -1.1928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8288 -1.1928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8288 -2.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8288 -2.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1280 -2.4710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1280 -2.4710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4270 -2.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4270 -2.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4270 -1.2568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4270 -1.2568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1280 -0.8522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1280 -0.8522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2740 -2.4710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2740 -2.4710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9748 -2.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9748 -2.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9748 -1.2568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9748 -1.2568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2740 -0.8522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2740 -0.8522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2740 -3.1020 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2740 -3.1020 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6755 -0.8524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6755 -0.8524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3900 -1.2648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3900 -1.2648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1044 -0.8524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1044 -0.8524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1044 -0.0274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1044 -0.0274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3900 0.3851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3900 0.3851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6755 -0.0274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6755 -0.0274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1280 -3.2786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1280 -3.2786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5326 -0.7283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5326 -0.7283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1280 0.0466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1280 0.0466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2025 2.8358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2025 2.8358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2686 1.5106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2686 1.5106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0962 1.1126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0962 1.1126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2357 0.4062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2357 0.4062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3353 1.5461 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3353 1.5461 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5101 1.9795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5101 1.9795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1359 2.9508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1359 2.9508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0362 2.0180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0362 2.0180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7228 0.1236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7228 0.1236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3070 1.3470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3070 1.3470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3070 2.3696 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3070 2.3696 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1086 2.3696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1086 2.3696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3070 3.2786 0.0000 O 0 5 0 0 0 0 0 0 0 0 0 0 | + | 2.3070 3.2786 0.0000 O 0 5 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2480 2.3696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2480 2.3696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0256 0.6402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0256 0.6402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1359 -0.0008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1359 -0.0008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6920 3.1755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6920 3.1755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5817 2.5345 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5817 2.5345 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 6 20 1 0 0 0 0 | + | 6 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 20 1 0 0 0 0 | + | 24 20 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 31 33 1 0 0 0 0 | + | 31 33 1 0 0 0 0 |
| − | 31 34 2 0 0 0 0 | + | 31 34 2 0 0 0 0 |
| − | 30 16 1 0 0 0 0 | + | 30 16 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 15 35 1 0 0 0 0 | + | 15 35 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 26 37 1 0 0 0 0 | + | 26 37 1 0 0 0 0 |
| − | M CHG 1 33 -1 | + | M CHG 1 33 -1 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 35 36 | + | M SAL 1 2 35 36 |
| − | M SBL 1 1 39 | + | M SBL 1 1 39 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 39 -0.9212 -0.6676 | + | M SBV 1 39 -0.9212 -0.6676 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 37 38 | + | M SAL 2 2 37 38 |
| − | M SBL 2 1 41 | + | M SBL 2 1 41 |
| − | M SMT 2 ^CH2OH | + | M SMT 2 ^CH2OH |
| − | M SBV 2 41 0.1819 -1.1959 | + | M SBV 2 41 0.1819 -1.1959 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FFCGSS002 | + | ID FL3FFCGSS002 |
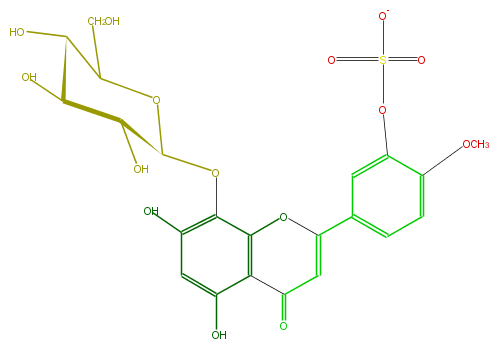
| − | FORMULA C22H21O15S | + | FORMULA C22H21O15S |
| − | EXACTMASS 557.060115692 | + | EXACTMASS 557.060115692 |
| − | AVERAGEMASS 557.45914 | + | AVERAGEMASS 557.45914 |
| − | SMILES Oc(c24)cc(c(c2OC(=CC4=O)c(c3)cc(OS([O-1])(=O)=O)c(OC)c3)OC(O1)C(O)C(C(C(CO)1)O)O)O | + | SMILES Oc(c24)cc(c(c2OC(=CC4=O)c(c3)cc(OS([O-1])(=O)=O)c(OC)c3)OC(O1)C(O)C(C(C(CO)1)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
38 41 0 0 0 0 0 0 0 0999 V2000
-1.8288 -1.1928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8288 -2.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1280 -2.4710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4270 -2.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4270 -1.2568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1280 -0.8522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2740 -2.4710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9748 -2.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9748 -1.2568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2740 -0.8522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2740 -3.1020 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6755 -0.8524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3900 -1.2648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1044 -0.8524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1044 -0.0274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3900 0.3851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6755 -0.0274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1280 -3.2786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5326 -0.7283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1280 0.0466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2025 2.8358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2686 1.5106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0962 1.1126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2357 0.4062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3353 1.5461 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5101 1.9795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1359 2.9508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0362 2.0180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7228 0.1236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3070 1.3470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3070 2.3696 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
3.1086 2.3696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3070 3.2786 0.0000 O 0 5 0 0 0 0 0 0 0 0 0 0
1.2480 2.3696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0256 0.6402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1359 -0.0008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6920 3.1755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5817 2.5345 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
6 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 20 1 0 0 0 0
30 31 1 0 0 0 0
31 32 2 0 0 0 0
31 33 1 0 0 0 0
31 34 2 0 0 0 0
30 16 1 0 0 0 0
35 36 1 0 0 0 0
15 35 1 0 0 0 0
37 38 1 0 0 0 0
26 37 1 0 0 0 0
M CHG 1 33 -1
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 35 36
M SBL 1 1 39
M SMT 1 OCH3
M SBV 1 39 -0.9212 -0.6676
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 37 38
M SBL 2 1 41
M SMT 2 ^CH2OH
M SBV 2 41 0.1819 -1.1959
S SKP 5
ID FL3FFCGSS002
FORMULA C22H21O15S
EXACTMASS 557.060115692
AVERAGEMASS 557.45914
SMILES Oc(c24)cc(c(c2OC(=CC4=O)c(c3)cc(OS([O-1])(=O)=O)c(OC)c3)OC(O1)C(O)C(C(C(CO)1)O)O)O
M END