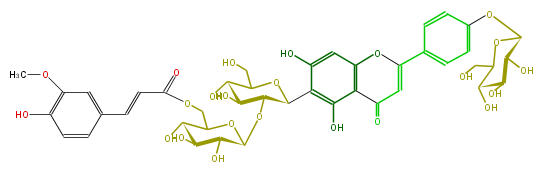
Mol:FL3FAADS0047
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 66 72 0 0 0 0 0 0 0 0999 V2000 | + | 66 72 0 0 0 0 0 0 0 0999 V2000 |
| − | 4.1085 0.6547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1085 0.6547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3940 1.0672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3940 1.0672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6795 0.6547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6795 0.6547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6795 -0.1703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6795 -0.1703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3940 -0.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3940 -0.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1085 -0.1703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1085 -0.1703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9651 1.0672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9651 1.0672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2506 0.6547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2506 0.6547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2506 -0.1703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2506 -0.1703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9651 -0.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9651 -0.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3940 -1.1549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3940 -1.1549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9098 1.1174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9098 1.1174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6243 0.7049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6243 0.7049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3388 1.1174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3388 1.1174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3388 1.9424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3388 1.9424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6243 2.3549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6243 2.3549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9098 1.9424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9098 1.9424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0222 2.3369 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0222 2.3369 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9651 -1.3528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9651 -1.3528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5601 1.0533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5601 1.0533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4889 0.1436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4889 0.1436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0762 -0.5711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0762 -0.5711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2779 -0.3619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2779 -0.3619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5156 -0.5888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5156 -0.5888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1030 0.1260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1030 0.1260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6954 -0.0831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6954 -0.0831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8524 0.5028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8524 0.5028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9721 -0.3396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9721 -0.3396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5249 -0.3120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5249 -0.3120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4494 -1.0019 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4494 -1.0019 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3132 0.7688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3132 0.7688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5069 -0.2088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5069 -0.2088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1328 0.4391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1328 0.4391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3308 -0.2088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3308 -0.2088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7427 -0.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7427 -0.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5666 -0.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5666 -0.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9791 -0.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9791 -0.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.8041 -0.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.8041 -0.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.2166 -0.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.2166 -0.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.8041 -1.6367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.8041 -1.6367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9791 -1.6367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9791 -1.6367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -8.0405 -0.9223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -8.0405 -0.9223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.3823 0.3704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.3823 0.3704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -8.1084 0.3704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -8.1084 0.3704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6715 -0.0575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6715 -0.0575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4855 0.0788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4855 0.0788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5690 0.8999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5690 0.8999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0592 1.5638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0592 1.5638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2453 1.4275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2453 1.4275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1617 0.6064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1617 0.6064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5579 0.6644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5579 0.6644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1305 0.3119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1305 0.3119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9550 -0.6793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9550 -0.6793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0857 -0.2508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0857 -0.2508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1084 0.5153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1084 0.5153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9227 -1.2094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9227 -1.2094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5101 -1.9241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5101 -1.9241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7117 -1.7149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7117 -1.7149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9182 -1.9418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9182 -1.9418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3308 -1.2270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3308 -1.2270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1292 -1.4361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1292 -1.4361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2862 -0.8502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2862 -0.8502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7660 -0.5732 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7660 -0.5732 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4059 -1.6926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4059 -1.6926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9587 -1.6650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9587 -1.6650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8832 -2.3549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8832 -2.3549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 5 11 2 0 0 0 0 | + | 5 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 15 18 1 0 0 0 0 | + | 15 18 1 0 0 0 0 |
| − | 10 19 1 0 0 0 0 | + | 10 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 21 28 1 0 0 0 0 | + | 21 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 9 1 0 0 0 0 | + | 24 9 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 38 43 1 0 0 0 0 | + | 38 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 48 47 1 1 0 0 0 | + | 48 47 1 1 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 45 53 1 0 0 0 0 | + | 45 53 1 0 0 0 0 |
| − | 46 54 1 0 0 0 0 | + | 46 54 1 0 0 0 0 |
| − | 47 55 1 0 0 0 0 | + | 47 55 1 0 0 0 0 |
| − | 48 18 1 0 0 0 0 | + | 48 18 1 0 0 0 0 |
| − | 56 57 1 1 0 0 0 | + | 56 57 1 1 0 0 0 |
| − | 57 58 1 1 0 0 0 | + | 57 58 1 1 0 0 0 |
| − | 59 58 1 1 0 0 0 | + | 59 58 1 1 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 61 56 1 0 0 0 0 | + | 61 56 1 0 0 0 0 |
| − | 61 62 1 0 0 0 0 | + | 61 62 1 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | 56 64 1 0 0 0 0 | + | 56 64 1 0 0 0 0 |
| − | 57 65 1 0 0 0 0 | + | 57 65 1 0 0 0 0 |
| − | 58 66 1 0 0 0 0 | + | 58 66 1 0 0 0 0 |
| − | 59 30 1 0 0 0 0 | + | 59 30 1 0 0 0 0 |
| − | 32 63 1 0 0 0 0 | + | 32 63 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAADS0047 | + | ID FL3FAADS0047 |
| − | KNApSAcK_ID C00014083 | + | KNApSAcK_ID C00014083 |
| − | NAME Isovitexin 2''-O-(6'''-(E)-feruloyl)glucoside 4'-O-glucoside | + | NAME Isovitexin 2''-O-(6'''-(E)-feruloyl)glucoside 4'-O-glucoside |
| − | CAS_RN 372113-57-0 | + | CAS_RN 372113-57-0 |
| − | FORMULA C43H48O23 | + | FORMULA C43H48O23 |
| − | EXACTMASS 932.258637842 | + | EXACTMASS 932.258637842 |
| − | AVERAGEMASS 932.8274200000001 | + | AVERAGEMASS 932.8274200000001 |
| − | SMILES C(OC(C(c(c7O)c(c(C6=O)c(c7)OC(=C6)c(c5)ccc(c5)OC(O4)C(O)C(O)C(C4CO)O)O)3)C(C(O)C(O3)CO)O)(O1)C(O)C(O)C(O)C1COC(=O)C=Cc(c2)cc(c(O)c2)OC | + | SMILES C(OC(C(c(c7O)c(c(C6=O)c(c7)OC(=C6)c(c5)ccc(c5)OC(O4)C(O)C(O)C(C4CO)O)O)3)C(C(O)C(O3)CO)O)(O1)C(O)C(O)C(O)C1COC(=O)C=Cc(c2)cc(c(O)c2)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
66 72 0 0 0 0 0 0 0 0999 V2000
4.1085 0.6547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3940 1.0672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6795 0.6547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6795 -0.1703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3940 -0.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1085 -0.1703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9651 1.0672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2506 0.6547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2506 -0.1703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9651 -0.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3940 -1.1549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9098 1.1174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6243 0.7049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3388 1.1174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3388 1.9424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6243 2.3549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9098 1.9424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.0222 2.3369 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9651 -1.3528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5601 1.0533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4889 0.1436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0762 -0.5711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2779 -0.3619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5156 -0.5888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1030 0.1260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6954 -0.0831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8524 0.5028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9721 -0.3396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5249 -0.3120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4494 -1.0019 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3132 0.7688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5069 -0.2088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1328 0.4391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3308 -0.2088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7427 -0.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5666 -0.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.9791 -0.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.8041 -0.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.2166 -0.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.8041 -1.6367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.9791 -1.6367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-8.0405 -0.9223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-7.3823 0.3704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-8.1084 0.3704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.6715 -0.0575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.4855 0.0788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.5690 0.8999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.0592 1.5638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.2453 1.4275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.1617 0.6064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5579 0.6644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.1305 0.3119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.9550 -0.6793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.0857 -0.2508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.1084 0.5153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9227 -1.2094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5101 -1.9241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7117 -1.7149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9182 -1.9418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3308 -1.2270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1292 -1.4361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2862 -0.8502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7660 -0.5732 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4059 -1.6926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9587 -1.6650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8832 -2.3549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
3 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 4 1 0 0 0 0
5 11 2 0 0 0 0
1 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
15 18 1 0 0 0 0
10 19 1 0 0 0 0
8 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
26 27 1 0 0 0 0
27 31 1 0 0 0 0
21 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 9 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 40 1 0 0 0 0
40 41 2 0 0 0 0
41 36 1 0 0 0 0
39 42 1 0 0 0 0
38 43 1 0 0 0 0
43 44 1 0 0 0 0
45 46 1 1 0 0 0
46 47 1 1 0 0 0
48 47 1 1 0 0 0
48 49 1 0 0 0 0
49 50 1 0 0 0 0
50 45 1 0 0 0 0
50 51 1 0 0 0 0
51 52 1 0 0 0 0
45 53 1 0 0 0 0
46 54 1 0 0 0 0
47 55 1 0 0 0 0
48 18 1 0 0 0 0
56 57 1 1 0 0 0
57 58 1 1 0 0 0
59 58 1 1 0 0 0
59 60 1 0 0 0 0
60 61 1 0 0 0 0
61 56 1 0 0 0 0
61 62 1 0 0 0 0
62 63 1 0 0 0 0
56 64 1 0 0 0 0
57 65 1 0 0 0 0
58 66 1 0 0 0 0
59 30 1 0 0 0 0
32 63 1 0 0 0 0
S SKP 8
ID FL3FAADS0047
KNApSAcK_ID C00014083
NAME Isovitexin 2''-O-(6'''-(E)-feruloyl)glucoside 4'-O-glucoside
CAS_RN 372113-57-0
FORMULA C43H48O23
EXACTMASS 932.258637842
AVERAGEMASS 932.8274200000001
SMILES C(OC(C(c(c7O)c(c(C6=O)c(c7)OC(=C6)c(c5)ccc(c5)OC(O4)C(O)C(O)C(C4CO)O)O)3)C(C(O)C(O3)CO)O)(O1)C(O)C(O)C(O)C1COC(=O)C=Cc(c2)cc(c(O)c2)OC
M END