Mol:FL2F1AGS0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.6484 0.2926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6484 0.2926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9358 0.7083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9358 0.7083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6452 -0.5324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6452 -0.5324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9285 -0.9417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9285 -0.9417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2158 -0.5260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2158 -0.5260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2195 0.2989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2195 0.2989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5005 -0.9354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5005 -0.9354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2130 -0.5197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2130 -0.5197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2093 0.3053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2093 0.3053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4932 0.7146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4932 0.7146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8631 0.6861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8631 0.6861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5792 0.2764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5792 0.2764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2920 0.6916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2920 0.6916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2889 1.5166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2889 1.5166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5728 1.9264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5728 1.9264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8599 1.5111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8599 1.5111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5015 -1.5786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5015 -1.5786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2739 0.6537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2739 0.6537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9961 1.9250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9961 1.9250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0235 -1.0361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0235 -1.0361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6691 -0.5220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6691 -0.5220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3439 0.2365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3439 0.2365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4505 1.0548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4505 1.0548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8048 0.5408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8048 0.5408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1301 -0.2178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1301 -0.2178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6928 -0.5543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6928 -0.5543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6214 -1.1433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6214 -1.1433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2439 -1.7767 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2439 -1.7767 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6286 -1.3007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6286 -1.3007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8852 0.8304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8852 0.8304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8252 -0.5407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8252 -0.5407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8252 -1.1039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8252 -1.1039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6412 -1.1153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6412 -1.1153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8090 0.1982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8090 0.1982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2734 -0.1807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2734 -0.1807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1905 -0.1807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1905 -0.1807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6412 -0.5407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6412 -0.5407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2313 0.0779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2313 0.0779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3032 0.4835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3032 0.4835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3303 -1.4062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3303 -1.4062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8852 -1.0974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8852 -1.0974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3221 -1.9264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3221 -1.9264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 1 3 1 0 0 0 0 | + | 1 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 2 1 0 0 0 0 | + | 6 2 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 6 1 0 0 0 0 | + | 10 6 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 14 19 1 0 0 0 0 | + | 14 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 20 28 1 0 0 0 0 | + | 20 28 1 0 0 0 0 |
| − | 21 29 1 0 0 0 0 | + | 21 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 37 31 1 1 0 0 0 | + | 37 31 1 1 0 0 0 |
| − | 36 31 1 1 0 0 0 | + | 36 31 1 1 0 0 0 |
| − | 35 37 1 1 0 0 0 | + | 35 37 1 1 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 37 33 1 0 0 0 0 | + | 37 33 1 0 0 0 0 |
| − | 38 35 1 0 0 0 0 | + | 38 35 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 31 34 1 0 0 0 0 | + | 31 34 1 0 0 0 0 |
| − | 18 35 1 0 0 0 0 | + | 18 35 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 40 42 1 0 0 0 0 | + | 40 42 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
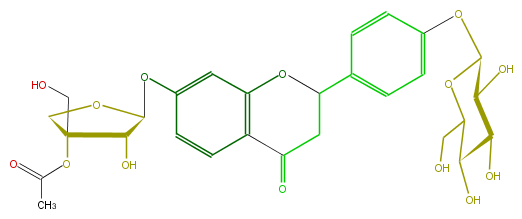
| − | ID FL2F1AGS0005 | + | ID FL2F1AGS0005 |
| − | FORMULA C28H32O14 | + | FORMULA C28H32O14 |
| − | EXACTMASS 592.179205732 | + | EXACTMASS 592.179205732 |
| − | AVERAGEMASS 592.54528 | + | AVERAGEMASS 592.54528 |
| − | SMILES OC(C(O)1)C(C(OC1Oc(c2)ccc(C(O5)CC(c(c45)ccc(c4)OC(O3)C(O)C(C3)(OC(C)=O)CO)=O)c2)CO)O | + | SMILES OC(C(O)1)C(C(OC1Oc(c2)ccc(C(O5)CC(c(c45)ccc(c4)OC(O3)C(O)C(C3)(OC(C)=O)CO)=O)c2)CO)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-1.6484 0.2926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9358 0.7083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6452 -0.5324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9285 -0.9417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2158 -0.5260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2195 0.2989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5005 -0.9354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2130 -0.5197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2093 0.3053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4932 0.7146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8631 0.6861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5792 0.2764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2920 0.6916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2889 1.5166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5728 1.9264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8599 1.5111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5015 -1.5786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2739 0.6537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9961 1.9250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0235 -1.0361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6691 -0.5220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3439 0.2365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4505 1.0548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8048 0.5408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1301 -0.2178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6928 -0.5543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6214 -1.1433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2439 -1.7767 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6286 -1.3007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8852 0.8304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8252 -0.5407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8252 -1.1039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6412 -1.1153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8090 0.1982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2734 -0.1807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1905 -0.1807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6412 -0.5407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2313 0.0779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3032 0.4835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3303 -1.4062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8852 -1.0974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3221 -1.9264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
1 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 2 1 0 0 0 0
5 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 6 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
1 18 1 0 0 0 0
14 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
20 28 1 0 0 0 0
21 29 1 0 0 0 0
22 30 1 0 0 0 0
23 19 1 0 0 0 0
37 31 1 1 0 0 0
36 31 1 1 0 0 0
35 37 1 1 0 0 0
31 32 1 0 0 0 0
37 33 1 0 0 0 0
38 35 1 0 0 0 0
34 39 1 0 0 0 0
36 38 1 0 0 0 0
31 34 1 0 0 0 0
18 35 1 0 0 0 0
32 40 1 0 0 0 0
40 41 2 0 0 0 0
40 42 1 0 0 0 0
S SKP 5
ID FL2F1AGS0005
FORMULA C28H32O14
EXACTMASS 592.179205732
AVERAGEMASS 592.54528
SMILES OC(C(O)1)C(C(OC1Oc(c2)ccc(C(O5)CC(c(c45)ccc(c4)OC(O3)C(O)C(C3)(OC(C)=O)CO)=O)c2)CO)O
M END