Mol:FL1DA9NC0012
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 60 66 0 0 0 0 0 0 0 0999 V2000 | + | 60 66 0 0 0 0 0 0 0 0999 V2000 |
| − | 5.3238 -2.6777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3238 -2.6777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9904 -2.2928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9904 -2.2928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9904 -1.5231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9904 -1.5231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3238 -1.1383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3238 -1.1383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6572 -1.5231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6572 -1.5231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6572 -2.2928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6572 -2.2928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9490 -2.7017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9490 -2.7017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1997 -2.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1997 -2.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4757 -2.6870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4757 -2.6870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7887 -2.2904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7887 -2.2904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7887 -1.5062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7887 -1.5062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1095 -1.1140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1095 -1.1140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4303 -1.5062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4303 -1.5062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4303 -2.2904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4303 -2.2904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1095 -2.6826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1095 -2.6826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4757 -3.2888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4757 -3.2888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1020 -1.1989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1020 -1.1989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1254 -2.6113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1254 -2.6113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1095 -3.2717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1095 -3.2717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8445 -2.1961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8445 -2.1961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8445 -1.4064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8445 -1.4064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5284 -1.0115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5284 -1.0115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2123 -1.4064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2123 -1.4064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2123 -2.1961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2123 -2.1961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5284 -2.5909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5284 -2.5909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5284 -3.1566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5284 -3.1566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1095 -0.3676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1095 -0.3676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3614 0.0643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3614 0.0643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3614 0.8048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3614 0.8048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2799 1.1750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2799 1.1750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9211 0.8048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9211 0.8048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9211 0.0643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9211 0.0643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2799 -0.3060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2799 -0.3060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1507 0.0778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1507 0.0778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1507 0.8211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1507 0.8211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7944 1.1927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7944 1.1927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4380 0.8211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4380 0.8211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4380 0.0778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4380 0.0778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7944 -0.2938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7944 -0.2938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8614 1.0934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8614 1.0934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0997 -0.3042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0997 -0.3042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6877 0.0353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6877 0.0353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6877 0.7873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6877 0.7873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3391 1.1634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3391 1.1634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9904 0.7873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9904 0.7873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9904 0.0353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9904 0.0353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3391 -0.3408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3391 -0.3408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3391 -0.9134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3391 -0.9134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3391 1.8598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3391 1.8598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7018 2.2278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7018 2.2278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0892 1.8741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0892 1.8741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4767 2.2278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4767 2.2278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4767 2.9351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4767 2.9351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0892 3.2888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0892 3.2888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7018 2.9351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7018 2.9351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1857 3.2145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1857 3.2145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5284 -0.3474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5284 -0.3474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7944 -0.6977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7944 -0.6977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3873 -1.1605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3873 -1.1605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1018 -1.5730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1018 -1.5730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 9 16 2 0 0 0 0 | + | 9 16 2 0 0 0 0 |
| − | 13 17 1 0 0 0 0 | + | 13 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 18 20 1 0 0 0 0 | + | 18 20 1 0 0 0 0 |
| − | 20 21 2 0 0 0 0 | + | 20 21 2 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 2 0 0 0 0 | + | 24 25 2 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 12 27 1 0 0 0 0 | + | 12 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 2 0 0 0 0 | + | 28 29 2 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 2 0 0 0 0 | + | 30 31 2 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 29 40 1 0 0 0 0 | + | 29 40 1 0 0 0 0 |
| − | 38 41 1 0 0 0 0 | + | 38 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 42 1 0 0 0 0 | + | 47 42 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 44 49 1 0 0 0 0 | + | 44 49 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 2 0 0 0 0 | + | 50 51 2 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 52 53 2 0 0 0 0 | + | 52 53 2 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 2 0 0 0 0 | + | 54 55 2 0 0 0 0 |
| − | 55 50 1 0 0 0 0 | + | 55 50 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 22 57 1 0 0 0 0 | + | 22 57 1 0 0 0 0 |
| − | 57 34 1 0 0 0 0 | + | 57 34 1 0 0 0 0 |
| − | 39 58 1 0 0 0 0 | + | 39 58 1 0 0 0 0 |
| − | 11 59 1 0 0 0 0 | + | 11 59 1 0 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 59 60 | + | M SAL 1 2 59 60 |
| − | M SBL 1 1 65 | + | M SBL 1 1 65 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 65 -7.4178 5.9839 | + | M SBV 1 65 -7.4178 5.9839 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1DA9NC0012 | + | ID FL1DA9NC0012 |
| − | KNApSAcK_ID C00008122 | + | KNApSAcK_ID C00008122 |
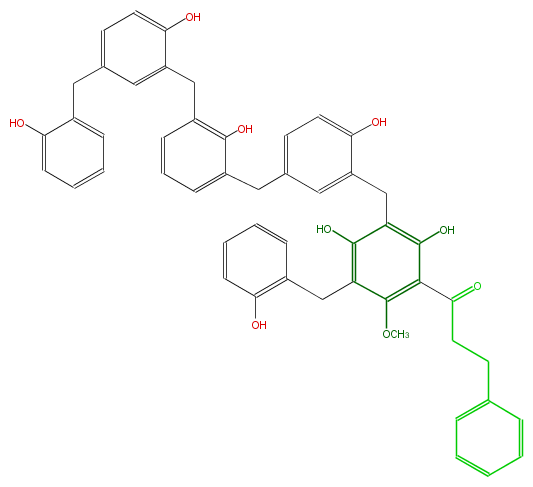
| − | NAME 2'''',2''''',2''''''-Trihydroxy-5''',3'''',5'''''-tribenzylisodiuvaretin | + | NAME 2'''',2''''',2''''''-Trihydroxy-5''',3'''',5'''''-tribenzylisodiuvaretin |
| − | CAS_RN 154879-10-4 | + | CAS_RN 154879-10-4 |
| − | FORMULA C51H46O9 | + | FORMULA C51H46O9 |
| − | EXACTMASS 802.31418307 | + | EXACTMASS 802.31418307 |
| − | AVERAGEMASS 802.90554 | + | AVERAGEMASS 802.90554 |
| − | SMILES c(c2Cc(c3)c(O)c(Cc(c4)ccc(O)c4Cc(c(O)6)c(c(c(OC)c(Cc(c7)c(ccc7)O)6)C(CCc(c5)cccc5)=O)O)cc3)c(ccc(O)2)Cc(c1O)cccc1 | + | SMILES c(c2Cc(c3)c(O)c(Cc(c4)ccc(O)c4Cc(c(O)6)c(c(c(OC)c(Cc(c7)c(ccc7)O)6)C(CCc(c5)cccc5)=O)O)cc3)c(ccc(O)2)Cc(c1O)cccc1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
60 66 0 0 0 0 0 0 0 0999 V2000
5.3238 -2.6777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9904 -2.2928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9904 -1.5231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3238 -1.1383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6572 -1.5231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6572 -2.2928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9490 -2.7017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1997 -2.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4757 -2.6870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7887 -2.2904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7887 -1.5062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1095 -1.1140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4303 -1.5062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4303 -2.2904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1095 -2.6826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4757 -3.2888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1020 -1.1989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1254 -2.6113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1095 -3.2717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8445 -2.1961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8445 -1.4064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5284 -1.0115 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2123 -1.4064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2123 -2.1961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5284 -2.5909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5284 -3.1566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1095 -0.3676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3614 0.0643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3614 0.8048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2799 1.1750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9211 0.8048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9211 0.0643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2799 -0.3060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1507 0.0778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1507 0.8211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7944 1.1927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4380 0.8211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4380 0.0778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7944 -0.2938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8614 1.0934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0997 -0.3042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6877 0.0353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6877 0.7873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3391 1.1634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.9904 0.7873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.9904 0.0353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3391 -0.3408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3391 -0.9134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3391 1.8598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7018 2.2278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0892 1.8741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4767 2.2278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4767 2.9351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0892 3.2888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7018 2.9351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1857 3.2145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5284 -0.3474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7944 -0.6977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3873 -1.1605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1018 -1.5730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
6 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
9 16 2 0 0 0 0
13 17 1 0 0 0 0
14 18 1 0 0 0 0
15 19 1 0 0 0 0
18 20 1 0 0 0 0
20 21 2 0 0 0 0
21 22 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
24 25 2 0 0 0 0
25 20 1 0 0 0 0
25 26 1 0 0 0 0
12 27 1 0 0 0 0
27 28 1 0 0 0 0
28 29 2 0 0 0 0
29 30 1 0 0 0 0
30 31 2 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
33 28 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 34 1 0 0 0 0
29 40 1 0 0 0 0
38 41 1 0 0 0 0
41 42 1 0 0 0 0
42 43 2 0 0 0 0
43 44 1 0 0 0 0
44 45 2 0 0 0 0
45 46 1 0 0 0 0
46 47 2 0 0 0 0
47 42 1 0 0 0 0
47 48 1 0 0 0 0
44 49 1 0 0 0 0
49 50 1 0 0 0 0
50 51 2 0 0 0 0
51 52 1 0 0 0 0
52 53 2 0 0 0 0
53 54 1 0 0 0 0
54 55 2 0 0 0 0
55 50 1 0 0 0 0
55 56 1 0 0 0 0
22 57 1 0 0 0 0
57 34 1 0 0 0 0
39 58 1 0 0 0 0
11 59 1 0 0 0 0
59 60 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 59 60
M SBL 1 1 65
M SMT 1 OCH3
M SBV 1 65 -7.4178 5.9839
S SKP 8
ID FL1DA9NC0012
KNApSAcK_ID C00008122
NAME 2'''',2''''',2''''''-Trihydroxy-5''',3'''',5'''''-tribenzylisodiuvaretin
CAS_RN 154879-10-4
FORMULA C51H46O9
EXACTMASS 802.31418307
AVERAGEMASS 802.90554
SMILES c(c2Cc(c3)c(O)c(Cc(c4)ccc(O)c4Cc(c(O)6)c(c(c(OC)c(Cc(c7)c(ccc7)O)6)C(CCc(c5)cccc5)=O)O)cc3)c(ccc(O)2)Cc(c1O)cccc1
M END