Mol:COX00102
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 39765 | + | 39765 |
| − | CDK 9/16/09,17:17 | + | CDK 9/16/09,17:17 |
| − | + | ||
| − | 97102 0 0 0 0 0 0 0 0999 V2000 | + | 97102 0 0 0 0 0 0 0 0999 V2000 |
| − | 10.3658 2.0580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.3658 2.0580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7158 -2.2495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7158 -2.2495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.7194 2.5961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.7194 2.5961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5760 -3.7529 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5760 -3.7529 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.6387 0.3028 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.6387 0.3028 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7319 -0.1579 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7319 -0.1579 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.1089 -0.1972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.1089 -0.1972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.1089 0.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.1089 0.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2429 -0.6972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.2429 -0.6972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3768 -0.1972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3768 -0.1972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4668 -0.7041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4668 -0.7041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.6387 0.3028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.6387 0.3028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.0551 1.1075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.0551 1.1075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.0551 -0.5019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.0551 -0.5019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4588 -1.7457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4588 -1.7457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2429 1.3028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.2429 1.3028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3768 0.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3768 0.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2589 -1.7387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.2589 -1.7387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3609 -2.2665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3609 -2.2665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5400 -0.1400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5400 -0.1400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.1089 1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.1089 1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5234 -2.2954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5234 -2.2954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5921 -0.6679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5921 -0.6679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4746 0.2959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4746 0.2959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5838 -1.7529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5838 -1.7529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.5047 -0.1972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.5047 -0.1972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.6387 1.3028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.6387 1.3028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.1387 -0.5632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.1387 -0.5632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.3708 0.3028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.3708 0.3028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.5047 1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.5047 1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.3708 1.3028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.3708 1.3028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7435 0.8420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7435 0.8420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8602 -0.6479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8602 -0.6479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.6979 2.8023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.6979 2.8023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8833 1.3520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8833 1.3520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -0.1379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -0.1379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0116 0.8620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0116 0.8620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7120 -3.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7120 -3.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.0086 3.7529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.0086 3.7529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8440 -3.7462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8440 -3.7462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.1936 -0.9927 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.1936 -0.9927 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9388 -1.0919 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9388 -1.0919 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0697 0.2028 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0697 0.2028 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.9204 -0.2495 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.9204 -0.2495 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.6676 1.2037 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.6676 1.2037 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.8041 -1.0688 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.8041 -1.0688 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.5925 -0.8112 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.5925 -0.8112 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4619 -2.5456 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4619 -2.5456 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8443 1.7777 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8443 1.7777 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.6414 1.7777 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.6414 1.7777 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7663 0.6951 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7663 0.6951 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1648 1.3854 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1648 1.3854 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.4770 -2.3191 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4770 -2.3191 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8681 -1.6232 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8681 -1.6232 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.7619 -2.7394 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.7619 -2.7394 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9636 -2.7424 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9636 -2.7424 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9482 0.3267 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9482 0.3267 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1501 0.3421 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1501 0.3421 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.4889 1.8028 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4889 1.8028 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.1089 2.4228 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.1089 2.4228 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.7289 1.8028 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.7289 1.8028 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9244 -2.7683 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9244 -2.7683 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1261 -2.7713 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1261 -2.7713 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0522 -0.9727 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0522 -0.9727 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8546 0.3007 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8546 0.3007 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4793 0.9159 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4793 0.9159 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0945 0.2911 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0945 0.2911 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5826 -2.3729 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5826 -2.3729 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.9033 -0.6722 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.9033 -0.6722 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.1062 -0.6722 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.1062 -0.6722 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.0281 1.1951 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.0281 1.1951 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.4267 1.8854 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.4267 1.8854 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.6757 -0.8732 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.6757 -0.8732 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.8287 -1.1002 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.8287 -1.1002 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.6018 -0.2532 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.6018 -0.2532 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.5828 -0.2798 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.5828 -0.2798 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.9814 0.4105 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.9814 0.4105 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.1062 2.2777 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.1062 2.2777 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.9033 2.2777 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.9033 2.2777 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.5828 1.8854 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.5828 1.8854 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.9814 1.1951 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.9814 1.1951 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9623 1.4221 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9623 1.4221 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3528 0.7273 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3528 0.7273 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2532 -1.1274 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2532 -1.1274 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4562 -1.1182 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4562 -1.1182 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2873 1.8223 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2873 1.8223 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4903 1.8315 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4903 1.8315 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3907 -0.0232 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3907 -0.0232 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7812 -0.7180 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7812 -0.7180 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8063 1.4470 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8063 1.4470 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3998 0.7614 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3998 0.7614 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.4193 3.9455 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.4193 3.9455 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.2012 4.3422 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.2012 4.3422 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.5979 3.5603 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.5979 3.5603 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1519 -4.2843 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1519 -4.2843 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3059 -4.0541 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3059 -4.0541 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5361 -3.2081 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5361 -3.2081 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
13 1 1 1 0 0 0 | 13 1 1 1 0 0 0 | ||
1 34 1 0 0 0 0 | 1 34 1 0 0 0 0 | ||
| Line 202: | Line 202: | ||
40 96 1 0 0 0 0 | 40 96 1 0 0 0 0 | ||
40 97 1 0 0 0 0 | 40 97 1 0 0 0 0 | ||
| − | M CHG 1 5 1 | + | M CHG 1 5 1 |
| − | S SKP | + | S SKP 6 |
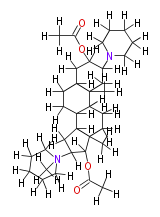
| − | NAME Vecuronium | + | NAME Vecuronium |
| + | ID COX00102 | ||
| + | FORMULA C34H57N2O4 | ||
| + | EXACTMASS 557.431833322 | ||
| + | AVERAGEMASS 557.82746 | ||
| + | SMILES C(C6([H])[H])(C(C([H])([H])N(C6([H])[H])(C(C([H])([H])1)([H])C([H])(C(C([H])([H])[H])(C([H])([H])5)C1([H])C([H])(C4([H])[H])C(C([H])([H])5)([H])C(C(C([H])([H])4)3[H])(C([H])([H])[H])C([H])([H])C(C(OC(=O)C([H])([H])[H])([H])C3([H])[H])([H])N(C([H])([H])2)C([H])([H])C(C([H])([H])C2([H])[H])([H])[H])OC(=O)C([H])([H])[H])C([H])([H])[H])([H])[H])([H])[H] | ||
M END | M END | ||
Latest revision as of 20:48, 21 February 2011
39765
CDK 9/16/09,17:17
97102 0 0 0 0 0 0 0 0999 V2000
10.3658 2.0580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7158 -2.2495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.7194 2.5961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5760 -3.7529 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.6387 0.3028 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
3.7319 -0.1579 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
9.1089 -0.1972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.1089 0.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.2429 -0.6972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3768 -0.1972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.4668 -0.7041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.6387 0.3028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.0551 1.1075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.0551 -0.5019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.4588 -1.7457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.2429 1.3028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3768 0.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.2589 -1.7387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3609 -2.2665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5400 -0.1400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.1089 1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5234 -2.2954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5921 -0.6679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.4746 0.2959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5838 -1.7529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.5047 -0.1972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.6387 1.3028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.1387 -0.5632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.3708 0.3028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.5047 1.8028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.3708 1.3028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7435 0.8420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8602 -0.6479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.6979 2.8023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8833 1.3520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -0.1379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0116 0.8620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7120 -3.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.0086 3.7529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8440 -3.7462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.1936 -0.9927 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.9388 -1.0919 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.0697 0.2028 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.9204 -0.2495 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.6676 1.2037 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.8041 -1.0688 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.5925 -0.8112 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.4619 -2.5456 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.8443 1.7777 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.6414 1.7777 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.7663 0.6951 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.1648 1.3854 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.4770 -2.3191 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.8681 -1.6232 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.7619 -2.7394 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.9636 -2.7424 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.9482 0.3267 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.1501 0.3421 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.4889 1.8028 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.1089 2.4228 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.7289 1.8028 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.9244 -2.7683 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.1261 -2.7713 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.0522 -0.9727 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.8546 0.3007 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.4793 0.9159 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.0945 0.2911 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.5826 -2.3729 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
12.9033 -0.6722 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
12.1062 -0.6722 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
11.0281 1.1951 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
11.4267 1.8854 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
11.6757 -0.8732 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.8287 -1.1002 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.6018 -0.2532 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
13.5828 -0.2798 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
13.9814 0.4105 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
12.1062 2.2777 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
12.9033 2.2777 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
13.5828 1.8854 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
13.9814 1.1951 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.9623 1.4221 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.3528 0.7273 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.2532 -1.1274 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.4562 -1.1182 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.2873 1.8223 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.4903 1.8315 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.3907 -0.0232 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.7812 -0.7180 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.8063 1.4470 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.3998 0.7614 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.4193 3.9455 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.2012 4.3422 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.5979 3.5603 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.1519 -4.2843 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.3059 -4.0541 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.5361 -3.2081 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
13 1 1 1 0 0 0
1 34 1 0 0 0 0
25 2 1 6 0 0 0
2 38 1 0 0 0 0
3 34 2 0 0 0 0
4 38 2 0 0 0 0
12 5 1 1 0 0 0
5 26 1 0 0 0 0
5 27 1 0 0 0 0
5 28 1 0 0 0 0
23 6 1 1 0 0 0
6 32 1 0 0 0 0
6 33 1 0 0 0 0
7 8 1 0 0 0 0
7 9 1 0 0 0 0
7 14 1 0 0 0 0
7 41 1 6 0 0 0
8 13 1 0 0 0 0
8 16 1 0 0 0 0
8 21 1 1 0 0 0
9 10 1 0 0 0 0
9 18 1 0 0 0 0
9 42 1 1 0 0 0
10 11 1 0 0 0 0
10 17 1 0 0 0 0
10 43 1 6 0 0 0
11 15 1 0 0 0 0
11 20 1 0 0 0 0
11 24 1 1 0 0 0
12 13 1 0 0 0 0
12 14 1 0 0 0 0
12 44 1 0 0 0 0
13 45 1 0 0 0 0
14 46 1 0 0 0 0
14 47 1 0 0 0 0
15 19 1 0 0 0 0
15 22 1 0 0 0 0
15 48 1 6 0 0 0
16 17 1 0 0 0 0
16 49 1 0 0 0 0
16 50 1 0 0 0 0
17 51 1 0 0 0 0
17 52 1 0 0 0 0
18 19 1 0 0 0 0
18 53 1 0 0 0 0
18 54 1 0 0 0 0
19 55 1 0 0 0 0
19 56 1 0 0 0 0
20 23 1 0 0 0 0
20 57 1 0 0 0 0
20 58 1 0 0 0 0
21 59 1 0 0 0 0
21 60 1 0 0 0 0
21 61 1 0 0 0 0
22 25 1 0 0 0 0
22 62 1 0 0 0 0
22 63 1 0 0 0 0
23 25 1 0 0 0 0
23 64 1 0 0 0 0
24 65 1 0 0 0 0
24 66 1 0 0 0 0
24 67 1 0 0 0 0
25 68 1 0 0 0 0
26 29 1 0 0 0 0
26 69 1 0 0 0 0
26 70 1 0 0 0 0
27 30 1 0 0 0 0
27 71 1 0 0 0 0
27 72 1 0 0 0 0
28 73 1 0 0 0 0
28 74 1 0 0 0 0
28 75 1 0 0 0 0
29 31 1 0 0 0 0
29 76 1 0 0 0 0
29 77 1 0 0 0 0
30 31 1 0 0 0 0
30 78 1 0 0 0 0
30 79 1 0 0 0 0
31 80 1 0 0 0 0
31 81 1 0 0 0 0
32 35 1 0 0 0 0
32 82 1 0 0 0 0
32 83 1 0 0 0 0
33 36 1 0 0 0 0
33 84 1 0 0 0 0
33 85 1 0 0 0 0
34 39 1 0 0 0 0
35 37 1 0 0 0 0
35 86 1 0 0 0 0
35 87 1 0 0 0 0
36 37 1 0 0 0 0
36 88 1 0 0 0 0
36 89 1 0 0 0 0
37 90 1 0 0 0 0
37 91 1 0 0 0 0
38 40 1 0 0 0 0
39 92 1 0 0 0 0
39 93 1 0 0 0 0
39 94 1 0 0 0 0
40 95 1 0 0 0 0
40 96 1 0 0 0 0
40 97 1 0 0 0 0
M CHG 1 5 1
S SKP 6
NAME Vecuronium
ID COX00102
FORMULA C34H57N2O4
EXACTMASS 557.431833322
AVERAGEMASS 557.82746
SMILES C(C6([H])[H])(C(C([H])([H])N(C6([H])[H])(C(C([H])([H])1)([H])C([H])(C(C([H])([H])[H])(C([H])([H])5)C1([H])C([H])(C4([H])[H])C(C([H])([H])5)([H])C(C(C([H])([H])4)3[H])(C([H])([H])[H])C([H])([H])C(C(OC(=O)C([H])([H])[H])([H])C3([H])[H])([H])N(C([H])([H])2)C([H])([H])C(C([H])([H])C2([H])[H])([H])[H])OC(=O)C([H])([H])[H])C([H])([H])[H])([H])[H])([H])[H]
M END