Mol:COX00069
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 19675 | + | 19675 |
| − | CDK 9/16/09,17:9 | + | CDK 9/16/09,17:9 |
| − | + | ||
| − | 59 62 0 0 0 0 0 0 0 0999 V2000 | + | 59 62 0 0 0 0 0 0 0 0999 V2000 |
| − | 7.1962 -4.2927 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1962 -4.2927 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 3.7073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 3.7073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.7224 -1.2685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.7224 -1.2685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4641 0.7073 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4641 0.7073 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1962 -2.2927 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1962 -2.2927 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7321 1.7073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 1.7073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7321 0.7073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 0.7073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5981 2.2073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5981 2.2073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5981 0.2073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5981 0.2073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4641 1.7073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4641 1.7073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 2.2073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 2.2073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3301 0.2073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3301 0.2073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3301 -0.7927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3301 -0.7927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 3.2073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 3.2073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1962 -1.2927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1962 -1.2927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0622 -2.7927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0622 -2.7927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3301 -2.7927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3301 -2.7927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0622 -3.7927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0622 -3.7927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3301 -3.7927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3301 -3.7927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9561 -2.2580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9561 -2.2580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4362 -2.2580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4362 -2.2580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9561 -4.3273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9561 -4.3273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.8622 -2.7719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.8622 -2.7719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4362 -4.3273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4362 -4.3273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.8622 -3.8135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.8622 -3.8135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5301 -2.7719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5301 -2.7719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5301 -3.8135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5301 -3.8135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.7263 -2.2685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.7263 -2.2685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.5942 -2.7652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.5942 -2.7652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1951 1.3973 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1951 1.3973 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1215 0.8150 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1215 0.8150 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5200 0.1247 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5200 0.1247 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1995 2.6823 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1995 2.6823 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9966 2.6823 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9966 2.6823 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1995 -0.2676 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1995 -0.2676 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9966 -0.2676 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9966 -0.2676 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0747 1.5997 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0747 1.5997 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6762 2.2899 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6762 2.2899 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6540 1.6247 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6540 1.6247 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2554 2.3150 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2554 2.3150 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5422 0.7899 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5422 0.7899 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9407 0.0997 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9407 0.0997 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1181 -1.3753 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1181 -1.3753 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7195 -0.6850 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7195 -0.6850 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0781 3.7899 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0781 3.7899 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4766 3.0997 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4766 3.0997 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4082 -0.7101 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4082 -0.7101 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8067 -1.4003 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8067 -1.4003 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 4.3273 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 4.3273 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9490 -1.6380 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9490 -1.6380 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4434 -1.6380 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4434 -1.6380 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9490 -4.9473 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9490 -4.9473 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4434 -4.9473 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4434 -4.9473 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.3979 -4.1256 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.3979 -4.1256 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9944 -2.4598 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9944 -2.4598 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9944 -4.1256 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9944 -4.1256 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.2863 -3.3033 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.2863 -3.3033 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.1324 -3.0731 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.1324 -3.0731 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.9022 -2.2271 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.9022 -2.2271 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
1 18 1 0 0 0 0 | 1 18 1 0 0 0 0 | ||
1 19 1 0 0 0 0 | 1 19 1 0 0 0 0 | ||
| Line 124: | Line 124: | ||
29 58 1 0 0 0 0 | 29 58 1 0 0 0 0 | ||
29 59 1 0 0 0 0 | 29 59 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
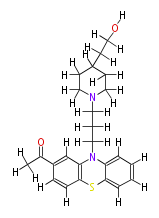
| − | NAME Piperacetazine | + | NAME Piperacetazine |
| + | ID COX00069 | ||
| + | FORMULA C24H30N2O2S | ||
| + | EXACTMASS 410.202798904 | ||
| + | AVERAGEMASS 410.57328000000007 | ||
| + | SMILES N(c43)(c(c([H])2)c(Sc(c([H])c([H])c([H])c([H])4)3)c([H])c([H])c(C(=O)C([H])([H])[H])2)C([H])([H])C([H])([H])C(N(C1([H])[H])C(C(C(C([H])([H])1)([H])C([H])([H])C([H])([H])O[H])([H])[H])([H])[H])([H])[H] | ||
M END | M END | ||
Latest revision as of 20:47, 21 February 2011
19675
CDK 9/16/09,17:9
59 62 0 0 0 0 0 0 0 0999 V2000
7.1962 -4.2927 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 3.7073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
10.7224 -1.2685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.4641 0.7073 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
7.1962 -2.2927 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
3.7321 1.7073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7321 0.7073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 2.2073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 0.2073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4641 1.7073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 2.2073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3301 0.2073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3301 -0.7927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 3.2073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.1962 -1.2927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.0622 -2.7927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3301 -2.7927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.0622 -3.7927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3301 -3.7927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.9561 -2.2580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4362 -2.2580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.9561 -4.3273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.8622 -2.7719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4362 -4.3273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.8622 -3.8135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5301 -2.7719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5301 -3.8135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.7263 -2.2685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.5942 -2.7652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1951 1.3973 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.1215 0.8150 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.5200 0.1247 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.1995 2.6823 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.9966 2.6823 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.1995 -0.2676 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.9966 -0.2676 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.0747 1.5997 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.6762 2.2899 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.6540 1.6247 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.2554 2.3150 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.5422 0.7899 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.9407 0.0997 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.1181 -1.3753 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.7195 -0.6850 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.0781 3.7899 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.4766 3.0997 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.4082 -0.7101 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.8067 -1.4003 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 4.3273 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.9490 -1.6380 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.4434 -1.6380 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.9490 -4.9473 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.4434 -4.9473 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.3979 -4.1256 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.9944 -2.4598 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.9944 -4.1256 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
11.2863 -3.3033 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
12.1324 -3.0731 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
11.9022 -2.2271 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1 18 1 0 0 0 0
1 19 1 0 0 0 0
2 14 1 0 0 0 0
2 49 1 0 0 0 0
3 28 2 0 0 0 0
4 9 1 0 0 0 0
4 10 1 0 0 0 0
4 12 1 0 0 0 0
5 15 1 0 0 0 0
5 16 1 0 0 0 0
5 17 1 0 0 0 0
6 7 1 0 0 0 0
6 8 1 0 0 0 0
6 11 1 0 0 0 0
6 30 1 0 0 0 0
7 9 1 0 0 0 0
7 31 1 0 0 0 0
7 32 1 0 0 0 0
8 10 1 0 0 0 0
8 33 1 0 0 0 0
8 34 1 0 0 0 0
9 35 1 0 0 0 0
9 36 1 0 0 0 0
10 37 1 0 0 0 0
10 38 1 0 0 0 0
11 14 1 0 0 0 0
11 39 1 0 0 0 0
11 40 1 0 0 0 0
12 13 1 0 0 0 0
12 41 1 0 0 0 0
12 42 1 0 0 0 0
13 15 1 0 0 0 0
13 43 1 0 0 0 0
13 44 1 0 0 0 0
14 45 1 0 0 0 0
14 46 1 0 0 0 0
15 47 1 0 0 0 0
15 48 1 0 0 0 0
16 18 2 0 0 0 0
16 20 1 0 0 0 0
17 19 1 0 0 0 0
17 21 2 0 0 0 0
18 22 1 0 0 0 0
19 24 2 0 0 0 0
20 23 2 0 0 0 0
20 50 1 0 0 0 0
21 26 1 0 0 0 0
21 51 1 0 0 0 0
22 25 2 0 0 0 0
22 52 1 0 0 0 0
23 25 1 0 0 0 0
23 28 1 0 0 0 0
24 27 1 0 0 0 0
24 53 1 0 0 0 0
25 54 1 0 0 0 0
26 27 2 0 0 0 0
26 55 1 0 0 0 0
27 56 1 0 0 0 0
28 29 1 0 0 0 0
29 57 1 0 0 0 0
29 58 1 0 0 0 0
29 59 1 0 0 0 0
S SKP 6
NAME Piperacetazine
ID COX00069
FORMULA C24H30N2O2S
EXACTMASS 410.202798904
AVERAGEMASS 410.57328000000007
SMILES N(c43)(c(c([H])2)c(Sc(c([H])c([H])c([H])c([H])4)3)c([H])c([H])c(C(=O)C([H])([H])[H])2)C([H])([H])C([H])([H])C(N(C1([H])[H])C(C(C(C([H])([H])1)([H])C([H])([H])C([H])([H])O[H])([H])[H])([H])[H])([H])[H]
M END