Mol:COX00066
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 4635 | + | 4635 |
| − | CDK 9/16/09,17:9 | + | CDK 9/16/09,17:9 |
| − | + | ||
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.2314 -0.4472 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2314 -0.4472 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3794 -1.9511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3794 -1.9511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -2.9021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -2.9021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 1.9022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 1.9022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1607 -1.4511 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1607 -1.4511 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6897 -0.9755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6897 -0.9755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5133 -1.4511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5133 -1.4511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3370 -0.9755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3370 -0.9755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 -1.4511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 -1.4511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2392 -0.2781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2392 -0.2781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6897 -0.0245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6897 -0.0245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5133 -2.4021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5133 -2.4021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3370 -0.0245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3370 -0.0245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5918 -0.2781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5918 -0.2781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5133 0.4511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5133 0.4511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6897 -2.8777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6897 -2.8777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 -2.4021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 -2.4021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 0.4511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 0.4511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8418 -2.1832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8418 -2.1832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5133 1.4022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5133 1.4022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 1.4022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 1.4022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6897 1.8777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6897 1.8777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 2.9021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 2.9021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8739 -0.6655 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8739 -0.6655 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2705 -1.6235 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2705 -1.6235 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3760 0.3266 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3760 0.3266 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6795 -0.0113 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6795 -0.0113 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7254 -2.9848 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7254 -2.9848 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1239 -2.2945 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1239 -2.2945 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9476 -0.1321 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9476 -0.1321 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5491 0.5581 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5491 0.5581 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1840 -0.0943 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1840 -0.0943 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4989 0.3349 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4989 0.3349 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2912 -3.3526 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2912 -3.3526 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0882 -3.3526 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0882 -3.3526 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3794 -2.5711 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3794 -2.5711 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3879 -2.6055 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3879 -2.6055 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2641 -2.6371 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2641 -2.6371 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2957 -1.7609 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2957 -1.7609 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0503 1.7122 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0503 1.7122 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6897 2.4977 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6897 2.4977 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6200 2.9021 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6200 2.9021 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 3.5222 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 3.5222 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3800 2.9021 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3800 2.9021 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
1 9 1 0 0 0 0 | 1 9 1 0 0 0 0 | ||
1 18 1 0 0 0 0 | 1 18 1 0 0 0 0 | ||
| Line 95: | Line 95: | ||
23 43 1 0 0 0 0 | 23 43 1 0 0 0 0 | ||
23 44 1 0 0 0 0 | 23 44 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
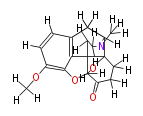
| − | NAME Oxycodone | + | NAME Oxycodone |
| + | ID COX00066 | ||
| + | FORMULA C18H21NO4 | ||
| + | EXACTMASS 315.147058165 | ||
| + | AVERAGEMASS 315.36368 | ||
| + | SMILES [H]C([H])([H])Oc(c([H])5)c(O4)c(c(c([H])5)1)C(C([H])([H])2)(C([H])43)C(O[H])(C([H])([H])C([H])([H])C(=O)3)C([H])(N(C([H])([H])[H])C([H])([H])2)C([H])([H])1 | ||
M END | M END | ||
Latest revision as of 20:47, 21 February 2011
4635
CDK 9/16/09,17:9
44 48 0 0 0 0 0 0 0 0999 V2000
2.2314 -0.4472 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3794 -1.9511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -2.9021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 1.9022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.1607 -1.4511 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
3.6897 -0.9755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5133 -1.4511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3370 -0.9755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 -1.4511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2392 -0.2781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6897 -0.0245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5133 -2.4021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3370 -0.0245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5918 -0.2781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5133 0.4511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6897 -2.8777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 -2.4021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 0.4511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.8418 -2.1832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5133 1.4022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 1.4022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6897 1.8777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 2.9021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.8739 -0.6655 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.2705 -1.6235 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.3760 0.3266 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.6795 -0.0113 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.7254 -2.9848 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.1239 -2.2945 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.9476 -0.1321 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.5491 0.5581 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.1840 -0.0943 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.4989 0.3349 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.2912 -3.3526 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.0882 -3.3526 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.3794 -2.5711 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.3879 -2.6055 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.2641 -2.6371 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.2957 -1.7609 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.0503 1.7122 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.6897 2.4977 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.6200 2.9021 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 3.5222 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.3800 2.9021 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1 9 1 0 0 0 0
1 18 1 0 0 0 0
2 7 1 0 0 0 0
2 36 1 0 0 0 0
3 17 2 0 0 0 0
4 21 1 0 0 0 0
4 23 1 0 0 0 0
5 8 1 0 0 0 0
5 14 1 0 0 0 0
5 19 1 0 0 0 0
6 7 1 0 0 0 0
6 9 1 0 0 0 0
6 10 1 0 0 0 0
6 11 1 0 0 0 0
7 8 1 0 0 0 0
7 12 1 0 0 0 0
8 13 1 0 0 0 0
8 24 1 0 0 0 0
9 17 1 0 0 0 0
9 25 1 0 0 0 0
10 14 1 0 0 0 0
10 26 1 0 0 0 0
10 27 1 0 0 0 0
11 15 2 0 0 0 0
11 18 1 0 0 0 0
12 16 1 0 0 0 0
12 28 1 0 0 0 0
12 29 1 0 0 0 0
13 15 1 0 0 0 0
13 30 1 0 0 0 0
13 31 1 0 0 0 0
14 32 1 0 0 0 0
14 33 1 0 0 0 0
15 20 1 0 0 0 0
16 17 1 0 0 0 0
16 34 1 0 0 0 0
16 35 1 0 0 0 0
18 21 2 0 0 0 0
19 37 1 0 0 0 0
19 38 1 0 0 0 0
19 39 1 0 0 0 0
20 22 2 0 0 0 0
20 40 1 0 0 0 0
21 22 1 0 0 0 0
22 41 1 0 0 0 0
23 42 1 0 0 0 0
23 43 1 0 0 0 0
23 44 1 0 0 0 0
S SKP 6
NAME Oxycodone
ID COX00066
FORMULA C18H21NO4
EXACTMASS 315.147058165
AVERAGEMASS 315.36368
SMILES [H]C([H])([H])Oc(c([H])5)c(O4)c(c(c([H])5)1)C(C([H])([H])2)(C([H])43)C(O[H])(C([H])([H])C([H])([H])C(=O)3)C([H])(N(C([H])([H])[H])C([H])([H])2)C([H])([H])1
M END