Mol:COX00058
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 4428 | + | 4428 |
| − | CDK 9/16/09,17:7 | + | CDK 9/16/09,17:7 |
| − | + | ||
| − | 48 53 0 0 0 0 0 0 0 0999 V2000 | + | 48 53 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.2314 -0.2572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2314 -0.2572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3794 -1.7611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3794 -1.7611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -2.7122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -2.7122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 2.0922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 2.0922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1607 -1.2611 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1607 -1.2611 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6897 -0.7855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6897 -0.7855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5133 -1.2611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5133 -1.2611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3370 -0.7855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3370 -0.7855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 -1.2611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 -1.2611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2392 -0.0881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2392 -0.0881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6897 0.1655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6897 0.1655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5133 -2.2122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5133 -2.2122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5918 -0.0881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5918 -0.0881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3370 0.1655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3370 0.1655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5133 0.6411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5133 0.6411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8418 -1.9932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8418 -1.9932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6897 -2.6877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6897 -2.6877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8164 -1.7694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8164 -1.7694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 -2.2122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 -2.2122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 0.6411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 0.6411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.7724 -2.0628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.7724 -2.0628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.5486 -1.0882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.5486 -1.0882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5133 1.5922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5133 1.5922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 1.5922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 1.5922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6897 2.0677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6897 2.0677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0731 -0.3605 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0731 -0.3605 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0496 -1.4975 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0496 -1.4975 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3760 0.5166 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3760 0.5166 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6795 0.1787 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6795 0.1787 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7254 -2.7948 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7254 -2.7948 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1239 -2.1045 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1239 -2.1045 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1840 0.0957 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1840 0.0957 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4989 0.5249 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4989 0.5249 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9476 0.0579 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9476 0.0579 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5491 0.7481 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5491 0.7481 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3215 -2.3303 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3215 -2.3303 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0834 -2.5642 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0834 -2.5642 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5260 -1.2216 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5260 -1.2216 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2912 -3.1626 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2912 -3.1626 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0882 -3.1626 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0882 -3.1626 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.6962 -2.6781 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.6962 -2.6781 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.3916 -2.0311 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.3916 -2.0311 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.1196 -0.8466 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.1196 -0.8466 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2115 -0.5678 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.2115 -0.5678 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3794 -2.3811 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3794 -2.3811 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0503 1.9022 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0503 1.9022 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6897 2.6877 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6897 2.6877 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 2.7122 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 2.7122 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
1 9 1 0 0 0 0 | 1 9 1 0 0 0 0 | ||
1 20 1 0 0 0 0 | 1 20 1 0 0 0 0 | ||
| Line 104: | Line 104: | ||
24 25 1 0 0 0 0 | 24 25 1 0 0 0 0 | ||
25 47 1 0 0 0 0 | 25 47 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
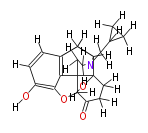
| − | NAME Naltrexone | + | NAME Naltrexone |
| + | ID COX00058 | ||
| + | FORMULA C20H23NO4 | ||
| + | EXACTMASS 341.162708229 | ||
| + | AVERAGEMASS 341.40095999999994 | ||
| + | SMILES [H]Oc(c([H])6)c(O5)c(c(c([H])6)3)C(C([H])([H])1)(C([H])54)C(O[H])(C([H])([H])C([H])([H])C(=O)4)C([H])(C([H])([H])3)N(C([H])([H])C([H])(C([H])([H])2)C([H])([H])2)C([H])([H])1 | ||
M END | M END | ||
Latest revision as of 20:47, 21 February 2011
4428
CDK 9/16/09,17:7
48 53 0 0 0 0 0 0 0 0999 V2000
2.2314 -0.2572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3794 -1.7611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -2.7122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 2.0922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.1607 -1.2611 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
3.6897 -0.7855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5133 -1.2611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3370 -0.7855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 -1.2611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2392 -0.0881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6897 0.1655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5133 -2.2122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5918 -0.0881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3370 0.1655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5133 0.6411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.8418 -1.9932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6897 -2.6877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.8164 -1.7694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 -2.2122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 0.6411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.7724 -2.0628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.5486 -1.0882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5133 1.5922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 1.5922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6897 2.0677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0731 -0.3605 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.0496 -1.4975 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.3760 0.5166 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.6795 0.1787 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.7254 -2.7948 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.1239 -2.1045 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.1840 0.0957 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.4989 0.5249 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.9476 0.0579 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.5491 0.7481 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.3215 -2.3303 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.0834 -2.5642 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.5260 -1.2216 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.2912 -3.1626 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.0882 -3.1626 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.6962 -2.6781 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.3916 -2.0311 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.1196 -0.8466 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.2115 -0.5678 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.3794 -2.3811 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.0503 1.9022 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.6897 2.6877 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 2.7122 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1 9 1 0 0 0 0
1 20 1 0 0 0 0
2 7 1 0 0 0 0
2 45 1 0 0 0 0
3 19 2 0 0 0 0
4 24 1 0 0 0 0
4 48 1 0 0 0 0
5 8 1 0 0 0 0
5 13 1 0 0 0 0
5 16 1 0 0 0 0
6 7 1 0 0 0 0
6 9 1 0 0 0 0
6 10 1 0 0 0 0
6 11 1 0 0 0 0
7 8 1 0 0 0 0
7 12 1 0 0 0 0
8 14 1 0 0 0 0
8 26 1 0 0 0 0
9 19 1 0 0 0 0
9 27 1 0 0 0 0
10 13 1 0 0 0 0
10 28 1 0 0 0 0
10 29 1 0 0 0 0
11 15 2 0 0 0 0
11 20 1 0 0 0 0
12 17 1 0 0 0 0
12 30 1 0 0 0 0
12 31 1 0 0 0 0
13 32 1 0 0 0 0
13 33 1 0 0 0 0
14 15 1 0 0 0 0
14 34 1 0 0 0 0
14 35 1 0 0 0 0
15 23 1 0 0 0 0
16 18 1 0 0 0 0
16 36 1 0 0 0 0
16 37 1 0 0 0 0
17 19 1 0 0 0 0
17 39 1 0 0 0 0
17 40 1 0 0 0 0
18 21 1 0 0 0 0
18 22 1 0 0 0 0
18 38 1 0 0 0 0
20 24 2 0 0 0 0
21 22 1 0 0 0 0
21 41 1 0 0 0 0
21 42 1 0 0 0 0
22 43 1 0 0 0 0
22 44 1 0 0 0 0
23 25 2 0 0 0 0
23 46 1 0 0 0 0
24 25 1 0 0 0 0
25 47 1 0 0 0 0
S SKP 6
NAME Naltrexone
ID COX00058
FORMULA C20H23NO4
EXACTMASS 341.162708229
AVERAGEMASS 341.40095999999994
SMILES [H]Oc(c([H])6)c(O5)c(c(c([H])6)3)C(C([H])([H])1)(C([H])54)C(O[H])(C([H])([H])C([H])([H])C(=O)4)C([H])(C([H])([H])3)N(C([H])([H])C([H])(C([H])([H])2)C([H])([H])2)C([H])([H])1
M END