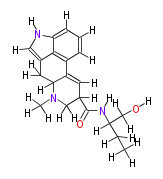
Mol:COX00056
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 4140 | + | 4140 |
| − | CDK 9/16/09,17:7 | + | CDK 9/16/09,17:7 |
| − | + | ||
| − | 50 53 0 0 0 0 0 0 0 0999 V2000 | + | 50 53 0 0 0 0 0 0 0 0999 V2000 |
| − | 3.7398 0.3119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7398 0.3119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8602 3.8086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8602 3.8086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2719 0.3117 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2719 0.3117 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2599 -3.8785 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.2599 -3.8785 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6000 1.8153 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6000 1.8153 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2558 -0.7298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2558 -0.7298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3898 -1.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3898 -1.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1219 -1.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1219 -1.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4718 0.3186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4718 0.3186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3739 0.8394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3739 0.8394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1219 -2.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1219 -2.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3898 -2.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3898 -2.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2558 -2.7298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2558 -2.7298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4798 -0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4798 -0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2719 -3.7713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2719 -3.7713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1436 0.8016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1436 0.8016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6038 0.8153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6038 0.8153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.7398 -3.0082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.7398 -3.0082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4798 -2.7367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4798 -2.7367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3739 -4.2991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3739 -4.2991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4718 -3.7783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4718 -3.7783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7320 2.3119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7320 2.3119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8680 1.8086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8680 1.8086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7282 3.3119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7282 3.3119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 2.3053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 2.3053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.7952 -0.4240 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.7952 -0.4240 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.7325 -1.3375 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.7325 -1.3375 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.3339 -0.6472 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.3339 -0.6472 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4706 0.9386 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4706 0.9386 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7748 1.3123 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7748 1.3123 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9766 1.3154 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9766 1.3154 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9465 -1.0392 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9465 -1.0392 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.4474 0.2611 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4474 0.2611 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.6841 1.1054 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.6841 1.1054 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8399 1.3421 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8399 1.3421 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.3576 -2.9564 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.3576 -2.9564 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9465 -2.4205 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9465 -2.4205 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.5459 -4.4286 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.5459 -4.4286 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3763 -4.9191 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3763 -4.9191 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1357 2.1273 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1357 2.1273 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9337 -4.0862 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9337 -4.0862 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2678 2.6240 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2678 2.6240 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4713 1.3321 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4713 1.3321 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2683 1.3352 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2683 1.3352 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3392 3.2066 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3392 3.2066 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9380 3.8953 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9380 3.8953 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3079 2.8434 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3079 2.8434 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4619 2.6132 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4619 2.6132 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6921 1.7671 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6921 1.7671 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8579 4.4286 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8579 4.4286 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
1 17 2 0 0 0 0 | 1 17 2 0 0 0 0 | ||
2 24 1 0 0 0 0 | 2 24 1 0 0 0 0 | ||
| Line 106: | Line 106: | ||
25 48 1 0 0 0 0 | 25 48 1 0 0 0 0 | ||
25 49 1 0 0 0 0 | 25 49 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
| − | NAME Methylergonovine | + | NAME Methylergonovine |
| + | ID COX00056 | ||
| + | FORMULA C20H25N3O2 | ||
| + | EXACTMASS 339.194677059 | ||
| + | AVERAGEMASS 339.43152000000003 | ||
| + | SMILES [H]OC([H])([H])C([H])(C([H])([H])C([H])([H])[H])N([H])C(=O)C([H])(C([H])=1)C([H])([H])N(C([H])([H])[H])C([H])(C([H])([H])2)C1c(c([H])4)c(c(c([H])c([H])4)3)c(c([H])n([H])3)2 | ||
M END | M END | ||
Latest revision as of 20:47, 21 February 2011
4140
CDK 9/16/09,17:7
50 53 0 0 0 0 0 0 0 0999 V2000
3.7398 0.3119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8602 3.8086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.2719 0.3117 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
8.2599 -3.8785 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
4.6000 1.8153 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
7.2558 -0.7298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3898 -1.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.1219 -1.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4718 0.3186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3739 0.8394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.1219 -2.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3898 -2.2298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.2558 -2.7298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4798 -0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.2719 -3.7713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.1436 0.8016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6038 0.8153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.7398 -3.0082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4798 -2.7367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3739 -4.2991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4718 -3.7783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7320 2.3119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8680 1.8086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7282 3.3119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 2.3053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.7952 -0.4240 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.7325 -1.3375 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.3339 -0.6472 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.4706 0.9386 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.7748 1.3123 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.9766 1.3154 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.9465 -1.0392 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.4474 0.2611 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.6841 1.1054 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.8399 1.3421 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.3576 -2.9564 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.9465 -2.4205 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.5459 -4.4286 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.3763 -4.9191 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.1357 2.1273 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.9337 -4.0862 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.2678 2.6240 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.4713 1.3321 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.2683 1.3352 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.3392 3.2066 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.9380 3.8953 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.3079 2.8434 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.4619 2.6132 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.6921 1.7671 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.8579 4.4286 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1 17 2 0 0 0 0
2 24 1 0 0 0 0
2 50 1 0 0 0 0
3 6 1 0 0 0 0
3 10 1 0 0 0 0
3 16 1 0 0 0 0
4 15 1 0 0 0 0
4 18 1 0 0 0 0
4 38 1 0 0 0 0
5 17 1 0 0 0 0
5 22 1 0 0 0 0
5 40 1 0 0 0 0
6 7 1 0 0 0 0
6 8 1 0 0 0 0
6 26 1 0 0 0 0
7 12 1 0 0 0 0
7 14 2 0 0 0 0
8 11 1 0 0 0 0
8 27 1 0 0 0 0
8 28 1 0 0 0 0
9 10 1 0 0 0 0
9 14 1 0 0 0 0
9 17 1 0 0 0 0
9 29 1 0 0 0 0
10 30 1 0 0 0 0
10 31 1 0 0 0 0
11 13 1 0 0 0 0
11 18 2 0 0 0 0
12 13 2 0 0 0 0
12 19 1 0 0 0 0
13 15 1 0 0 0 0
14 32 1 0 0 0 0
15 20 2 0 0 0 0
16 33 1 0 0 0 0
16 34 1 0 0 0 0
16 35 1 0 0 0 0
18 36 1 0 0 0 0
19 21 2 0 0 0 0
19 37 1 0 0 0 0
20 21 1 0 0 0 0
20 39 1 0 0 0 0
21 41 1 0 0 0 0
22 23 1 0 0 0 0
22 24 1 0 0 0 0
22 42 1 0 0 0 0
23 25 1 0 0 0 0
23 43 1 0 0 0 0
23 44 1 0 0 0 0
24 45 1 0 0 0 0
24 46 1 0 0 0 0
25 47 1 0 0 0 0
25 48 1 0 0 0 0
25 49 1 0 0 0 0
S SKP 6
NAME Methylergonovine
ID COX00056
FORMULA C20H25N3O2
EXACTMASS 339.194677059
AVERAGEMASS 339.43152000000003
SMILES [H]OC([H])([H])C([H])(C([H])([H])C([H])([H])[H])N([H])C(=O)C([H])(C([H])=1)C([H])([H])N(C([H])([H])[H])C([H])(C([H])([H])2)C1c(c([H])4)c(c(c([H])c([H])4)3)c(c([H])n([H])3)2
M END