Mol:COX00048
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 3783 | + | 3783 |
| − | CDK 9/16/09,17:5 | + | CDK 9/16/09,17:5 |
| − | + | ||
| − | 45 46 0 0 0 0 0 0 0 0999 V2000 | + | 45 46 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.0000 -0.5950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -0.5950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4641 2.4050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4641 2.4050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 -5.0950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 -5.0950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7321 0.4050 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 0.4050 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7321 -0.5950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 -0.5950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 -1.0950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 -1.0950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5981 0.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5981 0.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5981 -1.0950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5981 -1.0950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 -2.0950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 -2.0950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5981 1.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5981 1.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4641 0.4050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4641 0.4050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7321 -2.5950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 -2.5950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -2.5950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -2.5950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7321 -3.5950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 -3.5950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -3.5950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -3.5950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 -4.0950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 -4.0950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4641 3.4050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4641 3.4050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5981 3.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5981 3.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3301 3.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3301 3.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5981 4.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5981 4.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3301 4.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3301 4.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4641 5.4050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4641 5.4050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4682 -0.1700 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4682 -0.1700 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 -0.4750 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 -0.4750 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5981 0.0550 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5981 0.0550 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1951 0.7150 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1951 0.7150 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2881 -1.6319 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2881 -1.6319 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1350 -1.4050 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1350 -1.4050 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9081 -0.5581 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9081 -0.5581 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3860 2.4876 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3860 2.4876 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9875 1.7973 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9875 1.7973 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1541 -0.1319 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1541 -0.1319 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0010 0.0950 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0010 0.0950 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7741 0.9419 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7741 0.9419 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2690 -2.2850 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2690 -2.2850 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4631 -2.2850 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4631 -2.2850 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 0.0250 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 0.0250 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2690 -3.9050 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2690 -3.9050 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4631 -3.9050 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4631 -3.9050 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0611 3.5950 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0611 3.5950 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8671 3.5950 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8671 3.5950 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0611 5.2150 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0611 5.2150 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8671 5.2150 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8671 5.2150 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3291 -5.4050 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3291 -5.4050 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4641 6.0250 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4641 6.0250 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
1 6 1 0 0 0 0 | 1 6 1 0 0 0 0 | ||
1 37 1 0 0 0 0 | 1 37 1 0 0 0 0 | ||
| Line 94: | Line 94: | ||
21 43 1 0 0 0 0 | 21 43 1 0 0 0 0 | ||
22 45 1 0 0 0 0 | 22 45 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
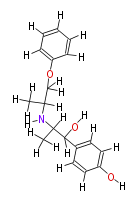
| − | NAME Isoxsuprine | + | NAME Isoxsuprine |
| + | ID COX00048 | ||
| + | FORMULA C18H23NO3 | ||
| + | EXACTMASS 301.167793607 | ||
| + | AVERAGEMASS 301.38016000000005 | ||
| + | SMILES [H]Oc(c([H])2)c([H])c([H])c(c([H])2)C([H])(O[H])C([H])(C([H])([H])[H])N([H])C([H])(C([H])([H])[H])C([H])([H])Oc(c([H])1)c([H])c([H])c([H])c([H])1 | ||
M END | M END | ||
Latest revision as of 20:47, 21 February 2011
3783
CDK 9/16/09,17:5
45 46 0 0 0 0 0 0 0 0999 V2000
2.0000 -0.5950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.4641 2.4050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 -5.0950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7321 0.4050 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
3.7321 -0.5950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 -1.0950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 0.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 -1.0950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 -2.0950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 1.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4641 0.4050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7321 -2.5950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -2.5950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7321 -3.5950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -3.5950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 -4.0950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4641 3.4050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 3.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3301 3.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 4.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3301 4.9050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4641 5.4050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4682 -0.1700 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 -0.4750 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 0.0550 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.1951 0.7150 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.2881 -1.6319 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.1350 -1.4050 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.9081 -0.5581 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.3860 2.4876 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.9875 1.7973 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.1541 -0.1319 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.0010 0.0950 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.7741 0.9419 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.2690 -2.2850 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.4631 -2.2850 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 0.0250 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.2690 -3.9050 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.4631 -3.9050 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.0611 3.5950 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.8671 3.5950 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.0611 5.2150 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.8671 5.2150 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.3291 -5.4050 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.4641 6.0250 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1 6 1 0 0 0 0
1 37 1 0 0 0 0
2 10 1 0 0 0 0
2 17 1 0 0 0 0
3 16 1 0 0 0 0
3 44 1 0 0 0 0
4 5 1 0 0 0 0
4 7 1 0 0 0 0
4 26 1 0 0 0 0
5 6 1 0 0 0 0
5 8 1 0 0 0 0
5 23 1 0 0 0 0
6 9 1 0 0 0 0
6 24 1 0 0 0 0
7 10 1 0 0 0 0
7 11 1 0 0 0 0
7 25 1 0 0 0 0
8 27 1 0 0 0 0
8 28 1 0 0 0 0
8 29 1 0 0 0 0
9 12 2 0 0 0 0
9 13 1 0 0 0 0
10 30 1 0 0 0 0
10 31 1 0 0 0 0
11 32 1 0 0 0 0
11 33 1 0 0 0 0
11 34 1 0 0 0 0
12 14 1 0 0 0 0
12 35 1 0 0 0 0
13 15 2 0 0 0 0
13 36 1 0 0 0 0
14 16 2 0 0 0 0
14 38 1 0 0 0 0
15 16 1 0 0 0 0
15 39 1 0 0 0 0
17 18 2 0 0 0 0
17 19 1 0 0 0 0
18 20 1 0 0 0 0
18 40 1 0 0 0 0
19 21 2 0 0 0 0
19 41 1 0 0 0 0
20 22 2 0 0 0 0
20 42 1 0 0 0 0
21 22 1 0 0 0 0
21 43 1 0 0 0 0
22 45 1 0 0 0 0
S SKP 6
NAME Isoxsuprine
ID COX00048
FORMULA C18H23NO3
EXACTMASS 301.167793607
AVERAGEMASS 301.38016000000005
SMILES [H]Oc(c([H])2)c([H])c([H])c(c([H])2)C([H])(O[H])C([H])(C([H])([H])[H])N([H])C([H])(C([H])([H])[H])C([H])([H])Oc(c([H])1)c([H])c([H])c([H])c([H])1
M END