Mol:COX00028
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 13505 | + | 13505 |
| − | CDK 9/16/09,17:1 | + | CDK 9/16/09,17:1 |
| − | + | ||
| − | 66 69 0 0 0 0 0 0 0 0999 V2000 | + | 66 69 0 0 0 0 0 0 0 0999 V2000 |
| − | 4.3660 3.4808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3660 3.4808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5397 4.4656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5397 4.4656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8660 0.6148 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8660 0.6148 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0000 -1.8852 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0000 -1.8852 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8660 2.6148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8660 2.6148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0000 2.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0000 2.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7321 2.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7321 2.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0000 1.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0000 1.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7321 1.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7321 1.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8660 -0.3852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8660 -0.3852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6321 3.2576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6321 3.2576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3660 3.4808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3660 3.4808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0000 -0.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0000 -0.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0000 -1.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0000 -1.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4584 4.2424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4584 4.2424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5718 2.9156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5718 2.9156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0000 -1.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0000 -1.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0000 -2.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0000 -2.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2245 4.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2245 4.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.3378 3.5584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.3378 3.5584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0000 -1.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0000 -1.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1642 4.5432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1642 4.5432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8660 4.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8660 4.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5000 -2.7512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5000 -2.7512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8660 -3.3852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8660 -3.3852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5000 -1.0192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5000 -1.0192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1340 -3.3852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1340 -3.3852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 4.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 4.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5000 -2.7512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5000 -2.7512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8660 -4.3852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8660 -4.3852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5000 -1.0192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5000 -1.0192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1340 -4.3852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1340 -4.3852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -1.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -1.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0000 -4.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0000 -4.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3894 2.0071 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3894 2.0071 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7879 2.6974 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7879 2.6974 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9441 2.6974 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9441 2.6974 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3426 2.0071 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3426 2.0071 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7879 0.5322 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7879 0.5322 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3894 1.2225 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3894 1.2225 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3426 1.2225 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3426 1.2225 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9441 0.5322 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9441 0.5322 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4766 -0.2775 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4766 -0.2775 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0781 -0.9678 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0781 -0.9678 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3894 -0.9929 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3894 -0.9929 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7879 -0.3026 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7879 -0.3026 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8758 4.4545 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8758 4.4545 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6794 2.3050 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6794 2.3050 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1168 5.4958 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1168 5.4958 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9204 3.3463 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9204 3.3463 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.6391 4.9417 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.6391 4.9417 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7584 4.9574 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7584 4.9574 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4486 4.5589 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4486 4.5589 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8100 -3.2882 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8100 -3.2882 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4030 -3.0752 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4030 -3.0752 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8100 -0.4822 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8100 -0.4822 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5970 -3.0752 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5970 -3.0752 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 4.9669 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 4.9669 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2460 4.3469 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2460 4.3469 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 3.7269 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 3.7269 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1900 -3.2882 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1900 -3.2882 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4030 -4.6952 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4030 -4.6952 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1900 -0.4822 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1900 -0.4822 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5970 -4.6952 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5970 -4.6952 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3800 -1.8852 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3800 -1.8852 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0000 -5.5052 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0000 -5.5052 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
1 12 1 0 0 0 0 | 1 12 1 0 0 0 0 | ||
1 23 1 0 0 0 0 | 1 23 1 0 0 0 0 | ||
| Line 138: | Line 138: | ||
33 65 1 0 0 0 0 | 33 65 1 0 0 0 0 | ||
34 66 1 0 0 0 0 | 34 66 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
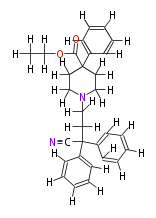
| − | NAME Diphenoxylate | + | NAME Diphenoxylate |
| + | ID COX00028 | ||
| + | FORMULA C30H32N2O2 | ||
| + | EXACTMASS 452.246378278 | ||
| + | AVERAGEMASS 452.58736000000005 | ||
| + | SMILES [H]C([H])([H])C([H])([H])OC(C(c(c4[H])c(c([H])c(c4[H])[H])[H])(C([H])([H])1)C([H])([H])C([H])([H])N(C(C(C(C#N)(c(c3[H])c(c([H])c(c([H])3)[H])[H])c(c2[H])c(c([H])c(c([H])2)[H])[H])([H])[H])([H])[H])C([H])([H])1)=O | ||
M END | M END | ||
Latest revision as of 20:46, 21 February 2011
13505
CDK 9/16/09,17:1
66 69 0 0 0 0 0 0 0 0999 V2000
4.3660 3.4808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5397 4.4656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.8660 0.6148 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
7.0000 -1.8852 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
5.8660 2.6148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0000 2.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.7321 2.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0000 1.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.7321 1.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.8660 -0.3852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.6321 3.2576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3660 3.4808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0000 -0.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0000 -1.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.4584 4.2424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.5718 2.9156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0000 -1.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0000 -2.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.2245 4.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.3378 3.5584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0000 -1.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.1642 4.5432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8660 4.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5000 -2.7512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.8660 -3.3852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5000 -1.0192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1340 -3.3852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 4.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5000 -2.7512 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.8660 -4.3852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5000 -1.0192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1340 -4.3852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -1.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0000 -4.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3894 2.0071 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.7879 2.6974 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.9441 2.6974 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.3426 2.0071 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.7879 0.5322 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.3894 1.2225 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.3426 1.2225 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.9441 0.5322 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.4766 -0.2775 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.0781 -0.9678 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.3894 -0.9929 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.7879 -0.3026 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.8758 4.4545 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.6794 2.3050 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.1168 5.4958 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.9204 3.3463 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.6391 4.9417 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.7584 4.9574 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.4486 4.5589 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.8100 -3.2882 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.4030 -3.0752 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.8100 -0.4822 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.5970 -3.0752 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 4.9669 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.2460 4.3469 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 3.7269 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.1900 -3.2882 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.4030 -4.6952 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.1900 -0.4822 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.5970 -4.6952 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.3800 -1.8852 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.0000 -5.5052 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1 12 1 0 0 0 0
1 23 1 0 0 0 0
2 12 2 0 0 0 0
3 8 1 0 0 0 0
3 9 1 0 0 0 0
3 10 1 0 0 0 0
4 21 3 0 0 0 0
5 6 1 0 0 0 0
5 7 1 0 0 0 0
5 11 1 0 0 0 0
5 12 1 0 0 0 0
6 8 1 0 0 0 0
6 35 1 0 0 0 0
6 36 1 0 0 0 0
7 9 1 0 0 0 0
7 37 1 0 0 0 0
7 38 1 0 0 0 0
8 39 1 0 0 0 0
8 40 1 0 0 0 0
9 41 1 0 0 0 0
9 42 1 0 0 0 0
10 13 1 0 0 0 0
10 43 1 0 0 0 0
10 44 1 0 0 0 0
11 15 2 0 0 0 0
11 16 1 0 0 0 0
13 14 1 0 0 0 0
13 45 1 0 0 0 0
13 46 1 0 0 0 0
14 17 1 0 0 0 0
14 18 1 0 0 0 0
14 21 1 0 0 0 0
15 19 1 0 0 0 0
15 47 1 0 0 0 0
16 20 2 0 0 0 0
16 48 1 0 0 0 0
17 24 2 0 0 0 0
17 26 1 0 0 0 0
18 25 2 0 0 0 0
18 27 1 0 0 0 0
19 22 2 0 0 0 0
19 49 1 0 0 0 0
20 22 1 0 0 0 0
20 50 1 0 0 0 0
22 51 1 0 0 0 0
23 28 1 0 0 0 0
23 52 1 0 0 0 0
23 53 1 0 0 0 0
24 29 1 0 0 0 0
24 54 1 0 0 0 0
25 30 1 0 0 0 0
25 55 1 0 0 0 0
26 31 2 0 0 0 0
26 56 1 0 0 0 0
27 32 2 0 0 0 0
27 57 1 0 0 0 0
28 58 1 0 0 0 0
28 59 1 0 0 0 0
28 60 1 0 0 0 0
29 33 2 0 0 0 0
29 61 1 0 0 0 0
30 34 2 0 0 0 0
30 62 1 0 0 0 0
31 33 1 0 0 0 0
31 63 1 0 0 0 0
32 34 1 0 0 0 0
32 64 1 0 0 0 0
33 65 1 0 0 0 0
34 66 1 0 0 0 0
S SKP 6
NAME Diphenoxylate
ID COX00028
FORMULA C30H32N2O2
EXACTMASS 452.246378278
AVERAGEMASS 452.58736000000005
SMILES [H]C([H])([H])C([H])([H])OC(C(c(c4[H])c(c([H])c(c4[H])[H])[H])(C([H])([H])1)C([H])([H])C([H])([H])N(C(C(C(C#N)(c(c3[H])c(c([H])c(c([H])3)[H])[H])c(c2[H])c(c([H])c(c([H])2)[H])[H])([H])[H])([H])[H])C([H])([H])1)=O
M END