Mol:COX00026
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 3066 | + | 3066 |
| − | CDK 9/16/09,17:1 | + | CDK 9/16/09,17:1 |
| − | + | ||
| − | 80 87 0 0 0 0 0 0 0 0999 V2000 | + | 80 87 0 0 0 0 0 0 0 0999 V2000 |
| − | 4.3933 0.5772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3933 0.5772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3710 1.1549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3710 1.1549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5851 4.5499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5851 4.5499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8554 1.3442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8554 1.3442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3743 -1.6248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3743 -1.6248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1950 1.9754 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1950 1.9754 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5724 3.1448 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5724 3.1448 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9065 -1.6251 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9065 -1.6251 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2345 -0.1215 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2345 -0.1215 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.8945 -5.8153 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.8945 -5.8153 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4717 2.1498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4717 2.1498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2830 1.5652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2830 1.5652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0244 -3.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0244 -3.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8904 -2.6666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8904 -2.6666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4996 1.9419 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4996 1.9419 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2957 2.9704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2957 2.9704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3666 0.3751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3666 0.3751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1063 -1.6182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1063 -1.6182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1144 -2.6598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1144 -2.6598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6617 3.5432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6617 3.5432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 2.8013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 2.8013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4844 3.5550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4844 3.5550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8611 1.2375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8611 1.2375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0084 -1.0974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0084 -1.0974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.7564 -3.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.7564 -3.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0244 -4.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0244 -4.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2076 3.3806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2076 3.3806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2384 -1.1215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2384 -1.1215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8904 -4.6666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8904 -4.6666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.7564 -4.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.7564 -4.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1625 -0.6038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1625 -0.6038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.7782 -1.1352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.7782 -1.1352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9065 -5.7081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9065 -5.7081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1144 -4.6735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1144 -4.6735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.3743 -4.9450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.3743 -4.9450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3083 4.3755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3083 4.3755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0084 -6.2359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0084 -6.2359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1063 -5.7151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1063 -5.7151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2203 4.7858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2203 4.7858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4970 4.9602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4970 4.9602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3210 5.7807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3210 5.7807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5977 5.9551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5977 5.9551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5097 6.3654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5097 6.3654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2971 1.3180 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2971 1.3180 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2915 -3.5973 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2915 -3.5973 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.6298 -2.2473 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.6298 -2.2473 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9338 1.6884 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9338 1.6884 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6923 1.3526 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6923 1.3526 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9852 2.4734 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9852 2.4734 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3727 -2.0474 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3727 -2.0474 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5027 -2.5584 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5027 -2.5584 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9098 -3.2451 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9098 -3.2451 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9685 4.0820 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9685 4.0820 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1582 3.9050 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1582 3.9050 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5832 3.2604 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5832 3.2604 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4997 2.4351 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4997 2.4351 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.4094 -0.6245 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4094 -0.6245 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6111 -0.6214 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6111 -0.6214 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.3670 -3.2743 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.3670 -3.2743 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.9685 -2.5840 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.9685 -2.5840 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7703 0.1906 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7703 0.1906 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3599 2.7796 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3599 2.7796 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8259 3.4262 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8259 3.4262 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5555 -0.4772 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5555 -0.4772 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0359 -1.2107 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0359 -1.2107 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7694 -0.7304 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7694 -0.7304 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3086 0.5381 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3086 0.5381 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.0820 -1.6756 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.0820 -1.6756 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.3187 -0.8314 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.3187 -0.8314 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.4744 -0.5947 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.4744 -0.5947 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5811 -4.3573 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5811 -4.3573 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.9922 -4.8932 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.9922 -4.8932 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.1805 -6.3654 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.1805 -6.3654 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0108 -6.8559 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0108 -6.8559 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5682 -6.0230 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5682 -6.0230 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.7233 4.4233 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.7233 4.4233 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9316 4.7058 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9316 4.7058 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8864 6.0351 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8864 6.0351 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0947 6.3176 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0947 6.3176 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5721 6.9822 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5721 6.9822 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
1 12 1 0 0 0 0 | 1 12 1 0 0 0 0 | ||
1 17 1 0 0 0 0 | 1 17 1 0 0 0 0 | ||
| Line 170: | Line 170: | ||
42 79 1 0 0 0 0 | 42 79 1 0 0 0 0 | ||
43 80 1 0 0 0 0 | 43 80 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
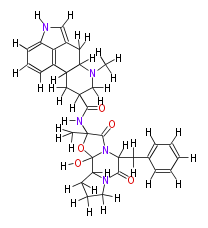
| − | NAME Dihydroergotamine | + | NAME Dihydroergotamine |
| + | ID COX00026 | ||
| + | FORMULA C33H37N5O5 | ||
| + | EXACTMASS 583.279469319 | ||
| + | AVERAGEMASS 583.67758 | ||
| + | SMILES [H]C(N13)(C(O4)(N(C(C(N([H])C(=O)C([H])(C([H])([H])5)C(N(C([H])(C8([H])[H])C5([H])c(c([H])7)c(c86)c(c([H])c7[H])n([H])c([H])6)C([H])([H])[H])([H])[H])4C([H])([H])[H])=O)C(C3=O)([H])C([H])([H])c(c2[H])c(c(c(c([H])2)[H])[H])[H])O[H])C([H])([H])C([H])([H])C1([H])[H] | ||
M END | M END | ||
Latest revision as of 20:46, 21 February 2011
3066
CDK 9/16/09,17:1
80 87 0 0 0 0 0 0 0 0999 V2000
4.3933 0.5772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3710 1.1549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5851 4.5499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.8554 1.3442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3743 -1.6248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1950 1.9754 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
3.5724 3.1448 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
8.9065 -1.6251 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
6.2345 -0.1215 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
9.8945 -5.8153 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
3.4717 2.1498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2830 1.5652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.0244 -3.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.8904 -2.6666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4996 1.9419 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2957 2.9704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3666 0.3751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.1063 -1.6182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.1144 -2.6598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6617 3.5432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 2.8013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4844 3.5550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.8611 1.2375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.0084 -1.0974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.7564 -3.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.0244 -4.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2076 3.3806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2384 -1.1215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.8904 -4.6666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.7564 -4.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1625 -0.6038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.7782 -1.1352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.9065 -5.7081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.1144 -4.6735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.3743 -4.9450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3083 4.3755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.0084 -6.2359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.1063 -5.7151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.2203 4.7858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4970 4.9602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3210 5.7807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5977 5.9551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5097 6.3654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2971 1.3180 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.2915 -3.5973 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.6298 -2.2473 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.9338 1.6884 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.6923 1.3526 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.9852 2.4734 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.3727 -2.0474 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.5027 -2.5584 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.9098 -3.2451 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.9685 4.0820 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.1582 3.9050 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.5832 3.2604 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.4997 2.4351 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.4094 -0.6245 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.6111 -0.6214 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.3670 -3.2743 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.9685 -2.5840 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.7703 0.1906 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.3599 2.7796 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.8259 3.4262 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.5555 -0.4772 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.0359 -1.2107 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.7694 -0.7304 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.3086 0.5381 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.0820 -1.6756 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.3187 -0.8314 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.4744 -0.5947 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.5811 -4.3573 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.9922 -4.8932 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.1805 -6.3654 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.0108 -6.8559 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.5682 -6.0230 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.7233 4.4233 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.9316 4.7058 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.8864 6.0351 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.0947 6.3176 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.5721 6.9822 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1 12 1 0 0 0 0
1 17 1 0 0 0 0
2 12 1 0 0 0 0
2 67 1 0 0 0 0
3 22 2 0 0 0 0
4 23 2 0 0 0 0
5 28 2 0 0 0 0
6 12 1 0 0 0 0
6 16 1 0 0 0 0
6 23 1 0 0 0 0
7 11 1 0 0 0 0
7 20 1 0 0 0 0
7 22 1 0 0 0 0
8 14 1 0 0 0 0
8 24 1 0 0 0 0
8 32 1 0 0 0 0
9 17 1 0 0 0 0
9 28 1 0 0 0 0
9 61 1 0 0 0 0
10 33 1 0 0 0 0
10 35 1 0 0 0 0
10 73 1 0 0 0 0
11 12 1 0 0 0 0
11 15 1 0 0 0 0
11 44 1 0 0 0 0
13 14 1 0 0 0 0
13 19 1 0 0 0 0
13 26 1 0 0 0 0
13 45 1 0 0 0 0
14 25 1 0 0 0 0
14 46 1 0 0 0 0
15 21 1 0 0 0 0
15 47 1 0 0 0 0
15 48 1 0 0 0 0
16 22 1 0 0 0 0
16 27 1 0 0 0 0
16 49 1 0 0 0 0
17 23 1 0 0 0 0
17 31 1 0 0 0 0
18 19 1 0 0 0 0
18 24 1 0 0 0 0
18 28 1 0 0 0 0
18 50 1 0 0 0 0
19 51 1 0 0 0 0
19 52 1 0 0 0 0
20 21 1 0 0 0 0
20 53 1 0 0 0 0
20 54 1 0 0 0 0
21 55 1 0 0 0 0
21 56 1 0 0 0 0
24 57 1 0 0 0 0
24 58 1 0 0 0 0
25 30 1 0 0 0 0
25 59 1 0 0 0 0
25 60 1 0 0 0 0
26 29 2 0 0 0 0
26 34 1 0 0 0 0
27 36 1 0 0 0 0
27 62 1 0 0 0 0
27 63 1 0 0 0 0
29 30 1 0 0 0 0
29 33 1 0 0 0 0
30 35 2 0 0 0 0
31 64 1 0 0 0 0
31 65 1 0 0 0 0
31 66 1 0 0 0 0
32 68 1 0 0 0 0
32 69 1 0 0 0 0
32 70 1 0 0 0 0
33 37 2 0 0 0 0
34 38 2 0 0 0 0
34 71 1 0 0 0 0
35 72 1 0 0 0 0
36 39 2 0 0 0 0
36 40 1 0 0 0 0
37 38 1 0 0 0 0
37 74 1 0 0 0 0
38 75 1 0 0 0 0
39 41 1 0 0 0 0
39 76 1 0 0 0 0
40 42 2 0 0 0 0
40 77 1 0 0 0 0
41 43 2 0 0 0 0
41 78 1 0 0 0 0
42 43 1 0 0 0 0
42 79 1 0 0 0 0
43 80 1 0 0 0 0
S SKP 6
NAME Dihydroergotamine
ID COX00026
FORMULA C33H37N5O5
EXACTMASS 583.279469319
AVERAGEMASS 583.67758
SMILES [H]C(N13)(C(O4)(N(C(C(N([H])C(=O)C([H])(C([H])([H])5)C(N(C([H])(C8([H])[H])C5([H])c(c([H])7)c(c86)c(c([H])c7[H])n([H])c([H])6)C([H])([H])[H])([H])[H])4C([H])([H])[H])=O)C(C3=O)([H])C([H])([H])c(c2[H])c(c(c(c([H])2)[H])[H])[H])O[H])C([H])([H])C([H])([H])C1([H])[H]
M END