Mol:COX00021
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 304 | + | 304 |
| − | CDK 9/16/09,17:0 | + | CDK 9/16/09,17:0 |
| − | + | ||
| − | 74 77 0 0 0 0 0 0 0 0999 V2000 | + | 74 77 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.5357 -3.6598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5357 -3.6598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9288 -0.6074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9288 -0.6074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9288 -1.6074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9288 -1.6074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0628 -2.1074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0628 -2.1074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1968 -1.6074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1968 -1.6074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8750 -0.3027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8750 -0.3027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0628 -0.1074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0628 -0.1074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2868 -2.1143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2868 -2.1143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8750 -1.9122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8750 -1.9122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1968 -0.6074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1968 -0.6074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.4587 -1.1074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.4587 -1.1074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0789 -3.1489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0789 -3.1489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.1857 0.6478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.1857 0.6478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9288 0.3926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9288 0.3926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2787 -3.1559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2787 -3.1559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3599 -1.5502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3599 -1.5502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1808 -3.6767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1808 -3.6767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2945 -1.1143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2945 -1.1143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3433 -3.7056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3433 -3.7056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4121 -2.0781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4121 -2.0781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.1642 0.8540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.1642 0.8540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4037 -3.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4037 -3.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.5179 1.3921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.5179 1.3921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.4749 1.8046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.4749 1.8046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.4534 2.0108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.4534 2.0108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.7640 2.9613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.7640 2.9613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.7425 3.1675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.7425 3.1675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.0962 3.7056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.0962 3.7056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0188 -2.4527 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0188 -2.4527 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8022 -2.5267 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8022 -2.5267 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9329 -1.1824 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9329 -1.1824 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.7148 -0.1708 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.7148 -0.1708 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4613 0.3675 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4613 0.3675 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6643 0.3675 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6643 0.3675 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.6240 -2.4791 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.6240 -2.4791 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.4124 -2.2214 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.4124 -2.2214 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9847 -0.0248 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9847 -0.0248 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5862 -0.7151 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5862 -0.7151 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.9195 -1.5222 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.9195 -1.5222 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.9195 -0.6927 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.9195 -0.6927 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2969 -3.7293 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2969 -3.7293 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6880 -3.0334 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6880 -3.0334 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.4498 1.4558 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.4498 1.4558 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.5488 0.3926 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.5488 0.3926 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9288 1.0126 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9288 1.0126 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3088 0.3926 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3088 0.3926 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7681 -1.0835 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7681 -1.0835 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9700 -1.0682 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9700 -1.0682 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1832 -4.2967 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1832 -4.2967 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9145 -1.1191 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9145 -1.1191 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2993 -0.4943 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2993 -0.4943 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6745 -1.1095 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6745 -1.1095 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9460 -4.1816 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9460 -4.1816 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7443 -4.1785 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7443 -4.1785 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2075 -1.4929 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2075 -1.4929 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8004 -2.1795 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8004 -2.1795 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.1848 0.2344 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.1848 0.2344 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.7780 0.7667 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.7780 0.7667 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4021 -4.0131 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4021 -4.0131 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9793 1.8062 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9793 1.8062 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1038 1.8536 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1038 1.8536 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0564 0.9781 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0564 0.9781 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.4543 2.4242 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.4543 2.4242 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.8611 1.8919 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.8611 1.8919 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -3.3477 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -3.3477 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.4739 1.3911 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.4739 1.3911 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.0672 1.9234 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.0672 1.9234 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.1781 2.4998 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.1781 2.4998 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.8704 2.5608 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.8704 2.5608 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.3492 3.2954 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.3492 3.2954 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.6147 3.7742 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.6147 3.7742 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.5577 4.1197 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.5577 4.1197 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.6821 4.1671 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.6821 4.1671 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.6347 3.2915 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.6347 3.2915 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
1 22 1 0 0 0 0 | 1 22 1 0 0 0 0 | ||
1 65 1 0 0 0 0 | 1 65 1 0 0 0 0 | ||
| Line 154: | Line 154: | ||
28 73 1 0 0 0 0 | 28 73 1 0 0 0 0 | ||
28 74 1 0 0 0 0 | 28 74 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
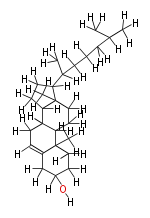
| − | NAME Cholesterol | + | NAME Cholesterol |
| + | ID COX00021 | ||
| + | FORMULA C27H46O | ||
| + | EXACTMASS 386.354866094 | ||
| + | AVERAGEMASS 386.65353999999996 | ||
| + | SMILES [H]C(C([H])([H])C([H])([H])C([H])([H])C(C(C4([H])[H])([H])C(C([H])([H])[H])(C3([H])[H])C(C4([H])[H])([H])C([H])(C([H])(C3([H])[H])1)C([H])([H])C([H])=C(C([H])([H])2)C1(C([H])([H])C([H])([H])C([H])(O[H])2)C([H])([H])[H])(C([H])([H])[H])[H])(C([H])([H])[H])C([H])([H])[H] | ||
M END | M END | ||
Latest revision as of 20:46, 21 February 2011
304
CDK 9/16/09,17:0
74 77 0 0 0 0 0 0 0 0999 V2000
2.5357 -3.6598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.9288 -0.6074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.9288 -1.6074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.0628 -2.1074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.1968 -1.6074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.8750 -0.3027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.0628 -0.1074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2868 -2.1143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.8750 -1.9122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.1968 -0.6074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.4587 -1.1074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.0789 -3.1489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.1857 0.6478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.9288 0.3926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2787 -3.1559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3599 -1.5502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.1808 -3.6767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2945 -1.1143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3433 -3.7056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4121 -2.0781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.1642 0.8540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4037 -3.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.5179 1.3921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.4749 1.8046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.4534 2.0108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.7640 2.9613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.7425 3.1675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.0962 3.7056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.0188 -2.4527 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.8022 -2.5267 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.9329 -1.1824 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.7148 -0.1708 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.4613 0.3675 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.6643 0.3675 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.6240 -2.4791 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.4124 -2.2214 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.9847 -0.0248 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.5862 -0.7151 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.9195 -1.5222 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.9195 -0.6927 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.2969 -3.7293 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.6880 -3.0334 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.4498 1.4558 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.5488 0.3926 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.9288 1.0126 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.3088 0.3926 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.7681 -1.0835 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.9700 -1.0682 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.1832 -4.2967 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.9145 -1.1191 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
5.2993 -0.4943 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.6745 -1.1095 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.9460 -4.1816 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.7443 -4.1785 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.2075 -1.4929 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.8004 -2.1795 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.1848 0.2344 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.7780 0.7667 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.4021 -4.0131 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.9793 1.8062 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.1038 1.8536 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.0564 0.9781 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.4543 2.4242 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.8611 1.8919 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -3.3477 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
11.4739 1.3911 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
12.0672 1.9234 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
12.1781 2.4998 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
12.8704 2.5608 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
13.3492 3.2954 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
12.6147 3.7742 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
11.5577 4.1197 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.6821 4.1671 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
10.6347 3.2915 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1 22 1 0 0 0 0
1 65 1 0 0 0 0
2 3 1 0 0 0 0
2 6 1 0 0 0 0
2 7 1 0 0 0 0
2 14 1 0 0 0 0
3 4 1 0 0 0 0
3 9 1 0 0 0 0
3 29 1 0 0 0 0
4 5 1 0 0 0 0
4 12 1 0 0 0 0
4 30 1 0 0 0 0
5 8 1 0 0 0 0
5 10 1 0 0 0 0
5 31 1 0 0 0 0
6 11 1 0 0 0 0
6 13 1 0 0 0 0
6 32 1 0 0 0 0
7 10 1 0 0 0 0
7 33 1 0 0 0 0
7 34 1 0 0 0 0
8 15 1 0 0 0 0
8 16 1 0 0 0 0
8 18 1 0 0 0 0
9 11 1 0 0 0 0
9 35 1 0 0 0 0
9 36 1 0 0 0 0
10 37 1 0 0 0 0
10 38 1 0 0 0 0
11 39 1 0 0 0 0
11 40 1 0 0 0 0
12 17 1 0 0 0 0
12 41 1 0 0 0 0
12 42 1 0 0 0 0
13 21 1 0 0 0 0
13 23 1 0 0 0 0
13 43 1 0 0 0 0
14 44 1 0 0 0 0
14 45 1 0 0 0 0
14 46 1 0 0 0 0
15 17 2 0 0 0 0
15 19 1 0 0 0 0
16 20 1 0 0 0 0
16 47 1 0 0 0 0
16 48 1 0 0 0 0
17 49 1 0 0 0 0
18 50 1 0 0 0 0
18 51 1 0 0 0 0
18 52 1 0 0 0 0
19 22 1 0 0 0 0
19 53 1 0 0 0 0
19 54 1 0 0 0 0
20 22 1 0 0 0 0
20 55 1 0 0 0 0
20 56 1 0 0 0 0
21 24 1 0 0 0 0
21 57 1 0 0 0 0
21 58 1 0 0 0 0
22 59 1 0 0 0 0
23 60 1 0 0 0 0
23 61 1 0 0 0 0
23 62 1 0 0 0 0
24 25 1 0 0 0 0
24 63 1 0 0 0 0
24 64 1 0 0 0 0
25 26 1 0 0 0 0
25 66 1 0 0 0 0
25 67 1 0 0 0 0
26 27 1 0 0 0 0
26 28 1 0 0 0 0
26 68 1 0 0 0 0
27 69 1 0 0 0 0
27 70 1 0 0 0 0
27 71 1 0 0 0 0
28 72 1 0 0 0 0
28 73 1 0 0 0 0
28 74 1 0 0 0 0
S SKP 6
NAME Cholesterol
ID COX00021
FORMULA C27H46O
EXACTMASS 386.354866094
AVERAGEMASS 386.65353999999996
SMILES [H]C(C([H])([H])C([H])([H])C([H])([H])C(C(C4([H])[H])([H])C(C([H])([H])[H])(C3([H])[H])C(C4([H])[H])([H])C([H])(C([H])(C3([H])[H])1)C([H])([H])C([H])=C(C([H])([H])2)C1(C([H])([H])C([H])([H])C([H])(O[H])2)C([H])([H])[H])(C([H])([H])[H])[H])(C([H])([H])[H])C([H])([H])[H]
M END