Mol:COX00003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | 6077 | + | 6077 |
| − | CDK 9/16/09,16:56 | + | CDK 9/16/09,16:56 |
| − | + | ||
| − | 45 47 0 0 0 0 0 0 0 0999 V2000 | + | 45 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 4.6660 -2.9827 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6660 -2.9827 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1923 0.0415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1923 0.0415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6660 -0.9827 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6660 -0.9827 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9340 2.0173 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9340 2.0173 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6660 0.0173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6660 0.0173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8000 0.5173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8000 0.5173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5321 -1.4827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5321 -1.4827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8000 -1.4827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8000 -1.4827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8000 1.5173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8000 1.5173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5321 -2.4827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5321 -2.4827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8000 -2.4827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8000 -2.4827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4260 -0.9480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4260 -0.9480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9061 -0.9480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9061 -0.9480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3321 -1.4619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3321 -1.4619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4260 -3.0173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4260 -3.0173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9061 -3.0173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9061 -3.0173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3321 -2.5035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3321 -2.5035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -1.4619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -1.4619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9340 3.0173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9340 3.0173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0680 1.5173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0680 1.5173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -2.5035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -2.5035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.1962 -0.9585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.1962 -0.9585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.0641 -1.4552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.0641 -1.4552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2766 -0.0903 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2766 -0.0903 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8781 0.5999 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8781 0.5999 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1894 0.6250 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1894 0.6250 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5880 -0.0653 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5880 -0.0653 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4106 1.4097 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4106 1.4097 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0121 2.0999 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0121 2.0999 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4188 -0.3281 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4188 -0.3281 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9132 -0.3281 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9132 -0.3281 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4188 -3.6373 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4188 -3.6373 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9132 -3.6373 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9132 -3.6373 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8678 -2.8156 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8678 -2.8156 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4643 -1.1498 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4643 -1.1498 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5540 3.0173 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5540 3.0173 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9340 3.6373 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9340 3.6373 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3140 3.0173 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3140 3.0173 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7580 2.0543 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7580 2.0543 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5310 1.2073 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5310 1.2073 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3780 0.9804 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3780 0.9804 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4643 -2.8156 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4643 -2.8156 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.7562 -1.9933 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.7562 -1.9933 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.6022 -1.7631 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.6022 -1.7631 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.3720 -0.9171 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.3720 -0.9171 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
1 10 1 0 0 0 0 | 1 10 1 0 0 0 0 | ||
1 11 1 0 0 0 0 | 1 11 1 0 0 0 0 | ||
| Line 95: | Line 95: | ||
23 44 1 0 0 0 0 | 23 44 1 0 0 0 0 | ||
23 45 1 0 0 0 0 | 23 45 1 0 0 0 0 | ||
| − | S SKP | + | S SKP 6 |
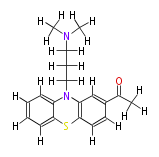
| − | NAME Acepromazine | + | NAME Acepromazine |
| + | ID COX00003 | ||
| + | FORMULA C19H22N2OS | ||
| + | EXACTMASS 326.145284026 | ||
| + | AVERAGEMASS 326.45686 | ||
| + | SMILES [H]C([H])([H])C(=O)c(c([H])3)c([H])c(c1c([H])3)N(C([H])([H])C([H])([H])C([H])([H])N(C([H])([H])[H])C([H])([H])[H])c(c([H])2)c(c([H])c([H])c([H])2)S1 | ||
M END | M END | ||
Latest revision as of 20:46, 21 February 2011
6077
CDK 9/16/09,16:56
45 47 0 0 0 0 0 0 0 0999 V2000
4.6660 -2.9827 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
8.1923 0.0415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6660 -0.9827 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
2.9340 2.0173 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
4.6660 0.0173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8000 0.5173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5321 -1.4827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8000 -1.4827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8000 1.5173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5321 -2.4827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8000 -2.4827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.4260 -0.9480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9061 -0.9480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3321 -1.4619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.4260 -3.0173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9061 -3.0173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3321 -2.5035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -1.4619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9340 3.0173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0680 1.5173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -2.5035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.1962 -0.9585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.0641 -1.4552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2766 -0.0903 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.8781 0.5999 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.1894 0.6250 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.5880 -0.0653 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.4106 1.4097 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
4.0121 2.0999 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.4188 -0.3281 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.9132 -0.3281 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
6.4188 -3.6373 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.9132 -3.6373 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
7.8678 -2.8156 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.4643 -1.1498 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
3.5540 3.0173 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.9340 3.6373 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.3140 3.0173 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.7580 2.0543 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.5310 1.2073 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
2.3780 0.9804 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1.4643 -2.8156 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
8.7562 -1.9933 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.6022 -1.7631 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
9.3720 -0.9171 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
1 10 1 0 0 0 0
1 11 1 0 0 0 0
2 22 2 0 0 0 0
3 5 1 0 0 0 0
3 7 1 0 0 0 0
3 8 1 0 0 0 0
4 9 1 0 0 0 0
4 19 1 0 0 0 0
4 20 1 0 0 0 0
5 6 1 0 0 0 0
5 24 1 0 0 0 0
5 25 1 0 0 0 0
6 9 1 0 0 0 0
6 26 1 0 0 0 0
6 27 1 0 0 0 0
7 10 2 0 0 0 0
7 12 1 0 0 0 0
8 11 1 0 0 0 0
8 13 2 0 0 0 0
9 28 1 0 0 0 0
9 29 1 0 0 0 0
10 15 1 0 0 0 0
11 16 2 0 0 0 0
12 14 2 0 0 0 0
12 30 1 0 0 0 0
13 18 1 0 0 0 0
13 31 1 0 0 0 0
14 17 1 0 0 0 0
14 22 1 0 0 0 0
15 17 2 0 0 0 0
15 32 1 0 0 0 0
16 21 1 0 0 0 0
16 33 1 0 0 0 0
17 34 1 0 0 0 0
18 21 2 0 0 0 0
18 35 1 0 0 0 0
19 36 1 0 0 0 0
19 37 1 0 0 0 0
19 38 1 0 0 0 0
20 39 1 0 0 0 0
20 40 1 0 0 0 0
20 41 1 0 0 0 0
21 42 1 0 0 0 0
22 23 1 0 0 0 0
23 43 1 0 0 0 0
23 44 1 0 0 0 0
23 45 1 0 0 0 0
S SKP 6
NAME Acepromazine
ID COX00003
FORMULA C19H22N2OS
EXACTMASS 326.145284026
AVERAGEMASS 326.45686
SMILES [H]C([H])([H])C(=O)c(c([H])3)c([H])c(c1c([H])3)N(C([H])([H])C([H])([H])C([H])([H])N(C([H])([H])[H])C([H])([H])[H])c(c([H])2)c(c([H])c([H])c([H])2)S1
M END