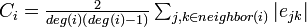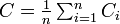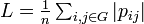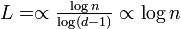Aritalab:Lecture/NetworkBiology/Erdos-Renyi Model
From Metabolomics.JP
ここでは頂点がすべて連結した無向グラフを扱う。
Contents |
歴史
- 1998 D. J. Watts, Steven Strogatz "Collective dynamics of 'small-world' networks". Nature 393: 440–442. doi:10.1038/30918.
クラスター係数と平均頂点間距離
Link Analysisではグラフにおける各頂点の尺度としてcentralityとprestigeを紹介した。ここではネットワーク全体についての尺度としてクラスター係数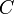 と平均頂点間距離
と平均頂点間距離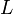 を紹介する。
を紹介する。
クラスター係数
クラスター係数は隣接する頂点間の辺の密度の平均値にあたる。まず各頂点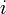 におけるクラスター係数を以下のように定義する。辺の長さはすべて1とする
におけるクラスター係数を以下のように定義する。辺の長さはすべて1とする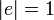 。
。
グラフ全体のクラスター係数はその平均値になる。
平均頂点間距離
平均頂点間距離はその字のごとく全頂点間の最短路の平均値にあたる。ここでは頂点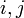 間の最短経路を
間の最短経路を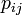 と書く。距離
と書く。距離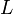 以内にある頂点の数を数えることで求められる。
以内にある頂点の数を数えることで求められる。
様々なグラフ
完全グラフ
全ての頂点間に辺を持つグラフは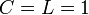 。
。
格子
平面の場合は三角格子、正方格子が考えられる。特に、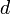 次元空間において辺の長さが単位距離の格子をZdと書く。
次元空間において辺の長さが単位距離の格子をZdと書く。
- 例. Z2において、
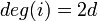
クラスター係数は定義のままだと0になって面白くないので、最短距離が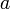 以下の点には全て辺を持つZ'dを考えよう。
以下の点には全て辺を持つZ'dを考えよう。
- 例. Z'dのクラスター係数は
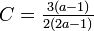 、平均頂点間距離は
、平均頂点間距離は![\textstyle L=\sqrt[d]{n}/a](/mediawiki/images/math/f/5/8/f583b41db535b1a73d0d831b87ea2808.png)
木
全頂点が同じ次数を持つ木を考える。中心の頂点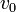 を根(root)と呼ぶ。木は定義からサイクルを持たないので
を根(root)と呼ぶ。木は定義からサイクルを持たないので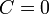 。平均頂点間距離は
。平均頂点間距離は
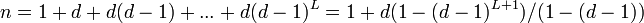 ∝
∝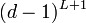
を用いて
Erdős–Rényiグラフ
各頂点間(全部で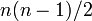 箇所)に一定の確率
箇所)に一定の確率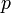 で独立に辺を作成したランダムグラフをErdős–Rényiグラフと呼ぶ。各頂点に注目してみると
で独立に辺を作成したランダムグラフをErdős–Rényiグラフと呼ぶ。各頂点に注目してみると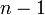 点に対して確率
点に対して確率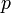 で辺を張るため、次数は二項分布
で辺を張るため、次数は二項分布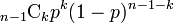 に従う。すなわち次数の平均は
に従う。すなわち次数の平均は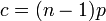 と書ける。
と書ける。
ネットワークの進化
次数の平均値 に基づくグラフ形状の変化を検証しよう。
に基づくグラフ形状の変化を検証しよう。
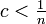
- 辺の生成される確率が
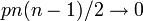 になるため、辺を持たない。
になるため、辺を持たない。
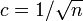
- 木が生成され始める。
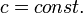
- 閉路を持つ。ただし、
 を定数とおいたことで二項分布をポアソン近似した場合、次数は
を定数とおいたことで二項分布をポアソン近似した場合、次数は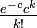 に従う。平均値はもちろん
に従う。平均値はもちろん 。
。
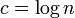
- 連結になる。ある頂点が辺を全く持たない確率は
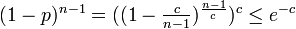 。したがって、そのような頂点がグラフ中に存在しない確率の上限は
。したがって、そのような頂点がグラフ中に存在しない確率の上限は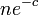 となり、これを0に収束させるには例えば
となり、これを0に収束させるには例えば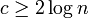 。
。
この結果は、Erdős–Rényiグラフが連結であるためには次数が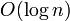 以上であることを意味し、現実のネットワークのモデルとしてはあまり適切でないことがわかる。
以上であることを意味し、現実のネットワークのモデルとしてはあまり適切でないことがわかる。
クラスター係数
辺が張られる確率は全て独立なので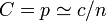 。
。