< Aritalab:Lecture | Biochem(Difference between revisions)
|
|
| Line 10: |
Line 10: |
| | ==フィードバックループ== | | ==フィードバックループ== |
| | フィードバックは生体内の代表的なモジュールで、特に遺伝子発現や代謝のホメオスタシスを保つのに必要です。 | | フィードバックは生体内の代表的なモジュールで、特に遺伝子発現や代謝のホメオスタシスを保つのに必要です。 |
| − | {|
| + | 入力信号を高めるように働く効果を正、弱めるように働く場合を負といいます。 |
| − | |valign="top"|
| + | |
| − | Mitogen-activated Protein Kinase (MAPK) は全身の細胞で発現し、細胞外のシグナルを核内に伝える代表的なシグナル伝達経路です。
| + | |
| − | MAPK とは総称で、代表的なものにERK1/2などがあります。ここでは MAPK が MAPキナーゼキナーゼ (MAPKK) により 2 段階のリン酸化を受け、MAPKK はMAPKKキナーゼにより 2 段階のリン酸化を受けるモデルになっています。最終的にできるMAPK二リン酸は、MKKK を抑制しています。
| + | |
| − | このサイクルに一つだけ「抑制」の効果があるので、負のフィードバックループになっています。
| + | |
| | | | |
| − | 初期値をみると MKKK, MKK, MAPK がそれぞれ 90, 280, 280 になっています。MKKKのリン酸化だけを考えてみましょう。もしもフィードバックループがないなら、MKKKとMKKK-Pは平衡状態になり、特に量の変動がない状態に落ち着くはずです。<br/>
| + | ===負のフィードバック=== |
| − | [[File:Lecture-Biochem-Modularity-MKKKonly.png|300px|MAPK pathway by Cell Designer]]
| + | 負のフィードバックは出力量に比例して入力部分を抑えるように働くため、出力を |
| − | |
| + | * 平衡状態に導いて安定させる、か |
| − | [[File:Lecture-Biochem-Modularity-MAPK.png|400px|MAPK pathway by Cell Designer]] | + | * 振動させる |
| − | |}
| + | 働きを持ちます。安定効果は想像しやすいですが、振動現象はわかりにくいかもしれません。 |
| − | {|
| + | シミュレーションソフトを用いると、その動きを実感することができます。([[Aritalab:Lecture/Biochem/Modularity/NF|Cell Designerによるシミュレーションページへ]]) |
| − | |valign="top"|
| + | ===正のフィードバック=== |
| − | つぎにMKKKとMKKだけに注目したネットワークを作って動きを見てみます。上の図にあった黄色と赤のライン (MKKK) はこの図において黄色と緑になっています。MKKの三つが左から順にそれぞれ赤、水色、青です。赤と青に注目してください。MKKKの動きと同じです。<br/>
| + | 正のフィードバックは入力を増強させるように働くため、出力を 0 か 1 かといった極端な値に固定する働きを持ちます。 |
| − | [[File:Lecture-Biochem-Modularity-MKKKandMKKsim.png|300px|MKKK and MKK]]
| + | |
| | | | |
| − | |valign="top"|
| |
| − | [[File:Lecture-Biochem-Modularity-MKKKandMKK.png|400px|MKKK and MKK]]
| |
| − | |}
| |
| − | {|
| |
| − | |valign="top"|
| |
| − | これはMKKK, MKK, MAPK とカスケードを足しても同じ動きをします。フィードバックがない状態では、全くリン酸化されていないMAPK (黄緑) と 二回リン酸化された MAPK (灰色) 関係は、初期状態から量が逆転しておわります。<br/>
| |
| − | [[File:Lecture-Biochem-Modularity-MKKKandMKKandMAPKsim.png|300px|MKKK and MKK and MAPK]]
| |
| − |
| |
| − | |valign="top"|
| |
| − | [[File:Lecture-Biochem-Modularity-MKKKandMKKandMAPK.png|400px|MKKK and MKK and MAPK]]
| |
| − | |}
| |
| − |
| |
| − | {|
| |
| − | |valign="top"|
| |
| − | ここで最初のネットワークに戻してシミュレーションをやってみましょう。黄色と水色、赤とマゼンタ、黄緑と灰色が相補的な動きを見せるところは一緒です。しかし、灰色が上がってくると量が増えていた水色 (MKKK) が減ってくることに注意してください。水色と黄色はゆっくりと反転して初期状態に戻っていきます。それに遅れて赤とマゼンタが戻っていくこともわかります。
| |
| − | |valign="top"|
| |
| − | [[File:Lecture-Biochem-Modularity-MAPKsim.png|400px|MKKK and MKK and MAPK]]
| |
| − | |}
| |
| | | | |
| | ==フィードフォワードループ== | | ==フィードフォワードループ== |
Revision as of 06:01, 20 June 2011
バイオロジーとモジュール
- From molecular to modular cell biology
- Hartwell LH, Hopfield JJ, Leibler S, Murray AW in Nature 402(6761 Suppl):C47-52
フィードバックループ
フィードバックは生体内の代表的なモジュールで、特に遺伝子発現や代謝のホメオスタシスを保つのに必要です。
入力信号を高めるように働く効果を正、弱めるように働く場合を負といいます。
負のフィードバック
負のフィードバックは出力量に比例して入力部分を抑えるように働くため、出力を
働きを持ちます。安定効果は想像しやすいですが、振動現象はわかりにくいかもしれません。
シミュレーションソフトを用いると、その動きを実感することができます。(Cell Designerによるシミュレーションページへ)
正のフィードバック
正のフィードバックは入力を増強させるように働くため、出力を 0 か 1 かといった極端な値に固定する働きを持ちます。
フィードフォワードループ
2000年を過ぎて急に注目され始めたモジュールが、フィードフォワードループです。とりわけ Uri Alon のグループが解析する大腸菌の転写因子に関するモデルが有名です。
大腸菌は周りの環境を数秒間で感知し、数分のうちに必要なタンパク質の合成を開始します。このような迅速な対応は、フィードバックループでは難しいと考えられています。大腸菌には転写制御因子が400ほどあり、これらが遺伝子とどのような相互作用をするのか、詳細なネットワーク解析がなされました。
- R Milo, S Shen-Orr, S Itzkovitz, N Kashtan, D Chklovskii, U Alon )2002) "Network Motifs: Simple Building Blocks of Complex Networks" Science, 298:824-827
- U Alon (2007) "Network motifs: theory and experimental approaches" Nature Reviews Genetics 8, 450-461
ネットワークモチーフ
|
転写制御ネットワークの中に、ランダムなネットワークと比較して頻出するパターンをネットワークモチーフと呼びます。
たとえば大腸菌の転写制御因子のおよそ1割は自分自身を制御するフィードバックループを持っています。フィードバックはタンパク質の産生量を安定化させるのに必要なモチーフというわけです。さらに、3つの遺伝子間の制御関係を調べてみると、考えられる13のパターンの中で、フィードフォワードループ (FFL) といわれる形が最も多い(40個程度ある)ことがわかりました。それに対して、3頂点のフィードバックループは大腸菌の転写制御因子には1つもありません。
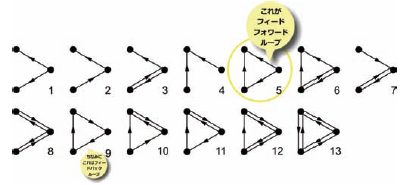
FFLには、負の制御と正の制御を考えれば 23 = 8 通りの制御関係が考えられます。そのうち、論理的につじつまがあう制御関係は半分です。
大腸菌と酵母の転写制御因子を調べると、8 通りの制御関係のうち 2 通りだけが頻出することがわかりました。
コヒーレントなフィードフォワードループは on/off 感受性を遅らせる ノイズ除去フィルター としてはたらくことがわかっています。
アラビノースの制御系で用いられていることがわかりました。グルコースが無く、アラビノースだけが存在する状況で必要な遺伝子を産生する際に利用されます。
またインコヒーレントなフィードフォワードループは パルス生成器 として働きます。応答時間を加速しているのです。
|
|

