Aritalab:Lecture/JSBi/Test/Bio
Contents |
タンパク質の構造
二次構造
タンパク質をアミノ酸の鎖として捉えたときの情報を、二次構造情報という。 二次構造には大きく分けてαへリックス、βシート、ループ領域がある。
- αへリックス ...
- 平行βシート ...
- 逆平行βシート ...
- ループ
ペプチド結合
ペプチド鎖では、アミノ酸のカルボキシル基とアミノ基の間がペプチド結合でつながれている。この結合は共鳴によって二重結合性を帯び、平面構造をとる。 そのため、Cα炭素(両方の基が結合している炭素)どうしの間には炭素と窒素が1個ずつ入るのだが(結合数は3本)、Cα-N結合角(φ)とCα-C結合角は(ψ)の二つだけで立体配置が決定できる。この二つの角度を二面角と呼んでいる。
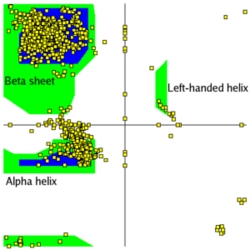
ラマチャンドラン マップ
|
ラマチャンドラン (Gopalasamudram Narayana Ramachandran) とは、インド出身の生物物理学者。 ペプチド鎖の特徴をみるのに、各アミノ酸のφ角度とψ角度を平面にプロットする方法を考え出した。 こうするとへリックスとシート構造がきれいに分離され、タンパク質の構造分類にも使えるので重宝されている。 |
|
コンタクトマップ
縦(上から下にN→C)横(左から右にN→C)にペプチド鎖を並べ、Cα原子間の距離が10オングストローム以内であれば色を塗る。 対角線上には同一の残基がくるので黒く塗られる。αへリックスはこの対角線上に重なる形で表示される。平行βシートは対角線から離れた位置に同じ角度で現れ、逆平行βシートは、対角線と直行する角度で現れる。
三次構造
タンパク質の立体構造は三次構造とも呼ばれる。正確な立体構造はX線結晶解析やNMRで決定するしかないが、進化の観点からは、構造の知られた既知配列に似る配列は、立体構造も似ていると仮定できる(ホモロジーモデリング)。
3D-1D法
タンパク質の立体構造を考える上で、20種のアミノ酸を区別する必然性は無い。そこで各アミノ酸に環境(極性か非極性か、タンパク質の内側か外側か)分類と二次構造のカテゴリーを割り当て、翻訳されたスコアに基づいて構造の類似性を判定する。
RMSD
三次元構造の重ね合わせ(構造アラインメント)の指標はRMSD (root mean square deviation) である。対応するCα原子間の距離の二乗和の平方根をとる。