Mol:PR100457
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | ACD/Labs08070915122D | + | ACD/Labs08070915122D |
| − | + | ||
| − | 42 46 0 0 1 0 0 0 0 0 2 V2000 | + | 42 46 0 0 1 0 0 0 0 0 2 V2000 |
| − | 19.8323 -8.2189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.8323 -8.2189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.8798 -6.3081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.8798 -6.3081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.8798 -7.2696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.8798 -7.2696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.7124 -7.7503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.7124 -7.7503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.5450 -7.2696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.5450 -7.2696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.5450 -6.3081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.5450 -6.3081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.7124 -5.8274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.7124 -5.8274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.3777 -7.7503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.3777 -7.7503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.2103 -7.2696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.2103 -7.2696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.2103 -6.3081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.2103 -6.3081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.3777 -5.8274 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 17.3777 -5.8274 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 19.0429 -5.8274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.0429 -5.8274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.8756 -6.3081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.8756 -6.3081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.7082 -5.8274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.7082 -5.8274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.7082 -4.8660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.7082 -4.8660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.8756 -4.3852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.8756 -4.3852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.0429 -4.8660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.0429 -4.8660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.7124 -8.7117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.7124 -8.7117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.8115 -5.8074 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.8115 -5.8074 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.6665 -4.4504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.6665 -4.4504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.8756 -3.4238 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.8756 -3.4238 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.3664 -7.2851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.3664 -7.2851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.3664 -8.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.3664 -8.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.1990 -8.7273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.1990 -8.7273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.0316 -8.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.0316 -8.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.0316 -7.2851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.0316 -7.2851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.1990 -6.8044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.1990 -6.8044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.1990 -5.8430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.1990 -5.8430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.0316 -5.3622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.0316 -5.3622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.8643 -6.8044 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.8643 -6.8044 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.2001 -10.4200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.2001 -10.4200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.8643 -8.7273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.8643 -8.7273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.4341 -10.9203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.4341 -10.9203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.4341 -11.8817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.4341 -11.8817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.2667 -12.3625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.2667 -12.3625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.0994 -11.8817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.0994 -11.8817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.0994 -10.9203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.0994 -10.9203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.2667 -10.4396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.2667 -10.4396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.2667 -9.4781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.2667 -9.4781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.4341 -9.9588 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.4341 -9.9588 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.9320 -12.3625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.9320 -12.3625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.9320 -10.4396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.9320 -10.4396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 2 0 0 0 0 | + | 6 7 2 0 0 0 0 |
| − | 7 2 1 0 0 0 0 | + | 7 2 1 0 0 0 0 |
| − | 5 8 1 0 0 0 0 | + | 5 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 6 1 0 0 0 0 | + | 11 6 1 0 0 0 0 |
| − | 10 12 1 0 0 0 0 | + | 10 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 4 18 1 0 0 0 0 | + | 4 18 1 0 0 0 0 |
| − | 2 19 1 0 0 0 0 | + | 2 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 1 9 1 0 0 0 0 | + | 1 9 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 6 0 0 0 | + | 27 28 1 6 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 26 30 1 1 0 0 0 | + | 26 30 1 1 0 0 0 |
| − | 24 31 1 1 0 0 0 | + | 24 31 1 1 0 0 0 |
| − | 25 32 1 6 0 0 0 | + | 25 32 1 6 0 0 0 |
| − | 23 1 1 6 0 0 0 | + | 23 1 1 6 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 38 39 1 6 0 0 0 | + | 38 39 1 6 0 0 0 |
| − | 33 40 1 6 0 0 0 | + | 33 40 1 6 0 0 0 |
| − | 36 41 1 6 0 0 0 | + | 36 41 1 6 0 0 0 |
| − | 37 42 1 1 0 0 0 | + | 37 42 1 1 0 0 0 |
| − | 31 33 1 0 0 0 0 | + | 31 33 1 0 0 0 0 |
| − | M CHG 1 11 1 | + | M CHG 1 11 1 |
| − | S SKP | + | S SKP 7 |
| − | CAS_RN 63525-17-1 | + | CAS_RN 63525-17-1 |
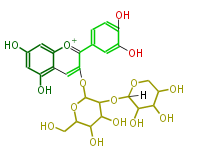
| − | NAME Cyanidin-3-O-(2''-O-beta-xylopyranosyl-beta-glucopyranoside) | + | NAME Cyanidin-3-O-(2''-O-beta-xylopyranosyl-beta-glucopyranoside) |
| + | ID PR100457 | ||
| + | FORMULA C26H29O15 | ||
| + | EXACTMASS 581.1506452579999 | ||
| + | AVERAGEMASS 581.49946 | ||
| + | SMILES C(C5O)(O)COC(C5O)(OC(C4O)C(OC(C4O)CO)Oc(c2)c([o+1]c(c3)c(c(O)cc3O)2)c(c1)ccc(O)c1O)[H] | ||
M END | M END | ||
Latest revision as of 16:44, 14 January 2011
ACD/Labs08070915122D 42 46 0 0 1 0 0 0 0 0 2 V2000 19.8323 -8.2189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 14.8798 -6.3081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 14.8798 -7.2696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 15.7124 -7.7503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 16.5450 -7.2696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 16.5450 -6.3081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 15.7124 -5.8274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.3777 -7.7503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 18.2103 -7.2696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 18.2103 -6.3081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.3777 -5.8274 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 19.0429 -5.8274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 19.8756 -6.3081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 20.7082 -5.8274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 20.7082 -4.8660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 19.8756 -4.3852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 19.0429 -4.8660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 15.7124 -8.7117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 13.8115 -5.8074 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 21.6665 -4.4504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 19.8756 -3.4238 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 21.3664 -7.2851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 21.3664 -8.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.1990 -8.7273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 23.0316 -8.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 23.0316 -7.2851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.1990 -6.8044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.1990 -5.8430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 23.0316 -5.3622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 23.8643 -6.8044 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 22.2001 -10.4200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 23.8643 -8.7273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 23.4341 -10.9203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 23.4341 -11.8817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 24.2667 -12.3625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 25.0994 -11.8817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 25.0994 -10.9203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 24.2667 -10.4396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 24.2667 -9.4781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 23.4341 -9.9588 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 25.9320 -12.3625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 25.9320 -10.4396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 2 3 2 0 0 0 0 3 4 1 0 0 0 0 4 5 2 0 0 0 0 5 6 1 0 0 0 0 6 7 2 0 0 0 0 7 2 1 0 0 0 0 5 8 1 0 0 0 0 8 9 2 0 0 0 0 9 10 1 0 0 0 0 10 11 2 0 0 0 0 11 6 1 0 0 0 0 10 12 1 0 0 0 0 12 13 2 0 0 0 0 13 14 1 0 0 0 0 14 15 2 0 0 0 0 15 16 1 0 0 0 0 16 17 2 0 0 0 0 17 12 1 0 0 0 0 4 18 1 0 0 0 0 2 19 1 0 0 0 0 15 20 1 0 0 0 0 1 9 1 0 0 0 0 16 21 1 0 0 0 0 22 23 1 0 0 0 0 23 24 1 0 0 0 0 24 25 1 0 0 0 0 25 26 1 0 0 0 0 26 27 1 0 0 0 0 27 22 1 0 0 0 0 27 28 1 6 0 0 0 28 29 1 0 0 0 0 26 30 1 1 0 0 0 24 31 1 1 0 0 0 25 32 1 6 0 0 0 23 1 1 6 0 0 0 33 34 1 0 0 0 0 34 35 1 0 0 0 0 35 36 1 0 0 0 0 36 37 1 0 0 0 0 37 38 1 0 0 0 0 38 33 1 0 0 0 0 38 39 1 6 0 0 0 33 40 1 6 0 0 0 36 41 1 6 0 0 0 37 42 1 1 0 0 0 31 33 1 0 0 0 0 M CHG 1 11 1 S SKP 7 CAS_RN 63525-17-1 NAME Cyanidin-3-O-(2''-O-beta-xylopyranosyl-beta-glucopyranoside) ID PR100457 FORMULA C26H29O15 EXACTMASS 581.1506452579999 AVERAGEMASS 581.49946 SMILES C(C5O)(O)COC(C5O)(OC(C4O)C(OC(C4O)CO)Oc(c2)c([o+1]c(c3)c(c(O)cc3O)2)c(c1)ccc(O)c1O)[H] M END