Mol:PR100345
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | ACD/Labs08070915132D | + | ACD/Labs08070915132D |
| − | + | ||
| − | 35 38 0 0 1 0 0 0 0 0 2 V2000 | + | 35 38 0 0 1 0 0 0 0 0 2 V2000 |
| − | 21.4826 -12.0739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.4826 -12.0739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.6499 -11.5932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.6499 -11.5932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.6499 -10.6317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.6499 -10.6317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.4826 -10.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.4826 -10.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.3152 -10.6317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.3152 -10.6317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.3152 -11.5932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.3152 -11.5932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.5031 -9.0136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.5031 -9.0136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.7051 -10.1510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.7051 -10.1510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.8507 -7.2695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.8507 -7.2695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.8507 -8.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.8507 -8.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.6833 -8.7117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.6833 -8.7117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.5160 -8.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.5160 -8.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.5160 -7.2695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.5160 -7.2695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.6833 -6.7888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.6833 -6.7888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.3486 -8.7117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.3486 -8.7117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.1812 -8.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.1812 -8.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.1812 -7.2695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.1812 -7.2695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.3486 -6.7888 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 19.3486 -6.7888 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 21.0139 -6.7888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.0139 -6.7888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.8465 -7.2695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.8465 -7.2695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.6791 -6.7888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.6791 -6.7888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.6791 -5.8274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.6791 -5.8274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.8465 -5.3466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.8465 -5.3466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.0139 -5.8274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.0139 -5.8274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.6833 -9.6732 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.6833 -9.6732 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.7824 -6.7688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.7824 -6.7688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.4826 -13.0353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.4826 -13.0353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.6375 -5.4118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.6375 -5.4118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.8173 -12.0739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.8173 -12.0739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.1478 -12.0739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.1478 -12.0739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.9805 -11.5932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.9805 -11.5932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.8465 -4.3852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.8465 -4.3852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.5118 -7.2695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.5118 -7.2695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.8789 -3.4170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.8789 -3.4170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.5118 -8.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.5118 -8.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 8 1 1 0 0 0 | + | 3 8 1 1 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 9 1 0 0 0 0 | + | 14 9 1 0 0 0 0 |
| − | 12 15 1 0 0 0 0 | + | 12 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 17 19 1 0 0 0 0 | + | 17 19 1 0 0 0 0 |
| − | 19 20 2 0 0 0 0 | + | 19 20 2 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 2 0 0 0 0 | + | 21 22 2 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 11 25 1 0 0 0 0 | + | 11 25 1 0 0 0 0 |
| − | 9 26 1 0 0 0 0 | + | 9 26 1 0 0 0 0 |
| − | 1 27 1 6 0 0 0 | + | 1 27 1 6 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 7 16 1 0 0 0 0 | + | 7 16 1 0 0 0 0 |
| − | 2 29 1 6 0 0 0 | + | 2 29 1 6 0 0 0 |
| − | 6 30 1 6 0 0 0 | + | 6 30 1 6 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 4 7 1 6 0 0 0 | + | 4 7 1 6 0 0 0 |
| − | 23 32 1 0 0 0 0 | + | 23 32 1 0 0 0 0 |
| − | 21 33 1 0 0 0 0 | + | 21 33 1 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | M CHG 1 18 1 | + | M CHG 1 18 1 |
| − | S SKP | + | S SKP 7 |
| − | CAS_RN 104880-34-4 | + | CAS_RN 104880-34-4 |
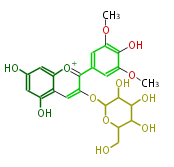
| − | NAME malvidin-3-galactoside | + | NAME malvidin-3-galactoside |
| + | ID PR100345 | ||
| + | FORMULA C23H25O12 | ||
| + | EXACTMASS 493.134601264 | ||
| + | AVERAGEMASS 493.4374 | ||
| + | SMILES OCC(O1)C(O)C(O)C(O)C1Oc(c3)c([o+1]c(c4)c(c(O)cc(O)4)3)c(c2)cc(OC)c(O)c(OC)2 | ||
M END | M END | ||
Latest revision as of 16:44, 14 January 2011
ACD/Labs08070915132D 35 38 0 0 1 0 0 0 0 0 2 V2000 21.4826 -12.0739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 20.6499 -11.5932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 20.6499 -10.6317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 21.4826 -10.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.3152 -10.6317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 22.3152 -11.5932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 21.5031 -9.0136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 19.7051 -10.1510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 16.8507 -7.2695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 16.8507 -8.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.6833 -8.7117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 18.5160 -8.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 18.5160 -7.2695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.6833 -6.7888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 19.3486 -8.7117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 20.1812 -8.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 20.1812 -7.2695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 19.3486 -6.7888 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 21.0139 -6.7888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 21.8465 -7.2695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.6791 -6.7888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.6791 -5.8274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 21.8465 -5.3466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 21.0139 -5.8274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.6833 -9.6732 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 15.7824 -6.7688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 21.4826 -13.0353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 23.6375 -5.4118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 19.8173 -12.0739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 23.1478 -12.0739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 23.9805 -11.5932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 21.8465 -4.3852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 23.5118 -7.2695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 22.8789 -3.4170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 23.5118 -8.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 1 2 1 0 0 0 0 2 3 1 0 0 0 0 3 4 1 0 0 0 0 4 5 1 0 0 0 0 5 6 1 0 0 0 0 6 1 1 0 0 0 0 3 8 1 1 0 0 0 9 10 2 0 0 0 0 10 11 1 0 0 0 0 11 12 2 0 0 0 0 12 13 1 0 0 0 0 13 14 2 0 0 0 0 14 9 1 0 0 0 0 12 15 1 0 0 0 0 15 16 2 0 0 0 0 16 17 1 0 0 0 0 17 18 2 0 0 0 0 18 13 1 0 0 0 0 17 19 1 0 0 0 0 19 20 2 0 0 0 0 20 21 1 0 0 0 0 21 22 2 0 0 0 0 22 23 1 0 0 0 0 23 24 2 0 0 0 0 24 19 1 0 0 0 0 11 25 1 0 0 0 0 9 26 1 0 0 0 0 1 27 1 6 0 0 0 22 28 1 0 0 0 0 7 16 1 0 0 0 0 2 29 1 6 0 0 0 6 30 1 6 0 0 0 30 31 1 0 0 0 0 4 7 1 6 0 0 0 23 32 1 0 0 0 0 21 33 1 0 0 0 0 32 34 1 0 0 0 0 33 35 1 0 0 0 0 M CHG 1 18 1 S SKP 7 CAS_RN 104880-34-4 NAME malvidin-3-galactoside ID PR100345 FORMULA C23H25O12 EXACTMASS 493.134601264 AVERAGEMASS 493.4374 SMILES OCC(O1)C(O)C(O)C(O)C1Oc(c3)c([o+1]c(c4)c(c(O)cc(O)4)3)c(c2)cc(OC)c(O)c(OC)2 M END