Mol:PR100342
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | ACD/Labs08070914182D | + | ACD/Labs08070914182D |
| − | + | ||
| − | 43 47 0 0 1 0 0 0 0 0 2 V2000 | + | 43 47 0 0 1 0 0 0 0 0 2 V2000 |
| − | 19.3717 -5.8737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.3717 -5.8737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.3752 -5.0496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.3752 -5.0496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.6614 -6.2875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.6614 -6.2875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.0855 -6.2909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.0855 -6.2909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.0924 -4.6392 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 20.0924 -4.6392 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 18.6614 -4.6358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.6614 -4.6358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.6648 -7.1082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.6648 -7.1082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.9476 -5.8737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.9476 -5.8737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.7993 -5.8806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.7993 -5.8806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.8028 -5.0565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.8028 -5.0565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.9476 -5.0496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.9476 -5.0496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.8304 -7.6909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.8304 -7.6909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.4545 -6.2634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.4545 -6.2634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.5200 -4.6461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.5200 -4.6461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.2338 -4.6358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.2338 -4.6358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.1166 -7.2806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.1166 -7.2806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.8304 -8.5151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.8304 -8.5151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.2269 -6.7185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.2269 -6.7185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.2304 -5.0634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.2304 -5.0634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.5200 -3.8220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.5200 -3.8220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.3993 -7.6909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.3993 -7.6909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.1166 -8.9289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.1166 -8.9289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.5442 -8.9289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.5442 -8.9289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.2166 -7.5392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.2166 -7.5392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.9442 -6.3116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.9442 -6.3116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.9476 -4.6530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.9476 -4.6530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.2373 -3.4151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.2373 -3.4151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.3993 -8.5151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.3993 -8.5151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.6855 -7.2771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.6855 -7.2771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.1166 -9.7530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.1166 -9.7530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.9269 -7.9599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.9269 -7.9599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.6545 -6.7323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.6545 -6.7323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.9545 -5.4875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.9545 -5.4875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.9511 -3.8289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.9511 -3.8289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.6579 -5.0703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.6579 -5.0703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.3811 -9.0892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.3811 -9.0892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.9717 -7.6909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.9717 -7.6909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.6442 -7.5599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.6442 -7.5599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.9166 -8.7875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.9166 -8.7875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.3752 -6.3289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.3752 -6.3289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.6683 -3.4185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.6683 -3.4185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.3545 -7.9806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.3545 -7.9806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.1959 -9.1909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.1959 -9.1909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 1 3 1 0 0 0 0 | + | 1 3 1 0 0 0 0 |
| − | 1 4 1 0 0 0 0 | + | 1 4 1 0 0 0 0 |
| − | 2 5 1 0 0 0 0 | + | 2 5 1 0 0 0 0 |
| − | 2 6 1 0 0 0 0 | + | 2 6 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 3 8 2 0 0 0 0 | + | 3 8 2 0 0 0 0 |
| − | 4 9 2 0 0 0 0 | + | 4 9 2 0 0 0 0 |
| − | 5 10 2 0 0 0 0 | + | 5 10 2 0 0 0 0 |
| − | 6 11 2 0 0 0 0 | + | 6 11 2 0 0 0 0 |
| − | 12 7 1 1 0 0 0 | + | 12 7 1 1 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 10 14 1 0 0 0 0 | + | 10 14 1 0 0 0 0 |
| − | 11 15 1 0 0 0 0 | + | 11 15 1 0 0 0 0 |
| − | 12 16 1 0 0 0 0 | + | 12 16 1 0 0 0 0 |
| − | 12 17 1 0 0 0 0 | + | 12 17 1 0 0 0 0 |
| − | 18 13 1 1 0 0 0 | + | 18 13 1 1 0 0 0 |
| − | 14 19 2 0 0 0 0 | + | 14 19 2 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 17 22 1 0 0 0 0 | + | 17 22 1 0 0 0 0 |
| − | 17 23 1 6 0 0 0 | + | 17 23 1 6 0 0 0 |
| − | 18 24 1 0 0 0 0 | + | 18 24 1 0 0 0 0 |
| − | 18 25 1 0 0 0 0 | + | 18 25 1 0 0 0 0 |
| − | 19 26 1 0 0 0 0 | + | 19 26 1 0 0 0 0 |
| − | 20 27 2 0 0 0 0 | + | 20 27 2 0 0 0 0 |
| − | 21 28 1 0 0 0 0 | + | 21 28 1 0 0 0 0 |
| − | 21 29 1 1 0 0 0 | + | 21 29 1 1 0 0 0 |
| − | 22 30 1 1 0 0 0 | + | 22 30 1 1 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 25 33 1 6 0 0 0 | + | 25 33 1 6 0 0 0 |
| − | 26 34 2 0 0 0 0 | + | 26 34 2 0 0 0 0 |
| − | 26 35 1 0 0 0 0 | + | 26 35 1 0 0 0 0 |
| − | 28 36 1 6 0 0 0 | + | 28 36 1 6 0 0 0 |
| − | 29 37 1 0 0 0 0 | + | 29 37 1 0 0 0 0 |
| − | 31 38 1 0 0 0 0 | + | 31 38 1 0 0 0 0 |
| − | 31 39 1 1 0 0 0 | + | 31 39 1 1 0 0 0 |
| − | 32 40 1 1 0 0 0 | + | 32 40 1 1 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 38 42 1 6 0 0 0 | + | 38 42 1 6 0 0 0 |
| − | 39 43 1 0 0 0 0 | + | 39 43 1 0 0 0 0 |
| − | 8 11 1 0 0 0 0 | + | 8 11 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 34 1 0 0 0 0 | + | 27 34 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | M CHG 1 5 1 | + | M CHG 1 5 1 |
| − | S SKP | + | S SKP 7 |
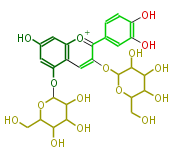
| − | CAS_RN 20905-74-2 | + | CAS_RN 20905-74-2 |
| − | NAME cyanidin-3,5-di-O-glucoside | + | NAME cyanidin-3,5-di-O-glucoside |
| + | ID PR100342 | ||
| + | FORMULA C27H31O16 | ||
| + | EXACTMASS 611.161209944 | ||
| + | AVERAGEMASS 611.52544 | ||
| + | SMILES c(c(c([o+1]2)c(OC(O5)C(C(C(C5CO)O)O)O)cc(c3OC(O4)C(C(C(C4CO)O)O)O)c(cc(c3)O)2)1)c(O)c(O)cc1 | ||
M END | M END | ||
Latest revision as of 16:44, 14 January 2011
ACD/Labs08070914182D 43 47 0 0 1 0 0 0 0 0 2 V2000 19.3717 -5.8737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 19.3752 -5.0496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 18.6614 -6.2875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 20.0855 -6.2909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 20.0924 -4.6392 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 18.6614 -4.6358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 18.6648 -7.1082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 17.9476 -5.8737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 20.7993 -5.8806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 20.8028 -5.0565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.9476 -5.0496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.8304 -7.6909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 21.4545 -6.2634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 21.5200 -4.6461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.2338 -4.6358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 17.1166 -7.2806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 17.8304 -8.5151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.2269 -6.7185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.2304 -5.0634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 21.5200 -3.8220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 16.3993 -7.6909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.1166 -8.9289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 18.5442 -8.9289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 22.2166 -7.5392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 22.9442 -6.3116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.9476 -4.6530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.2373 -3.4151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 16.3993 -8.5151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 15.6855 -7.2771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.1166 -9.7530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 22.9269 -7.9599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 23.6545 -6.7323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.9545 -5.4875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 22.9511 -3.8289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 23.6579 -5.0703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 15.3811 -9.0892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 14.9717 -7.6909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 23.6442 -7.5599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.9166 -8.7875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 24.3752 -6.3289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 23.6683 -3.4185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 24.3545 -7.9806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 22.1959 -9.1909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 1 2 2 0 0 0 0 1 3 1 0 0 0 0 1 4 1 0 0 0 0 2 5 1 0 0 0 0 2 6 1 0 0 0 0 3 7 1 0 0 0 0 3 8 2 0 0 0 0 4 9 2 0 0 0 0 5 10 2 0 0 0 0 6 11 2 0 0 0 0 12 7 1 1 0 0 0 9 13 1 0 0 0 0 10 14 1 0 0 0 0 11 15 1 0 0 0 0 12 16 1 0 0 0 0 12 17 1 0 0 0 0 18 13 1 1 0 0 0 14 19 2 0 0 0 0 14 20 1 0 0 0 0 16 21 1 0 0 0 0 17 22 1 0 0 0 0 17 23 1 6 0 0 0 18 24 1 0 0 0 0 18 25 1 0 0 0 0 19 26 1 0 0 0 0 20 27 2 0 0 0 0 21 28 1 0 0 0 0 21 29 1 1 0 0 0 22 30 1 1 0 0 0 24 31 1 0 0 0 0 25 32 1 0 0 0 0 25 33 1 6 0 0 0 26 34 2 0 0 0 0 26 35 1 0 0 0 0 28 36 1 6 0 0 0 29 37 1 0 0 0 0 31 38 1 0 0 0 0 31 39 1 1 0 0 0 32 40 1 1 0 0 0 34 41 1 0 0 0 0 38 42 1 6 0 0 0 39 43 1 0 0 0 0 8 11 1 0 0 0 0 9 10 1 0 0 0 0 22 28 1 0 0 0 0 27 34 1 0 0 0 0 32 38 1 0 0 0 0 M CHG 1 5 1 S SKP 7 CAS_RN 20905-74-2 NAME cyanidin-3,5-di-O-glucoside ID PR100342 FORMULA C27H31O16 EXACTMASS 611.161209944 AVERAGEMASS 611.52544 SMILES c(c(c([o+1]2)c(OC(O5)C(C(C(C5CO)O)O)O)cc(c3OC(O4)C(C(C(C4CO)O)O)O)c(cc(c3)O)2)1)c(O)c(O)cc1 M END