Mol:LBPR01070158
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 45 0 0 0 0 0 0 0 0999 V2000 | + | 44 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -6.5070 -0.9191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.5070 -0.9191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7837 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7837 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0604 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0604 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3371 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3371 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6137 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6137 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8904 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8904 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1671 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1671 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4438 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4438 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7204 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7204 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0029 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0029 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7262 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7262 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4496 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4496 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1729 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1729 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8962 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8962 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6195 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6195 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3429 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3429 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0662 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0662 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7895 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7895 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5129 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5129 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0604 1.5791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0604 1.5791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1671 1.5791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1671 1.5791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4496 -0.5125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4496 -0.5125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3429 -0.5125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3429 -0.5125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.2246 -0.5066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.2246 -0.5066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.9421 -0.9191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.9421 -0.9191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.9421 -1.7471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.9421 -1.7471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.2246 -2.1625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.2246 -2.1625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.5070 -1.7471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.5070 -1.7471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2304 0.3300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2304 0.3300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9421 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9421 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9421 1.5646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9421 1.5646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2304 1.9742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2304 1.9742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5129 1.5646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5129 1.5646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7982 1.9771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7982 1.9771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7924 -2.1596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7924 -2.1596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.9392 -0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.9392 -0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9450 -0.0825 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9450 -0.0825 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.5099 -0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.5099 -0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5158 -0.0825 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5158 -0.0825 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -8.6567 -2.1596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -8.6567 -2.1596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.6567 1.9771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.6567 1.9771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7837 -0.5008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7837 -0.5008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.4983 0.7367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.4983 0.7367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4770 2.1625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4770 2.1625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 7 21 1 0 0 0 0 | + | 7 21 1 0 0 0 0 |
| − | 12 22 1 0 0 0 0 | + | 12 22 1 0 0 0 0 |
| − | 16 23 1 0 0 0 0 | + | 16 23 1 0 0 0 0 |
| − | 24 1 1 0 0 0 0 | + | 24 1 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 1 2 0 0 0 0 | + | 28 1 2 0 0 0 0 |
| − | 29 19 1 0 0 0 0 | + | 29 19 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 19 33 1 0 0 0 0 | + | 19 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 28 35 1 0 0 0 0 | + | 28 35 1 0 0 0 0 |
| − | 24 36 1 1 0 0 0 | + | 24 36 1 1 0 0 0 |
| − | 29 37 1 1 0 0 0 | + | 29 37 1 1 0 0 0 |
| − | 24 38 1 6 0 0 0 | + | 24 38 1 6 0 0 0 |
| − | 29 39 1 6 0 0 0 | + | 29 39 1 6 0 0 0 |
| − | 19 18 1 1 0 0 0 | + | 19 18 1 1 0 0 0 |
| − | 26 40 1 1 0 0 0 | + | 26 40 1 1 0 0 0 |
| − | 31 41 1 6 0 0 0 | + | 31 41 1 6 0 0 0 |
| − | 2 42 1 0 0 0 0 | + | 2 42 1 0 0 0 0 |
| − | 1 42 1 0 0 0 0 | + | 1 42 1 0 0 0 0 |
| − | 43 2 2 0 0 0 0 | + | 43 2 2 0 0 0 0 |
| − | 20 44 1 0 0 0 0 | + | 20 44 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID LBPR01070158 | + | ID LBPR01070158 |
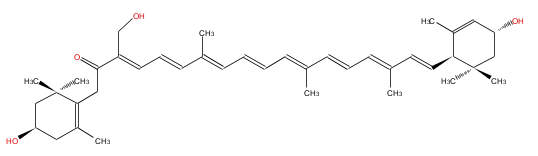
| − | FORMULA C40H56O4 | + | FORMULA C40H56O4 |
| − | EXACTMASS 600.41786028 | + | EXACTMASS 600.41786028 |
| − | AVERAGEMASS 600.8702400000001 | + | AVERAGEMASS 600.8702400000001 |
| − | SMILES CC(C)(C1)C(CC(=O)C(=CC=CC(C)=CC=CC=C(C=CC=C(C)C=CC(C2(C)C)C(=CC(C2)O)C)C)CO)=C(CC(O)1)C | + | SMILES CC(C)(C1)C(CC(=O)C(=CC=CC(C)=CC=CC=C(C=CC=C(C)C=CC(C2(C)C)C(=CC(C2)O)C)C)CO)=C(CC(O)1)C |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 45 0 0 0 0 0 0 0 0999 V2000
-6.5070 -0.9191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7837 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0604 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3371 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6137 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8904 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1671 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4438 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7204 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0029 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7262 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4496 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1729 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8962 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6195 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3429 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0662 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7895 0.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5129 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0604 1.5791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1671 1.5791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4496 -0.5125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3429 -0.5125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.2246 -0.5066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.9421 -0.9191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.9421 -1.7471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.2246 -2.1625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.5070 -1.7471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.2304 0.3300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.9421 0.7425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.9421 1.5646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.2304 1.9742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5129 1.5646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7982 1.9771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7924 -2.1596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.9392 -0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.9450 -0.0825 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.5099 -0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5158 -0.0825 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-8.6567 -2.1596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.6567 1.9771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7837 -0.5008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.4983 0.7367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4770 2.1625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
3 20 1 0 0 0 0
7 21 1 0 0 0 0
12 22 1 0 0 0 0
16 23 1 0 0 0 0
24 1 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 1 2 0 0 0 0
29 19 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
19 33 1 0 0 0 0
33 34 1 0 0 0 0
28 35 1 0 0 0 0
24 36 1 1 0 0 0
29 37 1 1 0 0 0
24 38 1 6 0 0 0
29 39 1 6 0 0 0
19 18 1 1 0 0 0
26 40 1 1 0 0 0
31 41 1 6 0 0 0
2 42 1 0 0 0 0
1 42 1 0 0 0 0
43 2 2 0 0 0 0
20 44 1 0 0 0 0
S SKP 5
ID LBPR01070158
FORMULA C40H56O4
EXACTMASS 600.41786028
AVERAGEMASS 600.8702400000001
SMILES CC(C)(C1)C(CC(=O)C(=CC=CC(C)=CC=CC=C(C=CC=C(C)C=CC(C2(C)C)C(=CC(C2)O)C)C)CO)=C(CC(O)1)C
M END