Mol:FLICALNI0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 31 33 0 0 0 0 0 0 0 0999 V2000 | + | 31 33 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4499 1.7168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4499 1.7168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4499 1.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4499 1.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8936 0.7532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8936 0.7532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3373 1.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3373 1.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3373 1.7168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3373 1.7168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8936 2.0380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8936 2.0380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2190 0.7532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2190 0.7532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7753 1.0744 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.7753 1.0744 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.7753 1.7168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7753 1.7168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2190 2.0380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2190 2.0380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3314 0.7534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3314 0.7534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3314 0.0666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3314 0.0666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9262 -0.2768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9262 -0.2768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5210 0.0666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5210 0.0666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5210 0.7534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5210 0.7534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9262 1.0968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9262 1.0968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7372 -0.2765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7372 -0.2765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9262 -0.9636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9262 -0.9636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5210 -1.3070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5210 -1.3070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1158 -0.2768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1158 -0.2768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5210 -1.9932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5210 -1.9932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1140 -2.3355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1140 -2.3355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9280 -2.3355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9280 -2.3355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0060 0.7534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0060 0.7534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0060 0.1126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0060 0.1126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5609 -0.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5609 -0.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5609 -0.8485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5609 -0.8485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1158 0.1126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1158 0.1126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8936 0.1111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8936 0.1111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8071 2.3355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8071 2.3355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3071 3.2015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3071 3.2015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 8 11 1 0 0 0 0 | + | 8 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 12 17 1 0 0 0 0 | + | 12 17 1 0 0 0 0 |
| − | 13 18 1 0 0 0 0 | + | 13 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 20 14 1 0 0 0 0 | + | 20 14 1 0 0 0 0 |
| − | 19 21 1 0 0 0 0 | + | 19 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 21 23 1 0 0 0 0 | + | 21 23 1 0 0 0 0 |
| − | 2 24 1 0 0 0 0 | + | 2 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 1 30 1 0 0 0 0 | + | 1 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 30 31 | + | M SAL 1 2 30 31 |
| − | M SBL 1 1 32 | + | M SBL 1 1 32 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 32 -1.8071 2.3355 | + | M SVB 1 32 -1.8071 2.3355 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLICALNI0001 | + | ID FLICALNI0001 |
| − | KNApSAcK_ID C00009739 | + | KNApSAcK_ID C00009739 |
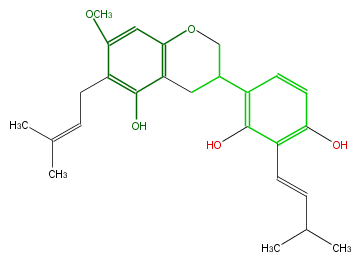
| − | NAME Licoricidin;5,2',4'-Trihydroxy-7-methoxy-6,3'-diprenylisoflavan | + | NAME Licoricidin;5,2',4'-Trihydroxy-7-methoxy-6,3'-diprenylisoflavan |
| − | CAS_RN 30508-27-1 | + | CAS_RN 30508-27-1 |
| − | FORMULA C26H32O5 | + | FORMULA C26H32O5 |
| − | EXACTMASS 424.224974134 | + | EXACTMASS 424.224974134 |
| − | AVERAGEMASS 424.52927999999997 | + | AVERAGEMASS 424.52927999999997 |
| − | SMILES C(C)(C)=CCc(c3OC)c(O)c(C1)c(c3)OCC(c(c2)c(c(c(c2)O)C=CC(C)C)O)1 | + | SMILES C(C)(C)=CCc(c3OC)c(O)c(C1)c(c3)OCC(c(c2)c(c(c(c2)O)C=CC(C)C)O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
31 33 0 0 0 0 0 0 0 0999 V2000
-1.4499 1.7168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4499 1.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8936 0.7532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3373 1.0744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3373 1.7168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8936 2.0380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2190 0.7532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7753 1.0744 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.7753 1.7168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2190 2.0380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3314 0.7534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3314 0.0666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9262 -0.2768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5210 0.0666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5210 0.7534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9262 1.0968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7372 -0.2765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9262 -0.9636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5210 -1.3070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1158 -0.2768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5210 -1.9932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1140 -2.3355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9280 -2.3355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0060 0.7534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0060 0.1126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5609 -0.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5609 -0.8485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1158 0.1126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8936 0.1111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8071 2.3355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3071 3.2015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
8 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 2 1 0 0 0 0
12 17 1 0 0 0 0
13 18 1 0 0 0 0
18 19 2 0 0 0 0
20 14 1 0 0 0 0
19 21 1 0 0 0 0
21 22 1 0 0 0 0
21 23 1 0 0 0 0
2 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
26 28 1 0 0 0 0
3 29 1 0 0 0 0
1 30 1 0 0 0 0
30 31 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 30 31
M SBL 1 1 32
M SMT 1 OCH3
M SVB 1 32 -1.8071 2.3355
S SKP 8
ID FLICALNI0001
KNApSAcK_ID C00009739
NAME Licoricidin;5,2',4'-Trihydroxy-7-methoxy-6,3'-diprenylisoflavan
CAS_RN 30508-27-1
FORMULA C26H32O5
EXACTMASS 424.224974134
AVERAGEMASS 424.52927999999997
SMILES C(C)(C)=CCc(c3OC)c(O)c(C1)c(c3)OCC(c(c2)c(c(c(c2)O)C=CC(C)C)O)1
M END