Mol:FLIAECGS0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 46 50 0 0 0 0 0 0 0 0999 V2000 | + | 46 50 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.2631 0.5016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2631 0.5016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2932 0.1804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2932 0.1804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8495 0.5016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8495 0.5016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4056 0.1806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4056 0.1806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4056 -0.4854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4056 -0.4854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9823 -0.8184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9823 -0.8184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5590 -0.4854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5590 -0.4854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5590 0.1806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5590 0.1806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9823 0.5135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9823 0.5135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2934 -0.4616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2934 -0.4616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8495 -0.7826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8495 -0.7826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9823 -1.4839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9823 -1.4839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1354 -0.8181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1354 -0.8181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1354 -1.4548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1354 -1.4548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6867 -1.7731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6867 -1.7731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2381 -1.4548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2381 -1.4548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2381 -0.8181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2381 -0.8181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6867 -0.4998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6867 -0.4998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3670 0.2979 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.3670 0.2979 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.0208 -0.1592 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.0208 -0.1592 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.5222 0.0347 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.5222 0.0347 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.0025 0.0290 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.0025 0.0290 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.3907 0.3896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3907 0.3896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9000 0.2067 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -1.9000 0.2067 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.8122 0.3369 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8122 0.3369 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1352 -0.6872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1352 -0.6872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2365 -0.4449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2365 -0.4449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2618 0.8334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2618 0.8334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7595 1.2879 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -4.7595 1.2879 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -4.4133 0.8308 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -4.4133 0.8308 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.9147 1.0247 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.9147 1.0247 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.3950 1.0190 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.3950 1.0190 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.7832 1.3796 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7832 1.3796 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2925 1.1967 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -4.2925 1.1967 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -5.1465 1.5114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1465 1.5114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7401 0.4006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7401 0.4006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6290 0.5451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6290 0.5451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7473 1.5359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7473 1.5359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7894 -0.4998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7894 -0.4998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8390 -1.4233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8390 -1.4233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4211 -0.3695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4211 -0.3695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4211 -1.1945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4211 -1.1945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1041 -1.9548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1041 -1.9548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9701 -2.4548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9701 -2.4548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4920 2.0735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4920 2.0735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4474 2.3689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4474 2.3689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 4 1 0 0 0 0 | + | 9 4 1 0 0 0 0 |
| − | 2 10 1 0 0 0 0 | + | 2 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 5 1 0 0 0 0 | + | 11 5 1 0 0 0 0 |
| − | 6 12 2 0 0 0 0 | + | 6 12 2 0 0 0 0 |
| − | 7 13 1 0 0 0 0 | + | 7 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 1 1 0 0 0 0 | + | 22 1 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 28 38 1 0 0 0 0 | + | 28 38 1 0 0 0 0 |
| − | 38 32 1 0 0 0 0 | + | 38 32 1 0 0 0 0 |
| − | 17 39 1 0 0 0 0 | + | 17 39 1 0 0 0 0 |
| − | 11 40 1 0 0 0 0 | + | 11 40 1 0 0 0 0 |
| − | 10 41 1 0 0 0 0 | + | 10 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 16 43 1 0 0 0 0 | + | 16 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 34 45 1 0 0 0 0 | + | 34 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 45 46 | + | M SAL 3 2 45 46 |
| − | M SBL 3 1 49 | + | M SBL 3 1 49 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 49 -4.492 2.0735 | + | M SVB 3 49 -4.492 2.0735 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 43 44 | + | M SAL 2 2 43 44 |
| − | M SBL 2 1 47 | + | M SBL 2 1 47 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 47 4.4322 -1.661 | + | M SVB 2 47 4.4322 -1.661 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 45 -0.4211 -0.3695 | + | M SVB 1 45 -0.4211 -0.3695 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLIAECGS0005 | + | ID FLIAECGS0005 |
| − | KNApSAcK_ID C00010148 | + | KNApSAcK_ID C00010148 |
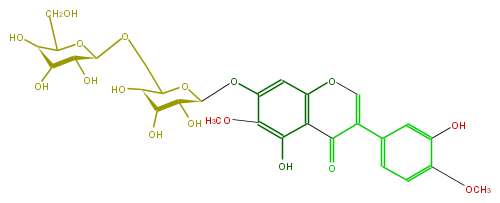
| − | NAME Iristectorigenin A 7-O-gentiobioside | + | NAME Iristectorigenin A 7-O-gentiobioside |
| − | CAS_RN 86849-71-0 | + | CAS_RN 86849-71-0 |
| − | FORMULA C29H34O17 | + | FORMULA C29H34O17 |
| − | EXACTMASS 654.179599662 | + | EXACTMASS 654.179599662 |
| − | AVERAGEMASS 654.57006 | + | AVERAGEMASS 654.57006 |
| − | SMILES [C@@H](OCC([C@@H]2O)O[C@@H](Oc(c5OC)cc(c3c5O)OC=C(c(c4)cc(c(c4)OC)O)C(=O)3)[C@@H](O)[C@@H](O)2)([C@@H](O)1)OC(CO)[C@H](O)[C@@H]1O | + | SMILES [C@@H](OCC([C@@H]2O)O[C@@H](Oc(c5OC)cc(c3c5O)OC=C(c(c4)cc(c(c4)OC)O)C(=O)3)[C@@H](O)[C@@H](O)2)([C@@H](O)1)OC(CO)[C@H](O)[C@@H]1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
46 50 0 0 0 0 0 0 0 0999 V2000
-0.2631 0.5016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2932 0.1804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8495 0.5016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4056 0.1806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4056 -0.4854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9823 -0.8184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5590 -0.4854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5590 0.1806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9823 0.5135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2934 -0.4616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8495 -0.7826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9823 -1.4839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1354 -0.8181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1354 -1.4548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6867 -1.7731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2381 -1.4548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2381 -0.8181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6867 -0.4998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3670 0.2979 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.0208 -0.1592 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.5222 0.0347 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.0025 0.0290 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.3907 0.3896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9000 0.2067 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.8122 0.3369 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1352 -0.6872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2365 -0.4449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2618 0.8334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7595 1.2879 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-4.4133 0.8308 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.9147 1.0247 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.3950 1.0190 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.7832 1.3796 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2925 1.1967 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-5.1465 1.5114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7401 0.4006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6290 0.5451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7473 1.5359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7894 -0.4998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8390 -1.4233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4211 -0.3695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4211 -1.1945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1041 -1.9548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.9701 -2.4548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4920 2.0735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4474 2.3689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 4 1 0 0 0 0
2 10 1 0 0 0 0
10 11 2 0 0 0 0
11 5 1 0 0 0 0
6 12 2 0 0 0 0
7 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 1 1 0 0 0 0
24 28 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
29 35 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
28 38 1 0 0 0 0
38 32 1 0 0 0 0
17 39 1 0 0 0 0
11 40 1 0 0 0 0
10 41 1 0 0 0 0
41 42 1 0 0 0 0
16 43 1 0 0 0 0
43 44 1 0 0 0 0
34 45 1 0 0 0 0
45 46 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 45 46
M SBL 3 1 49
M SMT 3 CH2OH
M SVB 3 49 -4.492 2.0735
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 43 44
M SBL 2 1 47
M SMT 2 OCH3
M SVB 2 47 4.4322 -1.661
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 45
M SMT 1 OCH3
M SVB 1 45 -0.4211 -0.3695
S SKP 8
ID FLIAECGS0005
KNApSAcK_ID C00010148
NAME Iristectorigenin A 7-O-gentiobioside
CAS_RN 86849-71-0
FORMULA C29H34O17
EXACTMASS 654.179599662
AVERAGEMASS 654.57006
SMILES [C@@H](OCC([C@@H]2O)O[C@@H](Oc(c5OC)cc(c3c5O)OC=C(c(c4)cc(c(c4)OC)O)C(=O)3)[C@@H](O)[C@@H](O)2)([C@@H](O)1)OC(CO)[C@H](O)[C@@H]1O
M END