Mol:FLIAAANF0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 31 34 0 0 0 0 0 0 0 0999 V2000 | + | 31 34 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.0677 -0.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0677 -0.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0677 -0.7705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0677 -0.7705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5114 -1.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5114 -1.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0449 -0.7705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0449 -0.7705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0449 -0.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0449 -0.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5114 0.1930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5114 0.1930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6012 -1.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6012 -1.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1575 -0.7705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1575 -0.7705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1575 -0.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1575 -0.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6012 0.1930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6012 0.1930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7136 -1.0916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7136 -1.0916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7136 -1.7784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7136 -1.7784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3084 -2.1218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3084 -2.1218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9032 -1.7784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9032 -1.7784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9032 -1.0916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9032 -1.0916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3084 -0.7482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3084 -0.7482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6012 -1.7338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6012 -1.7338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5114 -1.7338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5114 -1.7338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5114 0.8206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5114 0.8206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0753 1.1462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0753 1.1462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0753 1.7966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0753 1.7966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5121 2.1218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5121 2.1218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6386 2.1218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6386 2.1218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6786 0.0704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6786 0.0704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0562 -0.4493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0562 -0.4493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6786 -0.9690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6786 -0.9690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6983 -0.4493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6983 -0.4493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0194 0.1068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0194 0.1068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0194 -1.0054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0194 -1.0054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4974 -0.4493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4974 -0.4493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4974 -2.1214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4974 -2.1214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 8 11 1 0 0 0 0 | + | 8 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 11 2 0 0 0 0 | + | 16 11 2 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 6 19 1 0 0 0 0 | + | 6 19 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 2 0 0 0 0 | + | 20 21 2 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 21 23 1 0 0 0 0 | + | 21 23 1 0 0 0 0 |
| − | 1 24 1 0 0 0 0 | + | 1 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 2 1 0 0 0 0 | + | 26 2 1 0 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 14 31 1 0 0 0 0 | + | 14 31 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLIAAANF0005 | + | ID FLIAAANF0005 |
| − | KNApSAcK_ID C00009945 | + | KNApSAcK_ID C00009945 |
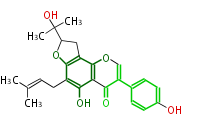
| − | NAME Senegalensin | + | NAME Senegalensin |
| − | CAS_RN 139051-61-9 | + | CAS_RN 139051-61-9 |
| − | FORMULA C25H26O6 | + | FORMULA C25H26O6 |
| − | EXACTMASS 422.172938564 | + | EXACTMASS 422.172938564 |
| − | AVERAGEMASS 422.47033999999996 | + | AVERAGEMASS 422.47033999999996 |
| − | SMILES O(C4C(C)(C)O)c(c(CC=C(C)C)1)c(C4)c(c(C2=O)c1OC=C(c(c3)ccc(c3)O)2)O | + | SMILES O(C4C(C)(C)O)c(c(CC=C(C)C)1)c(C4)c(c(C2=O)c1OC=C(c(c3)ccc(c3)O)2)O |
M END | M END | ||
| − | |||
Revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
31 34 0 0 0 0 0 0 0 0999 V2000
-1.0677 -0.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0677 -0.7705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5114 -1.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0449 -0.7705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0449 -0.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5114 0.1930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6012 -1.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1575 -0.7705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1575 -0.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6012 0.1930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7136 -1.0916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7136 -1.7784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3084 -2.1218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9032 -1.7784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9032 -1.0916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3084 -0.7482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6012 -1.7338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5114 -1.7338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5114 0.8206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0753 1.1462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0753 1.7966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5121 2.1218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6386 2.1218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6786 0.0704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0562 -0.4493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6786 -0.9690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6983 -0.4493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0194 0.1068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0194 -1.0054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4974 -0.4493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4974 -2.1214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
8 11 1 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 11 2 0 0 0 0
1 2 1 0 0 0 0
7 17 2 0 0 0 0
3 18 1 0 0 0 0
6 19 1 0 0 0 0
19 20 1 0 0 0 0
20 21 2 0 0 0 0
21 22 1 0 0 0 0
21 23 1 0 0 0 0
1 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 2 1 0 0 0 0
25 27 1 0 0 0 0
27 28 1 0 0 0 0
27 29 1 0 0 0 0
27 30 1 0 0 0 0
14 31 1 0 0 0 0
S SKP 8
ID FLIAAANF0005
KNApSAcK_ID C00009945
NAME Senegalensin
CAS_RN 139051-61-9
FORMULA C25H26O6
EXACTMASS 422.172938564
AVERAGEMASS 422.47033999999996
SMILES O(C4C(C)(C)O)c(c(CC=C(C)C)1)c(C4)c(c(C2=O)c1OC=C(c(c3)ccc(c3)O)2)O
M END