Mol:FL7ARXGL0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 50 0 0 0 0 0 0 0 0999 V2000 | + | 45 50 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.3533 -1.1365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3533 -1.1365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6496 -0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6496 -0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6496 0.0823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6496 0.0823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3533 0.4886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3533 0.4886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0569 0.0823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0569 0.0823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0569 -0.7302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0569 -0.7302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3533 1.3011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3533 1.3011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0569 1.7074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0569 1.7074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7606 1.3011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7606 1.3011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7606 0.4886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7606 0.4886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0541 0.4886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0541 0.4886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0541 1.3011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0541 1.3011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6496 1.7074 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -0.6496 1.7074 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8374 1.7533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8374 1.7533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5275 1.3549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5275 1.3549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2176 1.7533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2176 1.7533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2176 2.5502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2176 2.5502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5275 2.9486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5275 2.9486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8374 2.5502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8374 2.5502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3337 1.6320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3337 1.6320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3533 -1.7808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3533 -1.7808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6927 0.1670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6927 0.1670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5275 3.4835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5275 3.4835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9855 3.9415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9855 3.9415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6259 2.7859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6259 2.7859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6733 1.4902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6733 1.4902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3337 1.4902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3337 1.4902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0441 -2.1797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0441 -2.1797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0441 -2.9775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0441 -2.9775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3533 -3.3763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3533 -3.3763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6624 -2.9775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6624 -2.9775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6624 -2.1797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6624 -2.1797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3533 -3.9415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3533 -3.9415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2451 -3.3948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2451 -3.3948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4684 -2.1886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4684 -2.1886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1977 -1.8029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1977 -1.8029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0185 -0.9976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0185 -0.9976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2746 -0.2133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2746 -0.2133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5452 -0.5989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5452 -0.5989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7244 -1.4043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7244 -1.4043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3046 -1.5002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3046 -1.5002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0916 -1.8394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0916 -1.8394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9507 -2.7081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9507 -2.7081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4968 -1.2980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4968 -1.2980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0276 -2.3550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0276 -2.3550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 3 11 2 0 0 0 0 | + | 3 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 7 1 0 0 0 0 | + | 13 7 1 0 0 0 0 |
| − | 12 14 1 0 0 0 0 | + | 12 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 9 20 1 0 0 0 0 | + | 9 20 1 0 0 0 0 |
| − | 1 21 1 0 0 0 0 | + | 1 21 1 0 0 0 0 |
| − | 11 22 1 0 0 0 0 | + | 11 22 1 0 0 0 0 |
| − | 18 23 1 0 0 0 0 | + | 18 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 17 25 1 0 0 0 0 | + | 17 25 1 0 0 0 0 |
| − | 16 26 1 0 0 0 0 | + | 16 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 21 28 2 0 0 0 0 | + | 21 28 2 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 2 0 0 0 0 | + | 29 30 2 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 32 21 1 0 0 0 0 | + | 32 21 1 0 0 0 0 |
| − | 30 33 1 0 0 0 0 | + | 30 33 1 0 0 0 0 |
| − | 31 34 1 0 0 0 0 | + | 31 34 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 37 44 1 0 0 0 0 | + | 37 44 1 0 0 0 0 |
| − | 36 45 1 0 0 0 0 | + | 36 45 1 0 0 0 0 |
| − | 22 38 1 0 0 0 0 | + | 22 38 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7ARXGL0005 | + | ID FL7ARXGL0005 |
| − | KNApSAcK_ID C00011188 | + | KNApSAcK_ID C00011188 |
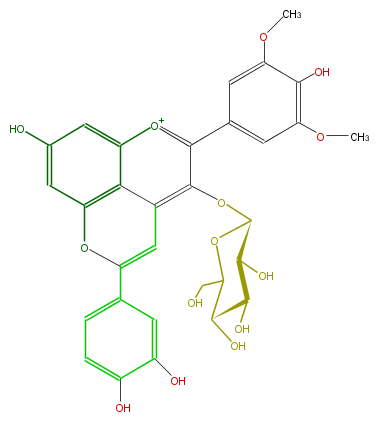
| − | NAME Malvidin 3-glucoside-4-vinylcatechol | + | NAME Malvidin 3-glucoside-4-vinylcatechol |
| − | CAS_RN 403982-47-8 | + | CAS_RN 403982-47-8 |
| − | FORMULA C31H29O14 | + | FORMULA C31H29O14 |
| − | EXACTMASS 625.155730636 | + | EXACTMASS 625.155730636 |
| − | AVERAGEMASS 625.5535600000001 | + | AVERAGEMASS 625.5535600000001 |
| − | SMILES C(C6O)(C(OC(C6O)Oc(c4c(c5)cc(OC)c(O)c(OC)5)c(C=2)c(c1[o+1]4)c(OC2c(c3)ccc(O)c3O)cc(c1)O)CO)O | + | SMILES C(C6O)(C(OC(C6O)Oc(c4c(c5)cc(OC)c(O)c(OC)5)c(C=2)c(c1[o+1]4)c(OC2c(c3)ccc(O)c3O)cc(c1)O)CO)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 50 0 0 0 0 0 0 0 0999 V2000
-1.3533 -1.1365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6496 -0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6496 0.0823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3533 0.4886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0569 0.0823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0569 -0.7302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3533 1.3011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0569 1.7074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7606 1.3011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7606 0.4886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0541 0.4886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0541 1.3011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6496 1.7074 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.8374 1.7533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5275 1.3549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2176 1.7533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2176 2.5502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5275 2.9486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8374 2.5502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3337 1.6320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3533 -1.7808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6927 0.1670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5275 3.4835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9855 3.9415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6259 2.7859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6733 1.4902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3337 1.4902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0441 -2.1797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0441 -2.9775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3533 -3.3763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6624 -2.9775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6624 -2.1797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3533 -3.9415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2451 -3.3948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4684 -2.1886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1977 -1.8029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0185 -0.9976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2746 -0.2133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5452 -0.5989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7244 -1.4043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3046 -1.5002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0916 -1.8394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9507 -2.7081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4968 -1.2980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0276 -2.3550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
3 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 7 1 0 0 0 0
12 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 14 1 0 0 0 0
9 20 1 0 0 0 0
1 21 1 0 0 0 0
11 22 1 0 0 0 0
18 23 1 0 0 0 0
23 24 1 0 0 0 0
17 25 1 0 0 0 0
16 26 1 0 0 0 0
26 27 1 0 0 0 0
21 28 2 0 0 0 0
28 29 1 0 0 0 0
29 30 2 0 0 0 0
30 31 1 0 0 0 0
31 32 2 0 0 0 0
32 21 1 0 0 0 0
30 33 1 0 0 0 0
31 34 1 0 0 0 0
35 36 1 1 0 0 0
36 37 1 1 0 0 0
38 37 1 1 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
40 35 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 0 0 0 0
35 43 1 0 0 0 0
37 44 1 0 0 0 0
36 45 1 0 0 0 0
22 38 1 0 0 0 0
S SKP 8
ID FL7ARXGL0005
KNApSAcK_ID C00011188
NAME Malvidin 3-glucoside-4-vinylcatechol
CAS_RN 403982-47-8
FORMULA C31H29O14
EXACTMASS 625.155730636
AVERAGEMASS 625.5535600000001
SMILES C(C6O)(C(OC(C6O)Oc(c4c(c5)cc(OC)c(O)c(OC)5)c(C=2)c(c1[o+1]4)c(OC2c(c3)ccc(O)c3O)cc(c1)O)CO)O
M END