Mol:FL7ARXGL0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.1683 1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1683 1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5803 1.3515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5803 1.3515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5803 2.0304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5803 2.0304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1683 2.3699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1683 2.3699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7562 2.0304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7562 2.0304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7562 1.3515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7562 1.3515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1683 0.3331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1683 0.3331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5803 -0.0063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5803 -0.0063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9924 0.3331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9924 0.3331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9924 1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9924 1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4044 1.3515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4044 1.3515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4044 2.0304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4044 2.0304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9924 2.3699 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -0.9924 2.3699 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3264 2.3596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3264 2.3596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5803 -0.6392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5803 -0.6392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1250 -0.9536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1250 -0.9536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0217 -0.9617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0217 -0.9617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1291 2.3385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1291 2.3385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6927 2.0131 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6927 2.0131 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2563 2.3385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2563 2.3385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2563 2.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2563 2.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6927 3.3146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6927 3.3146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1291 2.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1291 2.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6927 3.8013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6927 3.8013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7508 3.2747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7508 3.2747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1504 1.0312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1504 1.0312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3118 0.6475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3118 0.6475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5220 0.4091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5220 0.4091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2554 0.0313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2554 0.0313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6938 -0.6676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6938 -0.6676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4837 -0.4293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4837 -0.4293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7503 -0.0513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7503 -0.0513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8373 0.7746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8373 0.7746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2400 0.0799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2400 0.0799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4837 -1.0032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4837 -1.0032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2238 -0.8096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2238 -0.8096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2238 -1.4468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2238 -1.4468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5959 -2.0912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5959 -2.0912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3264 -2.0912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3264 -2.0912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2579 -2.6765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2579 -2.6765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6099 -3.2862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6099 -3.2862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2286 -3.2862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2286 -3.2862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3126 -3.8013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3126 -3.8013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 1 7 1 0 0 0 0 | + | 1 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 2 1 0 0 0 0 | + | 10 2 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 3 1 0 0 0 0 | + | 13 3 1 0 0 0 0 |
| − | 5 14 1 0 0 0 0 | + | 5 14 1 0 0 0 0 |
| − | 8 15 1 0 0 0 0 | + | 8 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 15 17 1 0 0 0 0 | + | 15 17 1 0 0 0 0 |
| − | 12 18 1 0 0 0 0 | + | 12 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 2 0 0 0 0 | + | 20 21 2 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 18 1 0 0 0 0 | + | 23 18 1 0 0 0 0 |
| − | 22 24 1 0 0 0 0 | + | 22 24 1 0 0 0 0 |
| − | 21 25 1 0 0 0 0 | + | 21 25 1 0 0 0 0 |
| − | 11 26 1 0 0 0 0 | + | 11 26 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 1 0 0 0 | + | 28 29 1 1 0 0 0 |
| − | 30 29 1 1 0 0 0 | + | 30 29 1 1 0 0 0 |
| − | 31 30 1 1 0 0 0 | + | 31 30 1 1 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 27 1 0 0 0 0 | + | 32 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 27 33 1 0 0 0 0 | + | 27 33 1 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 31 35 1 0 0 0 0 | + | 31 35 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 38 40 1 0 0 0 0 | + | 38 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 41 43 1 0 0 0 0 | + | 41 43 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7ARXGL0002 | + | ID FL7ARXGL0002 |
| − | KNApSAcK_ID C00011094 | + | KNApSAcK_ID C00011094 |
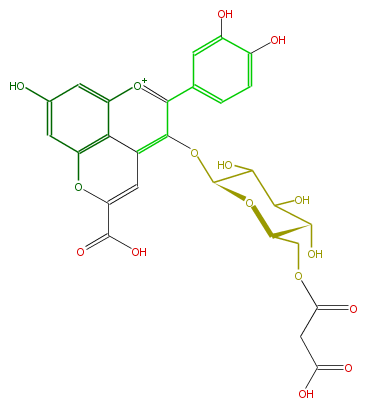
| − | NAME 5-Carboxypyranocyanidin 3-O-(6''-O-malonyl-beta-glucopyranoside) | + | NAME 5-Carboxypyranocyanidin 3-O-(6''-O-malonyl-beta-glucopyranoside) |
| − | CAS_RN 566942-53-8 | + | CAS_RN 566942-53-8 |
| − | FORMULA C27H23O16 | + | FORMULA C27H23O16 |
| − | EXACTMASS 603.098609688 | + | EXACTMASS 603.098609688 |
| − | AVERAGEMASS 603.46192 | + | AVERAGEMASS 603.46192 |
| − | SMILES c(c5)(ccc(c5O)O)c(c(OC(O4)C(C(O)C(C4COC(=O)CC(O)=O)O)O)2)[o+1]c(c13)cc(cc1OC(C(O)=O)=Cc23)O | + | SMILES c(c5)(ccc(c5O)O)c(c(OC(O4)C(C(O)C(C4COC(=O)CC(O)=O)O)O)2)[o+1]c(c13)cc(cc1OC(C(O)=O)=Cc23)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-2.1683 1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5803 1.3515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5803 2.0304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1683 2.3699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7562 2.0304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7562 1.3515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1683 0.3331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5803 -0.0063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9924 0.3331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9924 1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4044 1.3515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4044 2.0304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9924 2.3699 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-3.3264 2.3596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5803 -0.6392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1250 -0.9536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0217 -0.9617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1291 2.3385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6927 2.0131 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2563 2.3385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2563 2.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6927 3.3146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1291 2.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6927 3.8013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7508 3.2747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1504 1.0312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3118 0.6475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5220 0.4091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2554 0.0313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6938 -0.6676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4837 -0.4293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7503 -0.0513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8373 0.7746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2400 0.0799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4837 -1.0032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2238 -0.8096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2238 -1.4468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5959 -2.0912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3264 -2.0912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2579 -2.6765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6099 -3.2862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2286 -3.2862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3126 -3.8013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
1 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 2 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 3 1 0 0 0 0
5 14 1 0 0 0 0
8 15 1 0 0 0 0
15 16 2 0 0 0 0
15 17 1 0 0 0 0
12 18 1 0 0 0 0
18 19 2 0 0 0 0
19 20 1 0 0 0 0
20 21 2 0 0 0 0
21 22 1 0 0 0 0
22 23 2 0 0 0 0
23 18 1 0 0 0 0
22 24 1 0 0 0 0
21 25 1 0 0 0 0
11 26 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 1 0 0 0
30 29 1 1 0 0 0
31 30 1 1 0 0 0
31 32 1 0 0 0 0
32 27 1 0 0 0 0
26 28 1 0 0 0 0
27 33 1 0 0 0 0
32 34 1 0 0 0 0
31 35 1 0 0 0 0
30 36 1 0 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
38 40 1 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
41 43 1 0 0 0 0
S SKP 8
ID FL7ARXGL0002
KNApSAcK_ID C00011094
NAME 5-Carboxypyranocyanidin 3-O-(6''-O-malonyl-beta-glucopyranoside)
CAS_RN 566942-53-8
FORMULA C27H23O16
EXACTMASS 603.098609688
AVERAGEMASS 603.46192
SMILES c(c5)(ccc(c5O)O)c(c(OC(O4)C(C(O)C(C4COC(=O)CC(O)=O)O)O)2)[o+1]c(c13)cc(cc1OC(C(O)=O)=Cc23)O
M END