Mol:FL7AAIGL0031
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 57 62 0 0 0 0 0 0 0 0999 V2000 | + | 57 62 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.9484 0.1237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9484 0.1237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3379 0.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3379 0.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3379 1.1810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3379 1.1810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9484 1.5334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9484 1.5334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5588 1.1810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5588 1.1810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5588 0.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5588 0.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2725 0.1237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2725 0.1237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8830 0.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8830 0.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8830 1.1810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8830 1.1810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2725 1.5334 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.2725 1.5334 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4645 1.5167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4645 1.5167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0481 1.1798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0481 1.1798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6316 1.5167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6316 1.5167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6316 2.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6316 2.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0481 2.5275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0481 2.5275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4645 2.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4645 2.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2377 2.5405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2377 2.5405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0582 1.4693 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0582 1.4693 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9484 -0.5368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9484 -0.5368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3204 0.0412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3204 0.0412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0629 3.0772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0629 3.0772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5425 3.5568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5425 3.5568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2674 1.2292 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2674 1.2292 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8384 1.5589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8384 1.5589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4175 -0.1437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4175 -0.1437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6299 -0.3894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6299 -0.3894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3668 -0.7604 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3668 -0.7604 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8118 -1.4551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8118 -1.4551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5994 -1.2095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5994 -1.2095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8625 -0.8384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8625 -0.8384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8848 -0.0009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8848 -0.0009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4169 -0.6898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4169 -0.6898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7519 -1.7783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7519 -1.7783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3394 -1.7597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3394 -1.7597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3394 -2.3544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3394 -2.3544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5339 -1.4477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5339 -1.4477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8408 -1.8952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8408 -1.8952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2261 -1.3450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2261 -1.3450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4168 -1.1848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4168 -1.1848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1099 -0.7372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1099 -0.7372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7247 -1.2874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7247 -1.2874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1346 -0.8775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1346 -0.8775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0168 -0.4379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0168 -0.4379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8384 -1.9752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8384 -1.9752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3345 -1.8952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3345 -1.8952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2261 -1.8586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2261 -1.8586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7618 -2.6878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7618 -2.6878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2153 -2.3723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2153 -2.3723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7618 -3.2276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7618 -3.2276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1916 -3.5568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1916 -3.5568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5871 -3.2077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5871 -3.2077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5871 -2.5326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5871 -2.5326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0024 -2.1950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0024 -2.1950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4176 -2.5326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4176 -2.5326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4176 -3.2077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4176 -3.2077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0024 -3.5453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0024 -3.5453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0078 -2.2869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0078 -2.2869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 3 1 0 0 0 0 | + | 10 3 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 5 18 1 0 0 0 0 | + | 5 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 13 23 1 0 0 0 0 | + | 13 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 28 27 1 1 0 0 0 | + | 28 27 1 1 0 0 0 |
| − | 29 28 1 1 0 0 0 | + | 29 28 1 1 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 25 1 0 0 0 0 | + | 30 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 29 33 1 0 0 0 0 | + | 29 33 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 38 1 1 0 0 0 | + | 37 38 1 1 0 0 0 |
| − | 39 38 1 1 0 0 0 | + | 39 38 1 1 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 19 39 1 0 0 0 0 | + | 19 39 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 36 44 1 0 0 0 0 | + | 36 44 1 0 0 0 0 |
| − | 37 45 1 0 0 0 0 | + | 37 45 1 0 0 0 0 |
| − | 38 46 1 0 0 0 0 | + | 38 46 1 0 0 0 0 |
| − | 35 47 1 0 0 0 0 | + | 35 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 47 49 1 0 0 0 0 | + | 47 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 2 0 0 0 0 | + | 53 54 2 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 55 56 2 0 0 0 0 | + | 55 56 2 0 0 0 0 |
| − | 56 51 1 0 0 0 0 | + | 56 51 1 0 0 0 0 |
| − | 54 57 1 0 0 0 0 | + | 54 57 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAIGL0031 | + | ID FL7AAIGL0031 |
| − | KNApSAcK_ID C00011081 | + | KNApSAcK_ID C00011081 |
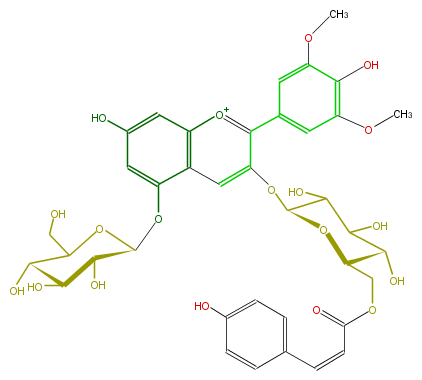
| − | NAME Malvidin 3-O-(6-O-(Z)-p-coumalonyl-beta-glucopyranoside)-5-O-beta-glucopyranoside | + | NAME Malvidin 3-O-(6-O-(Z)-p-coumalonyl-beta-glucopyranoside)-5-O-beta-glucopyranoside |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C38H41O19 | + | FORMULA C38H41O19 |
| − | EXACTMASS 801.22420413 | + | EXACTMASS 801.22420413 |
| − | AVERAGEMASS 801.72074 | + | AVERAGEMASS 801.72074 |
| − | SMILES C(=Cc(c6)ccc(O)c6)C(=O)OCC(O1)C(O)C(C(O)C1Oc(c(c(c5)cc(OC)c(c(OC)5)O)2)cc(c3OC(O4)C(O)C(C(O)C4CO)O)c(cc(c3)O)[o+1]2)O | + | SMILES C(=Cc(c6)ccc(O)c6)C(=O)OCC(O1)C(O)C(C(O)C1Oc(c(c(c5)cc(OC)c(c(OC)5)O)2)cc(c3OC(O4)C(O)C(C(O)C4CO)O)c(cc(c3)O)[o+1]2)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
57 62 0 0 0 0 0 0 0 0999 V2000
-0.9484 0.1237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3379 0.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3379 1.1810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9484 1.5334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5588 1.1810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5588 0.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2725 0.1237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8830 0.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8830 1.1810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2725 1.5334 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
1.4645 1.5167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0481 1.1798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6316 1.5167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6316 2.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0481 2.5275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4645 2.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2377 2.5405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0582 1.4693 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9484 -0.5368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3204 0.0412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0629 3.0772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5425 3.5568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2674 1.2292 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8384 1.5589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4175 -0.1437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6299 -0.3894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3668 -0.7604 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8118 -1.4551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5994 -1.2095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8625 -0.8384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8848 -0.0009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4169 -0.6898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7519 -1.7783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3394 -1.7597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3394 -2.3544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5339 -1.4477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8408 -1.8952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2261 -1.3450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4168 -1.1848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1099 -0.7372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7247 -1.2874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1346 -0.8775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0168 -0.4379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8384 -1.9752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3345 -1.8952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2261 -1.8586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7618 -2.6878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2153 -2.3723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7618 -3.2276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1916 -3.5568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5871 -3.2077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5871 -2.5326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0024 -2.1950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4176 -2.5326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4176 -3.2077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0024 -3.5453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0078 -2.2869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
2 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 3 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
5 18 1 0 0 0 0
1 19 1 0 0 0 0
8 20 1 0 0 0 0
15 21 1 0 0 0 0
21 22 1 0 0 0 0
13 23 1 0 0 0 0
23 24 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
28 27 1 1 0 0 0
29 28 1 1 0 0 0
29 30 1 0 0 0 0
30 25 1 0 0 0 0
20 26 1 0 0 0 0
25 31 1 0 0 0 0
30 32 1 0 0 0 0
29 33 1 0 0 0 0
28 34 1 0 0 0 0
34 35 1 0 0 0 0
36 37 1 1 0 0 0
37 38 1 1 0 0 0
39 38 1 1 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
41 36 1 0 0 0 0
19 39 1 0 0 0 0
41 42 1 0 0 0 0
42 43 1 0 0 0 0
36 44 1 0 0 0 0
37 45 1 0 0 0 0
38 46 1 0 0 0 0
35 47 1 0 0 0 0
47 48 2 0 0 0 0
47 49 1 0 0 0 0
49 50 2 0 0 0 0
50 51 1 0 0 0 0
51 52 2 0 0 0 0
52 53 1 0 0 0 0
53 54 2 0 0 0 0
54 55 1 0 0 0 0
55 56 2 0 0 0 0
56 51 1 0 0 0 0
54 57 1 0 0 0 0
S SKP 8
ID FL7AAIGL0031
KNApSAcK_ID C00011081
NAME Malvidin 3-O-(6-O-(Z)-p-coumalonyl-beta-glucopyranoside)-5-O-beta-glucopyranoside
CAS_RN -
FORMULA C38H41O19
EXACTMASS 801.22420413
AVERAGEMASS 801.72074
SMILES C(=Cc(c6)ccc(O)c6)C(=O)OCC(O1)C(O)C(C(O)C1Oc(c(c(c5)cc(OC)c(c(OC)5)O)2)cc(c3OC(O4)C(O)C(C(O)C4CO)O)c(cc(c3)O)[o+1]2)O
M END