Mol:FL7AAIGL0016
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 49 53 0 0 0 0 0 0 0 0999 V2000 | + | 49 53 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.7494 0.7729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7494 0.7729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7494 0.1306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7494 0.1306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1931 -0.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1931 -0.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6368 0.1306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6368 0.1306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6368 0.7729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6368 0.7729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1931 1.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1931 1.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0805 -0.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0805 -0.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5242 0.1306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5242 0.1306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5242 0.7729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5242 0.7729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0805 1.0941 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -1.0805 1.0941 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0319 1.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0319 1.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5989 0.7666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5989 0.7666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1658 1.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1658 1.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1658 1.7487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1658 1.7487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5989 2.0760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5989 2.0760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0319 1.7487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0319 1.7487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3055 1.0940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3055 1.0940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0318 2.2488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0318 2.2488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1931 -0.8327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1931 -0.8327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1081 -0.5901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1081 -0.5901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3947 -0.6139 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.3947 -0.6139 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.1265 -1.0785 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.1265 -1.0785 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.6423 -0.9311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6423 -0.9311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1612 -1.0785 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.1612 -1.0785 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.4295 -0.6139 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.4295 -0.6139 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.9137 -0.7613 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.9137 -0.7613 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.0302 -0.2493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0302 -0.2493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0554 -0.2325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0554 -0.2325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6912 -0.3020 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6912 -0.3020 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5636 -1.4809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5636 -1.4809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3425 -2.3063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3425 -2.3063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2273 -3.0588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2273 -3.0588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6724 -3.3791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6724 -3.3791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8478 -3.4716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8478 -3.4716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0631 -1.2307 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -4.0631 -1.2307 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.6920 -1.7206 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.6920 -1.7206 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.1575 -1.5128 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.1575 -1.5128 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.6417 -1.5072 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.6417 -1.5072 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.0165 -1.1324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0165 -1.1324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4841 -1.3792 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.4841 -1.3792 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.6540 -1.1789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6540 -1.1789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0423 -2.1818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0423 -2.1818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8512 -2.0269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8512 -2.0269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8619 -0.8446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8619 -0.8446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7364 -0.1326 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7364 -0.1326 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8823 2.6521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8823 2.6521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3237 3.5494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3237 3.5494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1658 1.0940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1658 1.0940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1658 1.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1658 1.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | 38 19 1 0 0 0 0 | + | 38 19 1 0 0 0 0 |
| − | 40 44 1 0 0 0 0 | + | 40 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 15 46 1 0 0 0 0 | + | 15 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 13 48 1 0 0 0 0 | + | 13 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 44 45 | + | M SAL 3 2 44 45 |
| − | M SBL 3 1 48 | + | M SBL 3 1 48 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 48 -3.949 -0.9183 | + | M SVB 3 48 -3.949 -0.9183 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 48 49 | + | M SAL 2 2 48 49 |
| − | M SBL 2 1 52 | + | M SBL 2 1 52 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 52 1.3754 0.8877 | + | M SVB 2 52 1.3754 0.8877 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 46 47 | + | M SAL 1 2 46 47 |
| − | M SBL 1 1 50 | + | M SBL 1 1 50 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 50 0.8823 2.6521 | + | M SVB 1 50 0.8823 2.6521 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAIGL0016 | + | ID FL7AAIGL0016 |
| − | KNApSAcK_ID C00006907 | + | KNApSAcK_ID C00006907 |
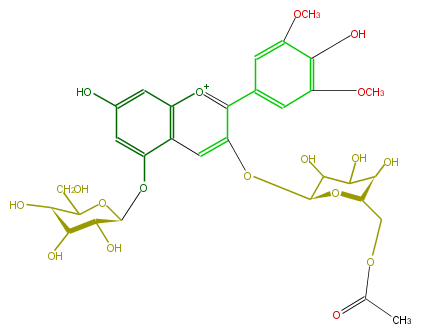
| − | NAME Malvidin 3-(6''-acetylglucoside)-5-glucoside | + | NAME Malvidin 3-(6''-acetylglucoside)-5-glucoside |
| − | CAS_RN 164266-06-2 | + | CAS_RN 164266-06-2 |
| − | FORMULA C31H37O18 | + | FORMULA C31H37O18 |
| − | EXACTMASS 697.1979893800001 | + | EXACTMASS 697.1979893800001 |
| − | AVERAGEMASS 697.61468 | + | AVERAGEMASS 697.61468 |
| − | SMILES O([C@@H](C(O)5)O[C@@H]([C@@H](C5O)O)COC(C)=O)c(c3)c(c(c4)cc(c(O)c4OC)OC)[o+1]c(c31)cc(O)cc1O[C@@H]([C@H]2O)OC([C@H](O)[C@@H]2O)CO | + | SMILES O([C@@H](C(O)5)O[C@@H]([C@@H](C5O)O)COC(C)=O)c(c3)c(c(c4)cc(c(O)c4OC)OC)[o+1]c(c31)cc(O)cc1O[C@@H]([C@H]2O)OC([C@H](O)[C@@H]2O)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
49 53 0 0 0 0 0 0 0 0999 V2000
-2.7494 0.7729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7494 0.1306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1931 -0.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6368 0.1306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6368 0.7729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1931 1.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0805 -0.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5242 0.1306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5242 0.7729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0805 1.0941 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.0319 1.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5989 0.7666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1658 1.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1658 1.7487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5989 2.0760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0319 1.7487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3055 1.0940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0318 2.2488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1931 -0.8327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1081 -0.5901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3947 -0.6139 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.1265 -1.0785 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.6423 -0.9311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1612 -1.0785 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.4295 -0.6139 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.9137 -0.7613 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.0302 -0.2493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0554 -0.2325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6912 -0.3020 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5636 -1.4809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3425 -2.3063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2273 -3.0588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6724 -3.3791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8478 -3.4716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0631 -1.2307 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.6920 -1.7206 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.1575 -1.5128 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.6417 -1.5072 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.0165 -1.1324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4841 -1.3792 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.6540 -1.1789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0423 -2.1818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8512 -2.0269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8619 -0.8446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7364 -0.1326 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8823 2.6521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3237 3.5494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1658 1.0940 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1658 1.0940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
22 20 1 0 0 0 0
24 30 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
35 36 1 1 0 0 0
36 37 1 1 0 0 0
38 37 1 1 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
40 35 1 0 0 0 0
35 41 1 0 0 0 0
36 42 1 0 0 0 0
37 43 1 0 0 0 0
38 19 1 0 0 0 0
40 44 1 0 0 0 0
44 45 1 0 0 0 0
15 46 1 0 0 0 0
46 47 1 0 0 0 0
13 48 1 0 0 0 0
48 49 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 44 45
M SBL 3 1 48
M SMT 3 CH2OH
M SVB 3 48 -3.949 -0.9183
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 48 49
M SBL 2 1 52
M SMT 2 OCH3
M SVB 2 52 1.3754 0.8877
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 46 47
M SBL 1 1 50
M SMT 1 OCH3
M SVB 1 50 0.8823 2.6521
S SKP 8
ID FL7AAIGL0016
KNApSAcK_ID C00006907
NAME Malvidin 3-(6''-acetylglucoside)-5-glucoside
CAS_RN 164266-06-2
FORMULA C31H37O18
EXACTMASS 697.1979893800001
AVERAGEMASS 697.61468
SMILES O([C@@H](C(O)5)O[C@@H]([C@@H](C5O)O)COC(C)=O)c(c3)c(c(c4)cc(c(O)c4OC)OC)[o+1]c(c31)cc(O)cc1O[C@@H]([C@H]2O)OC([C@H](O)[C@@H]2O)CO
M END