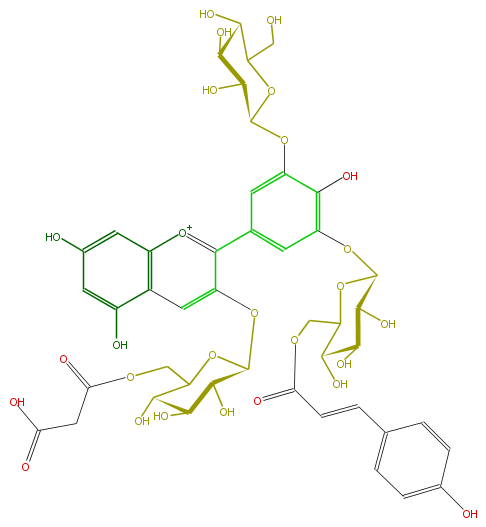
Mol:FL7AAGGL0055
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 72 78 0 0 0 0 0 0 0 0999 V2000 | + | 72 78 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.3969 0.2521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3969 0.2521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3969 -0.5729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3969 -0.5729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6825 -0.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6825 -0.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9680 -0.5729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9680 -0.5729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9680 0.2521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9680 0.2521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6825 0.6646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6825 0.6646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2535 -0.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2535 -0.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5390 -0.5729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5390 -0.5729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5390 0.2521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5390 0.2521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2535 0.6646 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -1.2535 0.6646 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2375 0.7004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2375 0.7004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9520 0.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9520 0.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6664 0.7004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6664 0.7004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6664 1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6664 1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9520 1.9379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9520 1.9379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2375 1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2375 1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3066 1.8950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3066 1.8950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3195 -1.0686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3195 -1.0686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9769 0.5869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9769 0.5869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6825 -1.7481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6825 -1.7481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9520 2.6691 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9520 2.6691 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3314 0.3165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3314 0.3165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8750 -2.9173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8750 -2.9173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1152 -3.2393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1152 -3.2393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6038 -2.5915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6038 -2.5915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1663 -2.2948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1663 -2.2948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5935 -1.9726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5935 -1.9726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1050 -2.6204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1050 -2.6204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5831 -2.2472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5831 -2.2472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1928 -3.4002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1928 -3.4002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6293 -3.3027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6293 -3.3027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3553 -3.2057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3553 -3.2057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7454 -2.0186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7454 -2.0186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5403 -1.7971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5403 -1.7971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5368 -0.9719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5368 -0.9719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9543 -0.2600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9543 -0.2600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1592 -0.4814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1592 -0.4814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1627 -1.3067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1627 -1.3067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0929 -2.6070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0929 -2.6070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1776 -2.1671 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1776 -2.1671 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1138 -1.2974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1138 -1.2974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4853 -1.2341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4853 -1.2341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1822 -1.6496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1822 -1.6496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6339 -4.3933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6339 -4.3933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3612 -3.6158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3612 -3.6158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8982 -2.9910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8982 -2.9910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5516 -3.4633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5516 -3.4633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2789 -2.6858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2789 -2.6858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8158 -2.0609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8158 -2.0609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3742 -2.4514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3742 -2.4514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0014 5.1802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0014 5.1802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4637 4.4966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4637 4.4966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0734 3.8700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0734 3.8700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2162 3.0572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2162 3.0572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6787 3.7408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6787 3.7408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1416 4.3674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1416 4.3674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6472 5.4035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6472 5.4035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4269 5.0134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4269 5.0134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5772 3.7445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5772 3.7445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7048 4.7508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7048 4.7508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6667 5.2636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6667 5.2636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1614 -2.7390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1614 -2.7390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3678 -2.9601 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3678 -2.9601 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7497 -3.3158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7497 -3.3158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5434 -3.0947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5434 -3.0947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1317 -3.6715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1317 -3.6715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9260 -4.4705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9260 -4.4705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5151 -5.0481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5151 -5.0481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3099 -4.8267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3099 -4.8267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5155 -4.0278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5155 -4.0278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9264 -3.4502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9264 -3.4502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8982 -5.4035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8982 -5.4035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 8 18 1 0 0 0 0 | + | 8 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 13 22 1 0 0 0 0 | + | 13 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 26 18 1 0 0 0 0 | + | 26 18 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 36 22 1 0 0 0 0 | + | 36 22 1 0 0 0 0 |
| − | 38 42 1 0 0 0 0 | + | 38 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 45 47 1 0 0 0 0 | + | 45 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 48 50 1 0 0 0 0 | + | 48 50 1 0 0 0 0 |
| − | 50 29 1 0 0 0 0 | + | 50 29 1 0 0 0 0 |
| − | 51 52 1 1 0 0 0 | + | 51 52 1 1 0 0 0 |
| − | 52 53 1 1 0 0 0 | + | 52 53 1 1 0 0 0 |
| − | 54 53 1 1 0 0 0 | + | 54 53 1 1 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 51 1 0 0 0 0 | + | 56 51 1 0 0 0 0 |
| − | 51 57 1 0 0 0 0 | + | 51 57 1 0 0 0 0 |
| − | 52 58 1 0 0 0 0 | + | 52 58 1 0 0 0 0 |
| − | 53 59 1 0 0 0 0 | + | 53 59 1 0 0 0 0 |
| − | 56 60 1 0 0 0 0 | + | 56 60 1 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 21 54 1 0 0 0 0 | + | 21 54 1 0 0 0 0 |
| − | 62 63 2 0 0 0 0 | + | 62 63 2 0 0 0 0 |
| − | 62 64 1 0 0 0 0 | + | 62 64 1 0 0 0 0 |
| − | 64 65 2 0 0 0 0 | + | 64 65 2 0 0 0 0 |
| − | 65 66 1 0 0 0 0 | + | 65 66 1 0 0 0 0 |
| − | 66 67 2 0 0 0 0 | + | 66 67 2 0 0 0 0 |
| − | 67 68 1 0 0 0 0 | + | 67 68 1 0 0 0 0 |
| − | 68 69 2 0 0 0 0 | + | 68 69 2 0 0 0 0 |
| − | 69 70 1 0 0 0 0 | + | 69 70 1 0 0 0 0 |
| − | 70 71 2 0 0 0 0 | + | 70 71 2 0 0 0 0 |
| − | 71 66 1 0 0 0 0 | + | 71 66 1 0 0 0 0 |
| − | 69 72 1 0 0 0 0 | + | 69 72 1 0 0 0 0 |
| − | 43 62 1 0 0 0 0 | + | 43 62 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAGGL0055 | + | ID FL7AAGGL0055 |
| − | KNApSAcK_ID C00014806 | + | KNApSAcK_ID C00014806 |
| − | NAME Ternatin C3 | + | NAME Ternatin C3 |
| − | CAS_RN 215378-76-0 | + | CAS_RN 215378-76-0 |
| − | FORMULA C45H49O27 | + | FORMULA C45H49O27 |
| − | EXACTMASS 1021.2461213619999 | + | EXACTMASS 1021.2461213619999 |
| − | AVERAGEMASS 1021.8543599999999 | + | AVERAGEMASS 1021.8543599999999 |
| − | SMILES C(O)(C7O)C(CO)OC(C7O)Oc(c3)c(c(cc3c(c5OC(O6)C(O)C(C(O)C6COC(CC(O)=O)=O)O)[o+1]c(c(c5)4)cc(cc(O)4)O)OC(O1)C(O)C(O)C(C1COC(C=Cc(c2)ccc(c2)O)=O)O)O | + | SMILES C(O)(C7O)C(CO)OC(C7O)Oc(c3)c(c(cc3c(c5OC(O6)C(O)C(C(O)C6COC(CC(O)=O)=O)O)[o+1]c(c(c5)4)cc(cc(O)4)O)OC(O1)C(O)C(O)C(C1COC(C=Cc(c2)ccc(c2)O)=O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
72 78 0 0 0 0 0 0 0 0999 V2000
-3.3969 0.2521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3969 -0.5729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6825 -0.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9680 -0.5729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9680 0.2521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6825 0.6646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2535 -0.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5390 -0.5729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5390 0.2521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2535 0.6646 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.2375 0.7004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9520 0.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6664 0.7004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6664 1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9520 1.9379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2375 1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3066 1.8950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3195 -1.0686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9769 0.5869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6825 -1.7481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9520 2.6691 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3314 0.3165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8750 -2.9173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1152 -3.2393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6038 -2.5915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1663 -2.2948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5935 -1.9726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1050 -2.6204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5831 -2.2472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1928 -3.4002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6293 -3.3027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3553 -3.2057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7454 -2.0186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5403 -1.7971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5368 -0.9719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9543 -0.2600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1592 -0.4814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1627 -1.3067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0929 -2.6070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1776 -2.1671 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1138 -1.2974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4853 -1.2341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1822 -1.6496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6339 -4.3933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3612 -3.6158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8982 -2.9910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5516 -3.4633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2789 -2.6858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8158 -2.0609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3742 -2.4514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0014 5.1802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4637 4.4966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0734 3.8700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2162 3.0572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6787 3.7408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1416 4.3674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6472 5.4035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4269 5.0134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5772 3.7445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7048 4.7508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6667 5.2636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1614 -2.7390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3678 -2.9601 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7497 -3.3158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5434 -3.0947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1317 -3.6715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9260 -4.4705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5151 -5.0481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3099 -4.8267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5155 -4.0278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9264 -3.4502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8982 -5.4035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
8 18 1 0 0 0 0
1 19 1 0 0 0 0
3 20 1 0 0 0 0
15 21 1 0 0 0 0
13 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
25 32 1 0 0 0 0
26 18 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
35 41 1 0 0 0 0
36 22 1 0 0 0 0
38 42 1 0 0 0 0
42 43 1 0 0 0 0
44 45 2 0 0 0 0
45 46 1 0 0 0 0
45 47 1 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
48 50 1 0 0 0 0
50 29 1 0 0 0 0
51 52 1 1 0 0 0
52 53 1 1 0 0 0
54 53 1 1 0 0 0
54 55 1 0 0 0 0
55 56 1 0 0 0 0
56 51 1 0 0 0 0
51 57 1 0 0 0 0
52 58 1 0 0 0 0
53 59 1 0 0 0 0
56 60 1 0 0 0 0
60 61 1 0 0 0 0
21 54 1 0 0 0 0
62 63 2 0 0 0 0
62 64 1 0 0 0 0
64 65 2 0 0 0 0
65 66 1 0 0 0 0
66 67 2 0 0 0 0
67 68 1 0 0 0 0
68 69 2 0 0 0 0
69 70 1 0 0 0 0
70 71 2 0 0 0 0
71 66 1 0 0 0 0
69 72 1 0 0 0 0
43 62 1 0 0 0 0
S SKP 8
ID FL7AAGGL0055
KNApSAcK_ID C00014806
NAME Ternatin C3
CAS_RN 215378-76-0
FORMULA C45H49O27
EXACTMASS 1021.2461213619999
AVERAGEMASS 1021.8543599999999
SMILES C(O)(C7O)C(CO)OC(C7O)Oc(c3)c(c(cc3c(c5OC(O6)C(O)C(C(O)C6COC(CC(O)=O)=O)O)[o+1]c(c(c5)4)cc(cc(O)4)O)OC(O1)C(O)C(O)C(C1COC(C=Cc(c2)ccc(c2)O)=O)O)O
M END