Mol:FL7AAGGL0021
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 55 60 0 0 0 0 0 0 0 0999 V2000 | + | 55 60 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.5627 0.9248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5627 0.9248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5627 0.2824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5627 0.2824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0064 -0.0387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0064 -0.0387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4501 0.2824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4501 0.2824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4501 0.9248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4501 0.9248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0064 1.2460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0064 1.2460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1062 -0.0387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1062 -0.0387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6625 0.2824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6625 0.2824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6625 0.9248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6625 0.9248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1062 1.2460 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.1062 1.2460 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2186 1.2459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2186 1.2459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7856 0.9185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7856 0.9185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3525 1.2459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3525 1.2459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3525 1.9006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3525 1.9006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7856 2.2279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7856 2.2279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2186 1.9006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2186 1.9006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1188 1.2459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1188 1.2459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9194 2.2278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9194 2.2278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0064 -0.6809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0064 -0.6809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0786 -0.4383 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0786 -0.4383 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2360 -0.1382 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.2360 -0.1382 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.9352 -0.6592 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.9352 -0.6592 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.5137 -0.4939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5137 -0.4939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0956 -0.6592 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.0956 -0.6592 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.3965 -0.1382 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.3965 -0.1382 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.8180 -0.3035 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.8180 -0.3035 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.6773 -0.2879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6773 -0.2879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8595 0.1711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8595 0.1711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7509 0.2162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7509 0.2162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6472 -0.6592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6472 -0.6592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3302 -0.8519 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.3302 -0.8519 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.9590 -1.3419 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.9590 -1.3419 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.4245 -1.1340 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.4245 -1.1340 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.8135 -1.2821 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.8135 -1.2821 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.2835 -0.7536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2835 -0.7536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7511 -1.0004 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.7511 -1.0004 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.9211 -0.8002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9211 -0.8002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3093 -1.8031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3093 -1.8031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1182 -1.6481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1182 -1.6481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3112 -0.1720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3112 -0.1720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8378 -1.3704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8378 -1.3704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5484 -1.8717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5484 -1.8717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0332 -1.8717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0332 -1.8717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7389 -2.3815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7389 -2.3815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2496 -2.3815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2496 -2.3815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9604 -1.8806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9604 -1.8806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3820 -1.8806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3820 -1.8806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0928 -2.3815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0928 -2.3815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3820 -2.8824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3820 -2.8824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9604 -2.8824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9604 -2.8824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5153 -2.3815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5153 -2.3815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8440 -2.3837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8440 -2.3837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8738 0.7273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8738 0.7273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7856 2.8824 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7856 2.8824 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9194 0.9186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9194 0.9186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 34 19 1 0 0 0 0 | + | 34 19 1 0 0 0 0 |
| − | 30 41 1 0 0 0 0 | + | 30 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 48 51 1 0 0 0 0 | + | 48 51 1 0 0 0 0 |
| − | 42 52 2 0 0 0 0 | + | 42 52 2 0 0 0 0 |
| − | 40 53 1 0 0 0 0 | + | 40 53 1 0 0 0 0 |
| − | 15 54 1 0 0 0 0 | + | 15 54 1 0 0 0 0 |
| − | 13 55 1 0 0 0 0 | + | 13 55 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 53 | + | M SAL 1 2 40 53 |
| − | M SBL 1 1 43 | + | M SBL 1 1 43 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 43 -3.1369 -0.6147 | + | M SVB 1 43 -3.1369 -0.6147 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAGGL0021 | + | ID FL7AAGGL0021 |
| − | KNApSAcK_ID C00006884 | + | KNApSAcK_ID C00006884 |
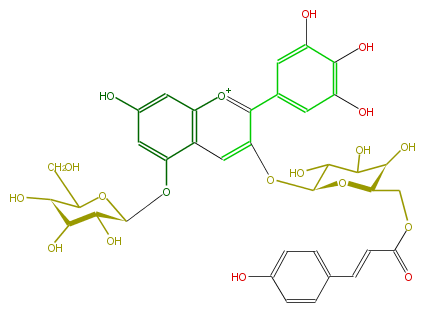
| − | NAME Awobanin;Delphinidin-3-(6-O-p-coumarylglucoside)-5-glucoside | + | NAME Awobanin;Delphinidin-3-(6-O-p-coumarylglucoside)-5-glucoside |
| − | CAS_RN 64615-55-0,136031-06-6 | + | CAS_RN 64615-55-0,136031-06-6 |
| − | FORMULA C36H37O19 | + | FORMULA C36H37O19 |
| − | EXACTMASS 773.192904002 | + | EXACTMASS 773.192904002 |
| − | AVERAGEMASS 773.66758 | + | AVERAGEMASS 773.66758 |
| − | SMILES O[C@@H](C(CO)1)[C@@H]([C@@H]([C@H](Oc(c62)cc(cc2[o+1]c(c(c6)O[C@H](O4)C(C([C@@H](O)[C@H]4COC(C=Cc(c5)ccc(c5)O)=O)O)O)c(c3)cc(O)c(O)c(O)3)O)O1)O)O | + | SMILES O[C@@H](C(CO)1)[C@@H]([C@@H]([C@H](Oc(c62)cc(cc2[o+1]c(c(c6)O[C@H](O4)C(C([C@@H](O)[C@H]4COC(C=Cc(c5)ccc(c5)O)=O)O)O)c(c3)cc(O)c(O)c(O)3)O)O1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
55 60 0 0 0 0 0 0 0 0999 V2000
-1.5627 0.9248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5627 0.2824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0064 -0.0387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4501 0.2824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4501 0.9248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0064 1.2460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1062 -0.0387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6625 0.2824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6625 0.9248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1062 1.2460 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
1.2186 1.2459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7856 0.9185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3525 1.2459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3525 1.9006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7856 2.2279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2186 1.9006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1188 1.2459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9194 2.2278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0064 -0.6809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0786 -0.4383 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2360 -0.1382 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.9352 -0.6592 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.5137 -0.4939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0956 -0.6592 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.3965 -0.1382 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.8180 -0.3035 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.6773 -0.2879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8595 0.1711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7509 0.2162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6472 -0.6592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3302 -0.8519 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.9590 -1.3419 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.4245 -1.1340 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.8135 -1.2821 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.2835 -0.7536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7511 -1.0004 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.9211 -0.8002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3093 -1.8031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1182 -1.6481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3112 -0.1720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8378 -1.3704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5484 -1.8717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0332 -1.8717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7389 -2.3815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2496 -2.3815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9604 -1.8806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3820 -1.8806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0928 -2.3815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3820 -2.8824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9604 -2.8824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5153 -2.3815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8440 -2.3837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8738 0.7273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7856 2.8824 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9194 0.9186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
22 20 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
36 40 1 0 0 0 0
34 19 1 0 0 0 0
30 41 1 0 0 0 0
41 42 1 0 0 0 0
42 43 1 0 0 0 0
43 44 2 0 0 0 0
44 45 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 45 1 0 0 0 0
48 51 1 0 0 0 0
42 52 2 0 0 0 0
40 53 1 0 0 0 0
15 54 1 0 0 0 0
13 55 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 53
M SBL 1 1 43
M SMT 1 CH2OH
M SVB 1 43 -3.1369 -0.6147
S SKP 8
ID FL7AAGGL0021
KNApSAcK_ID C00006884
NAME Awobanin;Delphinidin-3-(6-O-p-coumarylglucoside)-5-glucoside
CAS_RN 64615-55-0,136031-06-6
FORMULA C36H37O19
EXACTMASS 773.192904002
AVERAGEMASS 773.66758
SMILES O[C@@H](C(CO)1)[C@@H]([C@@H]([C@H](Oc(c62)cc(cc2[o+1]c(c(c6)O[C@H](O4)C(C([C@@H](O)[C@H]4COC(C=Cc(c5)ccc(c5)O)=O)O)O)c(c3)cc(O)c(O)c(O)3)O)O1)O)O
M END