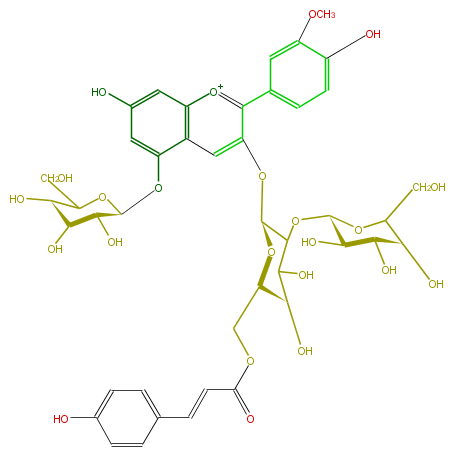
Mol:FL7AADGL0021
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 66 72 0 0 0 0 0 0 0 0999 V2000 | + | 66 72 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.2544 2.4398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2544 2.4398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2544 1.7974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2544 1.7974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6981 1.4762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6981 1.4762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1418 1.7974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1418 1.7974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1418 2.4398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1418 2.4398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6981 2.7610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6981 2.7610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5855 1.4762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5855 1.4762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0292 1.7974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0292 1.7974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0292 2.4398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0292 2.4398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5855 2.7610 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -0.5855 2.7610 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5269 2.7608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5269 2.7608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0939 2.4335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0939 2.4335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6608 2.7608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6608 2.7608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6608 3.4155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6608 3.4155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0939 3.7429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0939 3.7429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5269 3.4155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5269 3.4155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8105 2.7608 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8105 2.7608 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5268 3.9155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5268 3.9155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6981 0.8341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6981 0.8341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3869 1.0767 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3869 1.0767 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8654 -0.2374 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.8654 -0.2374 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.3584 0.1502 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.3584 0.1502 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.5679 -0.4477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5679 -0.4477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3148 -1.1458 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.3148 -1.1458 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.8654 -1.4637 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.8654 -1.4637 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.6907 -0.8524 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.6907 -0.8524 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.0401 0.1674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0401 0.1674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1922 -0.8963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1922 -0.8963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1699 -2.4162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1699 -2.4162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2099 -1.9971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2099 -1.9971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7098 0.2649 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 1.7098 0.2649 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 2.0134 -0.3270 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.0134 -0.3270 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.6220 -0.2060 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.6220 -0.2060 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.1728 -0.2969 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.1728 -0.2969 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.8438 0.1730 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.8438 0.1730 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.2990 -0.0021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2990 -0.0021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3913 -0.2483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3913 -0.2483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8518 -0.8115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8518 -0.8115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1526 -2.6312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1526 -2.6312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8569 0.5599 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.8569 0.5599 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.4857 0.0700 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.4857 0.0700 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.9512 0.2778 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.9512 0.2778 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.4355 0.2834 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.4355 0.2834 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.8102 0.6582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8102 0.6582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2778 0.4114 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.2778 0.4114 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.4478 0.6116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4478 0.6116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8360 -0.3912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8360 -0.3912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6450 -0.2363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6450 -0.2363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1853 -3.2164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1853 -3.2164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1424 -3.7841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1424 -3.7841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7521 -3.2164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7521 -3.2164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0762 -3.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0762 -3.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7031 -3.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7031 -3.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0155 -3.2365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0155 -3.2365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6405 -3.2365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6405 -3.2365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9530 -3.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9530 -3.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6405 -4.3190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6405 -4.3190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0155 -4.3190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0155 -4.3190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5773 -3.7777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5773 -3.7777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7804 -1.0912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7804 -1.0912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3773 4.3190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3773 4.3190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8187 5.2163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8187 5.2163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4574 0.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4574 0.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1332 1.3365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1332 1.3365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9941 1.0400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9941 1.0400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5754 1.6254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5754 1.6254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 30 39 1 0 0 0 0 | + | 30 39 1 0 0 0 0 |
| − | 40 41 1 1 0 0 0 | + | 40 41 1 1 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 43 42 1 1 0 0 0 | + | 43 42 1 1 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 40 1 0 0 0 0 | + | 45 40 1 0 0 0 0 |
| − | 40 46 1 0 0 0 0 | + | 40 46 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 19 1 0 0 0 0 | + | 43 19 1 0 0 0 0 |
| − | 39 49 1 0 0 0 0 | + | 39 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 49 51 1 0 0 0 0 | + | 49 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 2 0 0 0 0 | + | 53 54 2 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 55 56 2 0 0 0 0 | + | 55 56 2 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 57 58 2 0 0 0 0 | + | 57 58 2 0 0 0 0 |
| − | 58 53 1 0 0 0 0 | + | 58 53 1 0 0 0 0 |
| − | 56 59 1 0 0 0 0 | + | 56 59 1 0 0 0 0 |
| − | 34 60 1 0 0 0 0 | + | 34 60 1 0 0 0 0 |
| − | 15 61 1 0 0 0 0 | + | 15 61 1 0 0 0 0 |
| − | 61 62 1 0 0 0 0 | + | 61 62 1 0 0 0 0 |
| − | 35 63 1 0 0 0 0 | + | 35 63 1 0 0 0 0 |
| − | 63 64 1 0 0 0 0 | + | 63 64 1 0 0 0 0 |
| − | 45 65 1 0 0 0 0 | + | 45 65 1 0 0 0 0 |
| − | 65 66 1 0 0 0 0 | + | 65 66 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 65 66 | + | M SAL 3 2 65 66 |
| − | M SBL 3 1 71 | + | M SBL 3 1 71 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 71 -3.6613 1.2838 | + | M SVB 3 71 -3.6613 1.2838 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 63 64 | + | M SAL 2 2 63 64 |
| − | M SBL 2 1 69 | + | M SBL 2 1 69 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 69 3.6563 0.6447 | + | M SVB 2 69 3.6563 0.6447 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 61 62 | + | M SAL 1 2 61 62 |
| − | M SBL 1 1 67 | + | M SBL 1 1 67 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 67 1.3773 4.319 | + | M SVB 1 67 1.3773 4.319 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AADGL0021 | + | ID FL7AADGL0021 |
| − | KNApSAcK_ID C00006869 | + | KNApSAcK_ID C00006869 |
| − | NAME Peonidin 3-p-coumarylsophoroside-5-glucoside | + | NAME Peonidin 3-p-coumarylsophoroside-5-glucoside |
| − | CAS_RN 38718-99-9 | + | CAS_RN 38718-99-9 |
| − | FORMULA C43H49O23 | + | FORMULA C43H49O23 |
| − | EXACTMASS 933.266462874 | + | EXACTMASS 933.266462874 |
| − | AVERAGEMASS 933.83536 | + | AVERAGEMASS 933.83536 |
| − | SMILES O([C@@H]6COC(=O)C=Cc(c7)ccc(c7)O)[C@H](C(C([C@@H](O)6)O)O[C@@H](O5)[C@H](O)[C@@H]([C@@H](C(CO)5)O)O)Oc(c4)c([o+1]c(c42)cc(cc(O[C@H](O3)[C@@H](O)[C@H]([C@H](C(CO)3)O)O)2)O)c(c1)ccc(O)c1OC | + | SMILES O([C@@H]6COC(=O)C=Cc(c7)ccc(c7)O)[C@H](C(C([C@@H](O)6)O)O[C@@H](O5)[C@H](O)[C@@H]([C@@H](C(CO)5)O)O)Oc(c4)c([o+1]c(c42)cc(cc(O[C@H](O3)[C@@H](O)[C@H]([C@H](C(CO)3)O)O)2)O)c(c1)ccc(O)c1OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
66 72 0 0 0 0 0 0 0 0999 V2000
-2.2544 2.4398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2544 1.7974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6981 1.4762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1418 1.7974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1418 2.4398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6981 2.7610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5855 1.4762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0292 1.7974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0292 2.4398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5855 2.7610 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.5269 2.7608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0939 2.4335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6608 2.7608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6608 3.4155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0939 3.7429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5269 3.4155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8105 2.7608 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5268 3.9155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6981 0.8341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3869 1.0767 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8654 -0.2374 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.3584 0.1502 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.5679 -0.4477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3148 -1.1458 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.8654 -1.4637 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.6907 -0.8524 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.0401 0.1674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1922 -0.8963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1699 -2.4162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2099 -1.9971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7098 0.2649 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
2.0134 -0.3270 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.6220 -0.2060 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.1728 -0.2969 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.8438 0.1730 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.2990 -0.0021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3913 -0.2483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8518 -0.8115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1526 -2.6312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8569 0.5599 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.4857 0.0700 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.9512 0.2778 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.4355 0.2834 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.8102 0.6582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2778 0.4114 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.4478 0.6116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8360 -0.3912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6450 -0.2363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1853 -3.2164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1424 -3.7841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7521 -3.2164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0762 -3.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7031 -3.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0155 -3.2365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6405 -3.2365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9530 -3.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6405 -4.3190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0155 -4.3190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5773 -3.7777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7804 -1.0912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3773 4.3190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8187 5.2163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4574 0.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1332 1.3365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9941 1.0400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5754 1.6254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
27 31 1 0 0 0 0
22 20 1 0 0 0 0
30 39 1 0 0 0 0
40 41 1 1 0 0 0
41 42 1 1 0 0 0
43 42 1 1 0 0 0
43 44 1 0 0 0 0
44 45 1 0 0 0 0
45 40 1 0 0 0 0
40 46 1 0 0 0 0
41 47 1 0 0 0 0
42 48 1 0 0 0 0
43 19 1 0 0 0 0
39 49 1 0 0 0 0
49 50 2 0 0 0 0
49 51 1 0 0 0 0
51 52 2 0 0 0 0
52 53 1 0 0 0 0
53 54 2 0 0 0 0
54 55 1 0 0 0 0
55 56 2 0 0 0 0
56 57 1 0 0 0 0
57 58 2 0 0 0 0
58 53 1 0 0 0 0
56 59 1 0 0 0 0
34 60 1 0 0 0 0
15 61 1 0 0 0 0
61 62 1 0 0 0 0
35 63 1 0 0 0 0
63 64 1 0 0 0 0
45 65 1 0 0 0 0
65 66 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 65 66
M SBL 3 1 71
M SMT 3 CH2OH
M SVB 3 71 -3.6613 1.2838
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 63 64
M SBL 2 1 69
M SMT 2 CH2OH
M SVB 2 69 3.6563 0.6447
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 61 62
M SBL 1 1 67
M SMT 1 OCH3
M SVB 1 67 1.3773 4.319
S SKP 8
ID FL7AADGL0021
KNApSAcK_ID C00006869
NAME Peonidin 3-p-coumarylsophoroside-5-glucoside
CAS_RN 38718-99-9
FORMULA C43H49O23
EXACTMASS 933.266462874
AVERAGEMASS 933.83536
SMILES O([C@@H]6COC(=O)C=Cc(c7)ccc(c7)O)[C@H](C(C([C@@H](O)6)O)O[C@@H](O5)[C@H](O)[C@@H]([C@@H](C(CO)5)O)O)Oc(c4)c([o+1]c(c42)cc(cc(O[C@H](O3)[C@@H](O)[C@H]([C@H](C(CO)3)O)O)2)O)c(c1)ccc(O)c1OC
M END