Mol:FL7AADGL0012
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 39 42 0 0 0 0 0 0 0 0999 V2000 | + | 39 42 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.8080 0.9682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8080 0.9682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8080 0.3259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8080 0.3259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2517 0.0047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2517 0.0047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6954 0.3259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6954 0.3259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6954 0.9682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6954 0.9682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2517 1.2894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2517 1.2894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1391 0.0047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1391 0.0047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5828 0.3259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5828 0.3259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5828 0.9682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5828 0.9682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1391 1.2894 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -2.1391 1.2894 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0267 1.2893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0267 1.2893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4597 0.9620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4597 0.9620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1072 1.2893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1072 1.2893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1072 1.9440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1072 1.9440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4597 2.2713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4597 2.2713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0267 1.9440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0267 1.9440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3641 1.2893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3641 1.2893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9732 2.4440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9732 2.4440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2517 -0.6374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2517 -0.6374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1667 -0.3948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1667 -0.3948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3361 -0.4186 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.3361 -0.4186 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.0679 -0.8832 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.0679 -0.8832 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.5837 -0.7358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5837 -0.7358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1027 -0.8832 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.1027 -0.8832 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.3709 -0.4186 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.3709 -0.4186 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.8551 -0.5660 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.8551 -0.5660 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.0284 -0.0540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0284 -0.0540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9968 -0.0372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9968 -0.0372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6326 -0.1067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6326 -0.1067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5051 -1.2855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5051 -1.2855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0024 -2.0391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0024 -2.0391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8600 -2.7844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8600 -2.7844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2705 -3.1247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2705 -3.1247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4803 -3.0731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4803 -3.0731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4437 -3.7731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4437 -3.7731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0319 -4.1127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0319 -4.1127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7382 -4.1804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7382 -4.1804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1763 2.8475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1763 2.8475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2650 3.7448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2650 3.7448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 15 38 1 0 0 0 0 | + | 15 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 38 39 | + | M SAL 2 2 38 39 |
| − | M SBL 2 1 41 | + | M SBL 2 1 41 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 41 -0.1763 2.8475 | + | M SVB 2 41 -0.1763 2.8475 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 35 36 37 | + | M SAL 1 3 35 36 37 |
| − | M SBL 1 1 38 | + | M SBL 1 1 38 |
| − | M SMT 1 COOH | + | M SMT 1 COOH |
| − | M SVB 1 38 3.7759 -2.0328 | + | M SVB 1 38 3.7759 -2.0328 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AADGL0012 | + | ID FL7AADGL0012 |
| − | KNApSAcK_ID C00006860 | + | KNApSAcK_ID C00006860 |
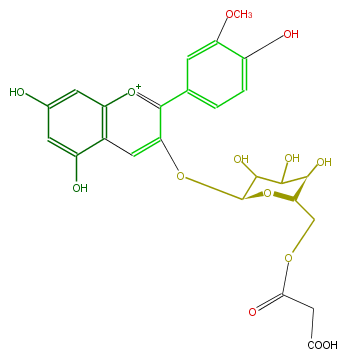
| − | NAME Peonidin 3-(6''-malonylglucoside) | + | NAME Peonidin 3-(6''-malonylglucoside) |
| − | CAS_RN 122856-11-5 | + | CAS_RN 122856-11-5 |
| − | FORMULA C25H25O14 | + | FORMULA C25H25O14 |
| − | EXACTMASS 549.124430508 | + | EXACTMASS 549.124430508 |
| − | AVERAGEMASS 549.4576 | + | AVERAGEMASS 549.4576 |
| − | SMILES O=C(CC(O)=O)OC[C@H]([C@H](O)4)O[C@H](C(C(O)4)O)Oc(c2)c(c(c3)cc(c(c3)O)OC)[o+1]c(c21)cc(cc1O)O | + | SMILES O=C(CC(O)=O)OC[C@H]([C@H](O)4)O[C@H](C(C(O)4)O)Oc(c2)c(c(c3)cc(c(c3)O)OC)[o+1]c(c21)cc(cc1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
39 42 0 0 0 0 0 0 0 0999 V2000
-3.8080 0.9682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8080 0.3259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2517 0.0047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6954 0.3259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6954 0.9682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2517 1.2894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1391 0.0047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5828 0.3259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5828 0.9682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1391 1.2894 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-1.0267 1.2893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4597 0.9620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1072 1.2893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1072 1.9440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4597 2.2713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0267 1.9440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3641 1.2893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9732 2.4440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2517 -0.6374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1667 -0.3948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3361 -0.4186 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.0679 -0.8832 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.5837 -0.7358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1027 -0.8832 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.3709 -0.4186 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.8551 -0.5660 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.0284 -0.0540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9968 -0.0372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6326 -0.1067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5051 -1.2855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0024 -2.0391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8600 -2.7844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2705 -3.1247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4803 -3.0731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4437 -3.7731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0319 -4.1127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7382 -4.1804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1763 2.8475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2650 3.7448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
22 20 1 0 0 0 0
24 30 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 2 0 0 0 0
35 37 1 0 0 0 0
15 38 1 0 0 0 0
38 39 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 38 39
M SBL 2 1 41
M SMT 2 OCH3
M SVB 2 41 -0.1763 2.8475
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 35 36 37
M SBL 1 1 38
M SMT 1 COOH
M SVB 1 38 3.7759 -2.0328
S SKP 8
ID FL7AADGL0012
KNApSAcK_ID C00006860
NAME Peonidin 3-(6''-malonylglucoside)
CAS_RN 122856-11-5
FORMULA C25H25O14
EXACTMASS 549.124430508
AVERAGEMASS 549.4576
SMILES O=C(CC(O)=O)OC[C@H]([C@H](O)4)O[C@H](C(C(O)4)O)Oc(c2)c(c(c3)cc(c(c3)O)OC)[o+1]c(c21)cc(cc1O)O
M END