Mol:FL7AACGL0109
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 76 83 0 0 0 0 0 0 0 0999 V2000 | + | 76 83 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4496 2.8293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4496 2.8293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4496 2.0043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4496 2.0043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7352 1.5918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7352 1.5918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0207 2.0043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0207 2.0043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0207 2.8293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0207 2.8293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7352 3.2418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7352 3.2418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6938 1.5918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6938 1.5918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4083 2.0043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4083 2.0043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4083 2.8293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4083 2.8293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6938 3.2418 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.6938 3.2418 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1848 3.2776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1848 3.2776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8993 2.8651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8993 2.8651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6137 3.2776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6137 3.2776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6137 4.1026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6137 4.1026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8993 4.5151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8993 4.5151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1848 4.1026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1848 4.1026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2539 4.4722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2539 4.4722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0524 1.6324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0524 1.6324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0296 3.1641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0296 3.1641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7352 0.8291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7352 0.8291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2738 2.8965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2738 2.8965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3092 -1.2520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3092 -1.2520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1269 -1.3635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1269 -1.3635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4505 -0.6043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4505 -0.6043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1157 -0.1159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1157 -0.1159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2980 -0.0043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2980 -0.0043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9744 -0.7635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9744 -0.7635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4151 -0.5289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4151 -0.5289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3953 -1.9299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3953 -1.9299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6473 -1.5595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6473 -1.5595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0659 -1.0987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0659 -1.0987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3596 0.7390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3596 0.7390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8717 0.0734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8717 0.0734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1008 0.3681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1008 0.3681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2874 0.2289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2874 0.2289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7752 0.8945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7752 0.8945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5461 0.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5461 0.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7659 1.1652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7659 1.1652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7874 0.2061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7874 0.2061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3459 0.2821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3459 0.2821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2017 -0.2867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2017 -0.2867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2418 1.5640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2418 1.5640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5860 -3.7875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5860 -3.7875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3491 -3.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3491 -3.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2476 -2.6543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2476 -2.6543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5777 -1.8979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5777 -1.8979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8146 -2.2121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8146 -2.2121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9159 -3.0311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9159 -3.0311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3145 -3.1090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3145 -3.1090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0008 -4.3306 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0008 -4.3306 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0328 -3.8837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0328 -3.8837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8592 -2.9091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8592 -2.9091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8627 -0.6754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8627 -0.6754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5958 -3.8006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5958 -3.8006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1838 -3.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1838 -3.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5958 -2.3736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5958 -2.3736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3599 -3.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3599 -3.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0520 -3.8006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0520 -3.8006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8759 -3.8006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8759 -3.8006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2884 -3.0862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2884 -3.0862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1134 -3.0862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1134 -3.0862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5259 -3.8006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5259 -3.8006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1134 -4.5151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1134 -4.5151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2884 -4.5151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2884 -4.5151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3498 -3.8006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3498 -3.8006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2598 -1.2556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2598 -1.2556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6981 -2.0202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6981 -2.0202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5641 -1.2556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5641 -1.2556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9760 -1.9691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9760 -1.9691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7999 -1.9691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7999 -1.9691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2124 -1.2547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2124 -1.2547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0374 -1.2547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0374 -1.2547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4499 -1.9691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4499 -1.9691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0374 -2.6836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0374 -2.6836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2124 -2.6836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2124 -2.6836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2738 -1.9691 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2738 -1.9691 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 8 18 1 0 0 0 0 | + | 8 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 13 21 1 0 0 0 0 | + | 13 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 33 40 1 0 0 0 0 | + | 33 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 42 38 1 0 0 0 0 | + | 42 38 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 43 1 0 0 0 0 | + | 48 43 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 43 50 1 0 0 0 0 | + | 43 50 1 0 0 0 0 |
| − | 44 51 1 0 0 0 0 | + | 44 51 1 0 0 0 0 |
| − | 45 52 1 0 0 0 0 | + | 45 52 1 0 0 0 0 |
| − | 28 53 1 0 0 0 0 | + | 28 53 1 0 0 0 0 |
| − | 31 46 1 0 0 0 0 | + | 31 46 1 0 0 0 0 |
| − | 20 35 1 0 0 0 0 | + | 20 35 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 55 56 2 0 0 0 0 | + | 55 56 2 0 0 0 0 |
| − | 55 57 1 0 0 0 0 | + | 55 57 1 0 0 0 0 |
| − | 57 58 2 0 0 0 0 | + | 57 58 2 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 60 2 0 0 0 0 | + | 59 60 2 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 61 62 2 0 0 0 0 | + | 61 62 2 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | 63 64 2 0 0 0 0 | + | 63 64 2 0 0 0 0 |
| − | 64 59 1 0 0 0 0 | + | 64 59 1 0 0 0 0 |
| − | 62 65 1 0 0 0 0 | + | 62 65 1 0 0 0 0 |
| − | 54 49 1 0 0 0 0 | + | 54 49 1 0 0 0 0 |
| − | 66 67 2 0 0 0 0 | + | 66 67 2 0 0 0 0 |
| − | 66 68 1 0 0 0 0 | + | 66 68 1 0 0 0 0 |
| − | 68 69 2 0 0 0 0 | + | 68 69 2 0 0 0 0 |
| − | 69 70 1 0 0 0 0 | + | 69 70 1 0 0 0 0 |
| − | 70 71 2 0 0 0 0 | + | 70 71 2 0 0 0 0 |
| − | 71 72 1 0 0 0 0 | + | 71 72 1 0 0 0 0 |
| − | 72 73 2 0 0 0 0 | + | 72 73 2 0 0 0 0 |
| − | 73 74 1 0 0 0 0 | + | 73 74 1 0 0 0 0 |
| − | 74 75 2 0 0 0 0 | + | 74 75 2 0 0 0 0 |
| − | 75 70 1 0 0 0 0 | + | 75 70 1 0 0 0 0 |
| − | 73 76 1 0 0 0 0 | + | 73 76 1 0 0 0 0 |
| − | 53 66 1 0 0 0 0 | + | 53 66 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGL0109 | + | ID FL7AACGL0109 |
| − | KNApSAcK_ID C00014766 | + | KNApSAcK_ID C00014766 |
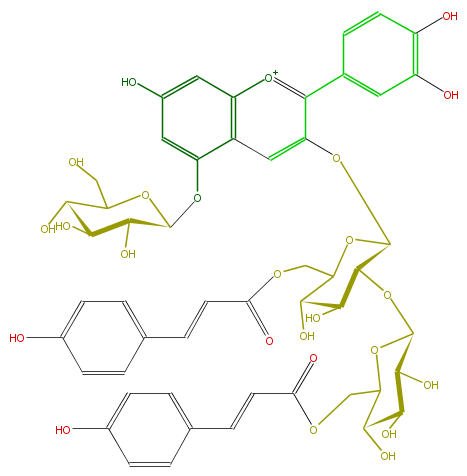
| − | NAME Cyanidin 3-[2-(6-p-coumarylglucosyl)-6-p-coumarylglucoside)]-5-glucoside | + | NAME Cyanidin 3-[2-(6-p-coumarylglucosyl)-6-p-coumarylglucoside)]-5-glucoside |
| − | CAS_RN 203396-74-1 | + | CAS_RN 203396-74-1 |
| − | FORMULA C51H53O25 | + | FORMULA C51H53O25 |
| − | EXACTMASS 1065.287592246 | + | EXACTMASS 1065.287592246 |
| − | AVERAGEMASS 1065.95152 | + | AVERAGEMASS 1065.95152 |
| − | SMILES c(c([o+1]8)c(cc(c78)c(cc(O)c7)OC(C6O)OC(C(C6O)O)CO)OC(C(OC(C(O)4)OC(COC(=O)C=Cc(c5)ccc(c5)O)C(C4O)O)3)OC(C(O)C3O)COC(C=Cc(c2)ccc(c2)O)=O)(c1)cc(c(O)c1)O | + | SMILES c(c([o+1]8)c(cc(c78)c(cc(O)c7)OC(C6O)OC(C(C6O)O)CO)OC(C(OC(C(O)4)OC(COC(=O)C=Cc(c5)ccc(c5)O)C(C4O)O)3)OC(C(O)C3O)COC(C=Cc(c2)ccc(c2)O)=O)(c1)cc(c(O)c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
76 83 0 0 0 0 0 0 0 0999 V2000
-1.4496 2.8293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4496 2.0043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7352 1.5918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0207 2.0043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0207 2.8293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7352 3.2418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6938 1.5918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4083 2.0043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4083 2.8293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6938 3.2418 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
2.1848 3.2776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8993 2.8651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6137 3.2776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6137 4.1026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8993 4.5151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1848 4.1026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2539 4.4722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0524 1.6324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0296 3.1641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7352 0.8291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2738 2.8965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3092 -1.2520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1269 -1.3635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4505 -0.6043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1157 -0.1159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2980 -0.0043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9744 -0.7635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4151 -0.5289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3953 -1.9299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6473 -1.5595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0659 -1.0987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3596 0.7390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8717 0.0734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1008 0.3681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2874 0.2289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7752 0.8945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5461 0.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7659 1.1652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7874 0.2061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3459 0.2821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2017 -0.2867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2418 1.5640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5860 -3.7875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3491 -3.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2476 -2.6543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5777 -1.8979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8146 -2.2121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9159 -3.0311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3145 -3.1090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0008 -4.3306 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0328 -3.8837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8592 -2.9091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8627 -0.6754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5958 -3.8006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1838 -3.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5958 -2.3736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3599 -3.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0520 -3.8006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8759 -3.8006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2884 -3.0862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1134 -3.0862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5259 -3.8006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1134 -4.5151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2884 -4.5151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3498 -3.8006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2598 -1.2556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6981 -2.0202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5641 -1.2556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9760 -1.9691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7999 -1.9691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2124 -1.2547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0374 -1.2547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4499 -1.9691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0374 -2.6836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2124 -2.6836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2738 -1.9691 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
8 18 1 0 0 0 0
1 19 1 0 0 0 0
3 20 1 0 0 0 0
13 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
25 18 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
37 38 1 0 0 0 0
32 39 1 0 0 0 0
33 40 1 0 0 0 0
34 41 1 0 0 0 0
42 38 1 0 0 0 0
43 44 1 1 0 0 0
44 45 1 1 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 48 1 0 0 0 0
48 43 1 0 0 0 0
48 49 1 0 0 0 0
43 50 1 0 0 0 0
44 51 1 0 0 0 0
45 52 1 0 0 0 0
28 53 1 0 0 0 0
31 46 1 0 0 0 0
20 35 1 0 0 0 0
54 55 1 0 0 0 0
55 56 2 0 0 0 0
55 57 1 0 0 0 0
57 58 2 0 0 0 0
58 59 1 0 0 0 0
59 60 2 0 0 0 0
60 61 1 0 0 0 0
61 62 2 0 0 0 0
62 63 1 0 0 0 0
63 64 2 0 0 0 0
64 59 1 0 0 0 0
62 65 1 0 0 0 0
54 49 1 0 0 0 0
66 67 2 0 0 0 0
66 68 1 0 0 0 0
68 69 2 0 0 0 0
69 70 1 0 0 0 0
70 71 2 0 0 0 0
71 72 1 0 0 0 0
72 73 2 0 0 0 0
73 74 1 0 0 0 0
74 75 2 0 0 0 0
75 70 1 0 0 0 0
73 76 1 0 0 0 0
53 66 1 0 0 0 0
S SKP 8
ID FL7AACGL0109
KNApSAcK_ID C00014766
NAME Cyanidin 3-[2-(6-p-coumarylglucosyl)-6-p-coumarylglucoside)]-5-glucoside
CAS_RN 203396-74-1
FORMULA C51H53O25
EXACTMASS 1065.287592246
AVERAGEMASS 1065.95152
SMILES c(c([o+1]8)c(cc(c78)c(cc(O)c7)OC(C6O)OC(C(C6O)O)CO)OC(C(OC(C(O)4)OC(COC(=O)C=Cc(c5)ccc(c5)O)C(C4O)O)3)OC(C(O)C3O)COC(C=Cc(c2)ccc(c2)O)=O)(c1)cc(c(O)c1)O
M END